XiaoMi-AI文件搜索系统
World File Search System揭秘 Akkermansia muciniphila Akk11
ATCC BAA-835 T [9, 34] 中这些基因的存在进一步证明这些风险基因可能是 A. muciniphila 所固有的。使用 ISfinder 和 blastn 分析了移动遗传元件 (MGE),包括质粒、插入序列 (IS) 和整合子。检测到 Akk11 中存在 IS,这与
生物信息学实验室 - 基因组装
即使使用SNP搜索工具,我们也不能依靠提出的变体。有一些结核分枝杆菌基因组的区域很难有效地映射,例如高度重复的PE / PPE / PGRS基因和IS(插入序列位点)。此外,当我们的样品中出现插入或缺失(Indel)相对于参考时,它可能会导致独特的核苷酸的假变体显得靠近Indel。最后,如果很少有序列映射到参考基因组的区域,这要么是由于缺失序列或由于基因组区域中的GC含量高,因此我们无法确定这些变体的鉴定质量。“ TB变体过滤器”工具可以根据各种标准(包括上面列出的标准)帮助过滤变体。
报告名称:农业生物技术年度
转基因修饰(GM)植物”(以下称为“指南”)以供公众评论。该指南将Mara的技术指南与2022年1月更新的“农业转基因生物的安全评估的行政措施”保持一致。该指南适用于国内生产(种植)和作为加工材料的进口的生物安全证书申请。指南草案罢工“品种”一词,并用“植物”或“事件”代替。此修订与更新的“农业转基因生物安全评估的行政措施”中包含的语言保持一致,该语言从“作物品种和事件”基础上的基础上修改了生物安全评估的性质,从而仅是“事件”的基础。此外,该指南草案还增加了“序列确定”,以分析植物基因组中插入序列的整合的方法。此语言似乎打开了使用下一代测序(NGS)的可能性。
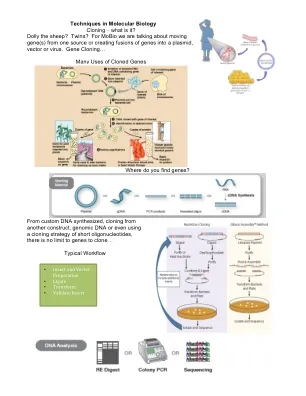
分子生物学克隆的技术
您的转弯读数在框架中您有一系列蛋白质的一部分,您希望使用虚构的GST质粒克隆到称为谷胱甘肽S转移酶(GST)的融合蛋白中。•您使用哪些PGSTPLASTID?a,b还是C?•您将与Bamhi和Ecori一起切入。•确保您具有正确的读取框架,可以在插入序列的末尾停止。请记住,GST的插入为5'。That is where the “ #1 bp ” is located • Google search for “ Restriction Sequence Translator ” to find a program to map for where the RE cuts sites are • Sequence GGATCCTGTAGATCTGCTGGCAGTTAAGAAGAAGCAGGAAACCAAACGTAGCATCAATGAGGA gattcatacccagttcctggatcatctgctgctgactggcatgaggacatctgcggcggtcactatgg tcaccatcaccacgaate•vdllavkkkkkkq etkrsineei htqfldhllt htqfldhllt giedicghyg hhh•找到地图并保留您的选择!•GST和C端的插入物在哪里?- 使用DNA到氨基酸翻译器在DNA序列中找到编码的氨基酸序列。
卢卡斯·基什内尔 - OPUS Würzburg
ACN 乙腈 AMP 抗菌肽 AMR 抗菌抗性 aq. 水溶液 ATC 无水四环素 CA 纤维素乙酸酯 CE 碰撞能量 cf. Confer (lt.) CLSI 临床和实验室标准研究所 CS 校准标准 CTA 纤维素三乙酸酯 DAP 达托霉素 DAP-R 达托霉素耐药性 DHA 脱氢丙氨酸 DNA 脱氧核糖核酸 drc 达托霉素耐药性簇 eg Exempli gratia EIC 提取离子色谱图 EMA 欧洲药品管理局 ESI 电喷雾电离 EUCAST 欧洲抗菌药物敏感性测试委员会 FA 甲酸 FDA 美国食品药品管理局 FV 碎裂电压 GUCS 一般未知物比较筛选 HGT 水平基因转移 (HP)LC(高效)液相色谱法 HRMS 高分辨率质谱法 ICH 人用药品技术要求国际协调会 IDA 信息依赖性采集 ie Id est (lt.) IS 插入序列 ISMF 内标标准化基质因子 ISTD 内标 Kyn 犬尿氨酸 LB(Eppendorf)蛋白质 LoBind ®
使用CRISPR-CAS9,单链DNA寡核苷酸和原生质体再生
基因组编辑需要将DNA序列插入特定位置。涉及定期间隔短的短文重复序列(CRISPRS)和CRISPR相关(CAS)蛋白质的方案依赖于同源性维修,需要费力的矢量构造,并且效率低。DNA寡核苷酸可以通过非同源末端连接用作靶向插入的供体。我们的简单协议通过使用聚乙烯乙二醇将非修饰的单链DNA DNA寡核苷酸和CRISPR-CAS9核糖核蛋白输送到原生质体中,从而消除了对昂贵的设备和矢量结构的需求。,我们在烟草本尼亚娜纳(Nicotiana Benthamiana)中实现了高达50.0%的靶向插入频率,而无需抗生素选择即可快速循环胸腺橄榄石。使用每个同源臂中包含27 nt的60 nt供体,22个再生植物中有6个显示出靶向插入,其中1个包含6 bp Eco RI位点的精确插入。全基因组测序表明,DNA仅插入靶向位置,遗传分析表明,插入到下一代的插入序列。
基因组纪事
十多年前 [1] ( ) 发现了 CRISPR/Cas9 系统,这使我们干预基因组的可能性增加了十倍,无论是在研究中,还是在最终使基因治疗成为现实 [2] ( )。 CRISPR 系统及其衍生物现在可以相对容易地在复杂基因组的特定点切割 DNA;切口的修复通常以某种随机的方式进行,在修复点添加一些核苷酸,导致目标基因失活。通过这种方式,可以“关闭”某个过度表达会导致疾病的基因,以达到基因治疗的目的,甚至可以在体内实现[3]( )。最近,这些系统得到了改进,可以实现真正的基因组编辑,即通过程序将一个核苷酸替换为另一个核苷酸,从而可以纠正有害突变 [4]。但是将几百或几千个核苷酸的序列精确插入到基因组的某个点仍然遥不可及,至少如果我们想有效地做到这一点的话(而不是在极小部分的被处理的细胞中)。这也解释了最近发表的一种新方法所引起人们的兴趣,该方法利用了插入序列家族编码的重组酶的特性 [5, 6],而且,它可以完全通过双特异性向导 RNA 的序列进行编程。
转座子相关的 TnpB 是一种可编程的 RNA-...
转座在重塑所有生物体的基因组中起着关键作用 1 。IS200/IS605 和 IS607 家族 2 的插入序列是最简单的移动遗传元件之一,仅包含其转座及其调控所需的基因。这些元件编码 tnpA 转座酶,这对于动员至关重要,并且通常携带辅助 tnpB 基因,而该基因对于转座而言并非必需。尽管 TnpA 在 IS200/IS605 转座子动员中的作用已得到充分证实,但 TnpB 的功能仍然很大程度上未知。有人提出 TnpB 在转座调控中发挥作用,尽管尚未确定相关机制 3–5 。生物信息学分析表明 TnpB 可能是 CRISPR–Cas9/Cas12 核酸酶的前身 6–8 。然而,尚未发现 TnpB 具有任何生化活性。我们在此表明,耐辐射奇球菌 ISDra2 的 TnpB 是一种 RNA 引导的核酸酶,受来自转座子右端元件的 RNA 引导,切割 5′-TTGAT 转座子相关基序旁的 DNA。我们还表明,TnpB 可以重新编程以切割人类细胞中的 DNA 靶位。总之,这项研究通过强调 TnpB 在转座中的作用扩展了我们对转座机制的理解,通过实验证实了 TnpB 是 CRISPR-Cas 核酸酶的功能性前体,并将 TnpB 确立为基因组编辑新系统的原型。
非小细胞肺癌中的 EGFR 外显子 20 插入突变
其结构主要由胞外区、跨膜区和胞内酪氨酸激酶结构域三部分组成。EGFR基因全长192kbp,由28个外显子组成,位于7号染色体短臂7p21-14区域。大多数突变发生在18~21外显子,不同类型的突变对EGFR TKI临床疗效的影响不同。外显子19的缺失和外显子21的L858R替换是EGFR最常见的两种突变,且对TKI敏感。EGFR Ex20Ins突变是第三种最典型的EGFR突变类型,已知其与吉非替尼、厄洛替尼等常见TKI耐药有关。目前,EGFR外显子20插入突变类型共122种,位于C螺旋后的Met766-Cys775,少数位于C螺旋后的G1u762-Tyr764。其中20.5%的插入发生在Val769位氨基酸之后,28.7%的插入发生在Asp770位氨基酸之后,17.2%的插入发生在Pro772位氨基酸之后,14%的插入发生在His773位氨基酸之后(5)。最常见的突变类型为Asp770_Asn771ins,其次为Va1769_Asp770ins、Asp770_Asn771ins、A1a767_Va1769、Va1769_Asp770ins和Ser768_Asp770,其插入序列基本相似。 EGFR Ex20Ins 是一个高度异质性的激活突变家族,其分子结构、生物学特性和对 EGFR TKI 的反应存在复杂的差异。对 EGFR 外显子 20 突变进行了分析
移动DNA充满了热点,用于基因调节的从头出现
产生新基因表达的抽象DNA突变是达尔文进化的重要原材料。新基因调控的一个潜在来源是移动DNA,有时可以通过向外指导的启动子来驱动其在基因组中插入位点附近的基因的表达。但是,我们不知道这种能力的频率有多,也不知道移动DNA可能会发展起来。在这里,我们为插入序列家族IS3解决了这些问题,这是一种简单形式的原核生物移动DNA的家族。首先,我们估计至少有30%的IS3序列向外指导的启动子。第二,我们将高通量诱变与大量平行的记者测定法相结合,以表明在我们研究的所有IS3序列中,单点突变足以创建外向启动子。我们发现,在18'607突变体IS3序列中,有5.6%的启动子活性从头出现。启动子优先出现在每个IS3序列中的出现热点。这些热点与已经存在或通过突变新创建的启动子图案重叠。启动子活动的一条通用途径是获得一个现有-35盒子下游的-10盒子,我们称之为“ shiko出现”。总体而言,我们表明移动DNA具有驱动新基因表达的高潜力。这使移动DNA非常适合其宿主有机体驯化。它还提出了有关这种潜力如何发展的有趣问题。简介