XiaoMi-AI文件搜索系统
World File Search SystemExpressPlex™ 2.0 文库制备试剂盒 – 384 孔
正在申请专利的 ExpressPlex 2.0 文库制备试剂盒采用方便的 384 孔 PCR 板配置,可用于高通量多重文库制备。此升级版 ExpressPlex 使用 seqWell 的高性能 TnX ™ 转座酶,该转座酶专为 NGS 文库制备而设计。扩增子 (>350 bp) 和质粒 DNA 是适合该试剂盒的标准输入。附录 E 重点介绍了可以针对小型微生物全基因组测序进行的修改。ExpressPlex 文库与 Illumina MiSeq ™ 、NextSeq ™ 、iSeq ™ 和 NovaSeq ™ 测序平台兼容。每个 ExpressPlex 2.0 - 384 孔试剂盒都包含足够的试剂,可从 384 或 1,536 个单独的 DNA 样本制备与 Illumina 兼容的文库。每个库的标准制备量为 384 个样本,每个试剂盒最多 1,536 个样本。有四种不同的试剂盒可用于从 1,536 个样本中制备文库,在一次测序运行中可加载总共 6,144 种条形码组合。这种多重文库制备程序针对每 8 µl 反应 0.5 - 20 ng 质粒或扩增子 DNA 的输入进行了优化,通常可生成 400 – 1,200 bp 的文库片段长度。文库片段长度取决于 DNA 的质量和所用的磁珠清理率。使用 ExpressPlex 文库制备试剂盒的主要优势和好处是简化的一步式多重文库制备工作流程,可在 40 倍的 DNA 输入浓度范围内自动标准化每个样本的读取输出,同时最大限度地减少人工和耗材成本。使用 ExpressPlex 2.0 – 384 孔试剂盒,可在 120 分钟内制备 384 重文库以进行文库 QC 和测序,手动操作时间不到 30 分钟。
有机偏光发光二极管具有较高的亮度和颜色纯度朝向激光显示
抽象的宪法复杂染色体重排(CCR)是通过未知机制在种系中产生的罕见细胞遗传畸变。在这里,我们使用全面的基因组和表观基因组分析分析了微观三向或更复杂的易位的断点连接。所有这些易位连接均显示出伪造的基因组复杂性。这些断点聚集在小基因组域中,该结构域显示了微学或微插入。值得注意的是,所有从头案件都是父亲的起源。突破点分布特别对应于ATAC-SEQ(带测序的转座酶可访问染色质的测定)读取成熟精子的数据峰,而不是其他染色质标记或组织。我们提出,在脂肪生成后的精子发生过程中,CCR中的DNA断裂可能会在可接收的密集染色质区域中发展。
核CRISPR相关的转座子的宏基因组发现
摘要CRISPR相关的转座子(铸造)CAS基因用于RNA引导的转座。在基因组数据库中极为罕见。最近的调查报道了类似TN7样的转座子,该座子选择了I型I-F,I-B和V-K CRISPR效应子。在这里,我们通过对元基因组数据库的生物信息学搜索扩展了报告的铸造系统的多样性。我们发现了所有已知铸件的新架构,包括级联效应器的新布置,新的自动定位方式和最小的V-K系统。我们还描述了采用I型I-C和IV型CRISPR-CAS系统的新型演员群。我们对非TN7铸造的搜索确定了对水平基因转移的合作候选者。这些新系统阐明了CRISPR系统如何与转座酶一起进化并扩展可编程基因编辑工具包。
SLC30A8 基因座的多种遗传变异影响局部超级增强子活性并影响胰腺 β 细胞的存活和功能
缩写:3C,染色体构象捕获;4C,环状染色体构象捕获;ATAC-seq,使用测序检测转座酶可及染色质;Cas9,来自化脓性链球菌的内切酶;CHIP-seq,染色质免疫沉淀和 DNA 测序;CRISPR,成簇的规律间隔的短回文重复序列;CTCF,CCCTC 结合因子;EXT1,外骨化素糖基转移酶 1;GSIS,葡萄糖刺激的胰岛素分泌;GWAS,全基因组关联研究;MED30,RNA 聚合酶 II 转录亚基 30 的介质;pcHi-C,启动子捕获 Hi-C;R,调控区;RAD21,双链断裂修复蛋白 rad21 同源物;SLC30A8,溶质载体家族 30 成员 8;SNP,单核苷酸多态性; T2D,2 型糖尿病;TAD,拓扑关联结构域;UTP23,UTP23 小亚基加工体成分。
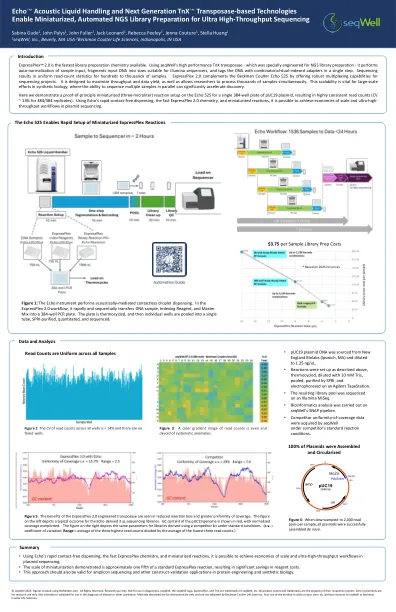
回声声学液体处理和下一代TNX ...
2.0是最快的图书馆准备化学。使用Seqwell的高性能TNX转座酶(专门针对NGS库制备设计),它对样品输入的自动归一化,片段输入DNA成适合Illumina测序仪的尺寸,并在单步中使用Sembers Input dna进行适用于Illumina序列的尺寸,并用组合型二异形指示剂将DNA标记。测序导致数百至数千个样本的统一读数统计数据。ExpressPlex 2.0通过为测序项目提供强大的多重功能来补充Beckman Coulter Echo 525。它旨在最大化吞吐量和数据产量,并允许研究人员同时处理数千个样本。这种可伸缩性对于合成生物学的大规模努力至关重要,在合成生物学方面,对并行多个样品进行测序的能力可以显着加速发现。
CRISPR 相关转座子的宏基因组学发现
摘要 CRISPR 相关转座子 (CAST) 会将 Cas 基因纳入 RNA 引导的转座。CAST 在基因组数据库中极为罕见;最近的调查报告称,Tn7 样转座子会将 IF、IB 和 VK 型 CRISPR 效应子纳入。在这里,我们通过对宏基因组数据库进行生物信息学搜索来扩展已报告的 CAST 系统的多样性。我们发现了所有已知 CAST 的新架构,包括级联效应子的新排列、新的自靶向模式和最小 VK 系统。我们还描述了已将 IC 型和 IV 型 CRISPR-Cas 系统纳入的新 CAST 家族。我们对非 Tn7 CAST 的搜索确定了将 Cas12a 纳入水平基因转移的推定候选者。这些新系统揭示了 CRISPR 系统如何与转座酶共同进化并扩展了可编程基因编辑工具包。
用于高通量筛选的标准化转座子工具
随着合成生物学研究的规模越来越大,在活细胞中设计预定义功能需要越来越精确的工具。此外,遗传构建体表型性能的表征需要细致的测量和广泛的数据采集,以便在设计-构建-测试生命周期中为数学模型提供信息并匹配预测。在这里,我们开发了一种简化高通量转座子插入测序 (TnSeq) 的遗传工具:携带 Himar1 Mariner 转座酶系统的 pBLAM1-x 质粒载体。这些质粒源自 mini-Tn5 转座子载体 pBAMD1- 2,并按照标准欧洲载体结构 (SEVA) 格式的模块化标准构建。为了展示它们的功能,我们分析了 60 个土壤细菌 Pseudomonas putida KT2440 克隆的测序结果。新的 pBLAM1-x 工具已经包含在最新的 SEVA 数据库版本中,我们在这里使用实验室自动化工作流程描述了它的性能。
联盟1联盟1
下一代测序(NGS)技术已被引入基因组研究已有15年以上。更多的研究人员正在使用NGS作为常规工具来解决他们的研究问题。随着测序质量的成熟和标准化,总体测序数据的质量更由构建的NGS库的质量确定。易于重复的NGS图书馆准备工作流程均匀性能对于研究人员至关重要,尤其是那些处理稀缺和珍贵样本的人。Micronbrane的Unison™Ultralow DNA ngs库Prep套件基于基于良好的转座酶的DNA标记,在PCR扩增步骤之前无需对低输入DNA本身的操作或最少操纵。将酶浓度仔细滴定并针对低输入DNA进行了优化。因此,该试剂盒不仅可以从低至10pg的DNA的超低DNA输入成功构建NGS库,还可以确保放大库的测序曲线是输入DNA样品的真实表示。
生物化学年度评论 利用 CRISPR 相关 Tn7 样转座子进行天然和人工引导 RNA 定向转座
CRISPR-Cas(成簇的规律间隔的短回文重复序列 - CRISPR 相关核酸酶)防御系统已多次自然地用于指导 RNA 定向转座。在所有情况下,转座子 Tn7 相关的各种元件都参与了转座。Tn7 严格控制转座;只有当专用靶位选择蛋白识别特殊靶标时,转座酶才会被激活。Tn7 和与 CRISPR-Cas 系统合作的 Tn7 样元件进化出了互补的靶向途径:一条途径识别染色体中高度保守的位点,另一条途径靶向能够进行细胞间转移的移动质粒。Tn7 和 Tn7 样元件将单一整合传递到它们识别的位点,并控制整合事件的方向,为未来用作可编程基因整合工具提供了潜力。早期研究表明,引导 RNA 介导的转座系统可以适应不同的宿主,甚至在微生物群落内,这表明将这些系统设计为强大的基因编辑工具具有巨大的潜力。
寻找并切割转移 (FiCAT) 哺乳动物基因组工程
虽然存在多种用于小等位基因基因组编辑的技术,但仍然缺乏用于在哺乳动物基因组中靶向整合大 DNA 片段的强大技术。在这里,我们开发了一种基因传递工具 (FiCAT),它结合了 CRISPR-Cas9(发现模块)的精确度和工程化 piggyBac 转座酶(切割和转移模块)的有效载荷转移效率。FiCAT 结合了 Cas9 DNA 扫描和靶向 DNA 的功能以及 piggyBac 供体 DNA 处理和转移能力。PiggyBac 功能域经过工程设计,可提高靶向整合率,同时减少脱靶事件。我们展示了在细胞(人类(Hek293T、K-562)和小鼠(C2C12))和小鼠肝脏体内有效传递和可编程插入小型和大型有效载荷。最后,我们通过生成 394,000 个变体的靶向多样性并进行 4 轮进化,开发出更高效的 FiCAT 版本。在这项工作中,我们开发了一种在哺乳动物基因组中精确、有效地靶向插入多千碱基 DNA 片段的方法。