XiaoMi-AI文件搜索系统
World File Search System吡格列酮作为2型糖尿病患者的附加疗法,用dapagliflozin和二甲甲蛋白不充分控制:双盲,Rando
1内分泌学和代谢司,内科,哈利姆大学神圣心脏医院,anyang,2内分泌学和代谢司,内科,诺伊·欧尔吉医学中心,埃尔吉大学医学院,纽约大学医学司,尤尔吉大学医学院,尤尔吉医学中心,尤尔吉大学医学院3和代谢,Daegu Daegu Fatima医院内部医学系,Daegu 5,内科和代谢部门5级,Hallym University Dongtan Sacred Hospital,Hwaseong,Hwaseong 6,内分泌学和代谢部,HALLYM University Hapernosic nounidest of Intersial of Intersial Medicine of Nifentary of Nifentary of Nifentary of Nifentary of Nifentary of Nifentain of Nifentary of Nifential forsip.首尔,韩国首尔大学医学院内科学系内部分泌学和新陈代谢8分司,
针对新诊断 2 型糖尿病且伴有严重高血糖症患者的强化简化策略:多中心、开放标签、随机试验
结果 412 名参与者被随机分配。基线时,平均年龄为 46.8 岁(标准差 11.2 岁),平均体重指数为 25.8(2.9),平均 HbA 1c 为 11.0%(1.9%)。第 48 周时,利格列汀联合二甲双胍组、利格列汀组和二甲双胍组分别有 80%(78/97)、72%(63/88)和 73%(69/95)的患者达到 HbA 1c <7.0%,而对照组为 60%(56/93)(总体 P=0.02;利格列汀联合二甲双胍组与对照组 P=0.003;利格列汀组与对照组 P=0.12;二甲双胍组与对照组 P=0.09)。此外,利格列汀联合二甲双胍组、利格列汀组和二甲双胍组分别有 70% (68/97)、68% (60/88) 和 68% (65/95) 的患者达到 HbA 1c <6.5%,而对照组只有 48% (45/93)(总体 P=0.005;利格列汀联合二甲双胍组 vs 对照组 P=0.005;利格列汀组 vs 对照组 P=0.01;二甲双胍组 vs 对照组 P=0.008;经过多重比较调整后均具有显著性)。因此,与对照组相比,利格列汀联合二甲双胍组参与者在第 48 周时更有可能达到 HbA 1c <7.0%(风险比 2.78,95% 置信区间 1.37 至 5.65;P=0.005)。此外,利格列汀联合二甲双胍组空腹血糖和β细胞功能指标改善最为显著。所有治疗均耐受性良好。
根据图统计信息生成神经图
我们描述了一种从聚合图统计数据(而不是图邻接矩阵)学习深度图生成模型 (GGM) 的新设置。匹配观察到的训练图的统计数据是学习传统 GGM(例如 BTER、Chung-Lu 和 Erdos-Renyi 模型)的主要方法。隐私研究人员已提出从图统计数据中学习作为保护隐私的一种方式。我们开发了一种架构来训练深度 GGM 以匹配统计数据,同时保留局部差异隐私保证。对 8 个数据集的实证评估表明,当两者都仅从图统计数据中学习时,我们的深度 GGM 比传统的非神经 GGM 生成更逼真的图。我们还将仅在统计数据上训练的深度 GGM 与在整个邻接矩阵上训练的最先进的深度 GGM 进行了比较。结果表明,图统计数据通常足以构建具有竞争力的深度 GGM,该深度 GGM 可生成逼真的图,同时保护本地隐私。
图1:准确性与CNN模型的时期图
摘要该项目提出了三种用于为EEG Net数据集创建神经网络模型的方法 - 使用CNN,CNN+LSTM和变异自动编码器(VAE)。研究评估并比较了两种方法在分类运动图像中的性能。结果表明,CNN+LSTM方法在准确性方面优于VAE方法。但是,VAE方法具有保留脑电图信号的关键特征的优势,同时降低其尺寸。两种方法都有其各自的优势和局限性,可以根据应用程序的特定要求使用。除了上述两种方法外,我们还为该数据集实施了随机森林,以对ML和DL模型的准确性成就进行比较分析。索引术语:机器学习(ML),深度学习(DL),VAE(变异自动编码器),长期短期存储网络(LSTM),脑电图(EEG)(EEG)
图 2:教学手册
有时索赔人选择向其保险公司提出索赔,要求立即完成维修。保险公司可能会向政府提出索赔,以收回他们已支付的费用。您的免赔额不会自动支付给保险公司;您必须向他们提供书面授权,授权他们代表他们收取这笔款项或为此提出单独的索赔。*上述内容是索赔人向美国空军 (USAF) 提出索赔的指南。本文件中的任何内容均不应被视为美国空军的法律建议,也不会产生任何诉讼原因,也不会对本文件中说明的任何内容对美国空军施加任何责任。这些说明或任何美国空军人员的声明均不应被解释为索赔(如果提交)将获得批准。文件的类型和数量因索赔而异,但此引用符合《联邦法规法典》(CFR)第 28 章第 14.4 部分的规定。
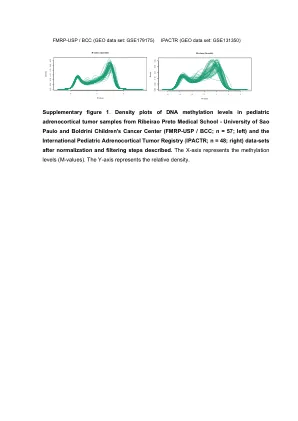
补充图1
补充图 1 | ERD7 转基因拟南芥突变株系的生成和表征。(A)根据 TAIR 提供的信息,描绘了拟南芥 ERD7 ORF (AT2G17840.1) 的插图,左侧为 5' 端。标示了 ERD7 基因外显子(框)和内含子(线)以及在相应的单拷贝、T 3 纯合 erd7-1 和 erd7-2 突变株系中针对 CRISPR/Cas9 基因组编辑的 T-DNA 插入和 sgRNA 区域的相对位置。还显示了 (C) 中用于基因分型和 RT-PCR 分析的引物对的相对位置。 (B) 野生型和 erd7-2 突变株系中 ERD7 基因和蛋白质的核苷酸和推导多肽序列的比较,表明 ERD7 基因(和相应的转录本)中预期有 1711 个核苷酸缺失,导致 erd7-2 突变株系中编码蛋白质有 398 个氨基酸缺失。ERD7 野生型和 erd7-2 突变体 DNA 序列中 CRISPR/Cas9 原间隔区相邻基序带下划线。使用 ClustalO 算法 (ebi.ac.uk/Tools/msa/clustalo) (Madeira et al., 2019) 对野生型和突变型 ERD7 蛋白质的推导氨基酸序列进行比对。(C) erd7-1 和 erd7-2 突变株系的 PCR 和 RT-PCR 分析。图中显示的是从 15 天大的 WT、erd7-1 和 erd7-2 植物的莲座叶中提取的 gDNA 的 PCR 分析(上图)和 mRNA 的 RT-PCR 分析(下图),并使用所示引物对进行评估;引物对的位置参见 (A)。有关用于生成和表征两个 erd7 突变系的所有引物序列,另请参阅补充表 1。通过 DNA 凝胶电泳和溴化乙锭染色分析 PCR 产物和 RT-PCR 产物。注意,在两个突变系中均不存在分别对应于 ERD7 基因和 ERD7 表达(即转录本)的 PCR 和 RT-PCR 产物,而 erd7-1 突变体中存在 T-DNA,这与预期一致。拟南芥 TUB4 用作内源对照。
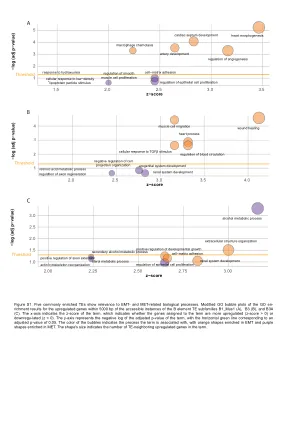
图S1
图S1。 五个通常富集的TE与EMT和MET相关的生物学过程相关。 在B1元素te subfamilies b1_mus1(a),b3(b)和b3a(c)的可访问实例的5000 bp内的GO富集结果的修改后的GO气泡图。 x轴表示该术语的z评分,该术语表明分配给该术语的基因是更上调的(z分数> 0)还是下降的(z <0)。 y轴表示该术语调整后的P值的负log,水平绿线对应于调整后的P值为0.05。 气泡的颜色表示该术语与之相关的过程,橙色形状富含EMT和紫色形状富含MET。 形状的大小指示了该术语中上调的基因的数量。图S1。五个通常富集的TE与EMT和MET相关的生物学过程相关。在B1元素te subfamilies b1_mus1(a),b3(b)和b3a(c)的可访问实例的5000 bp内的GO富集结果的修改后的GO气泡图。x轴表示该术语的z评分,该术语表明分配给该术语的基因是更上调的(z分数> 0)还是下降的(z <0)。y轴表示该术语调整后的P值的负log,水平绿线对应于调整后的P值为0.05。气泡的颜色表示该术语与之相关的过程,橙色形状富含EMT和紫色形状富含MET。形状的大小指示了该术语中上调的基因的数量。