XiaoMi-AI文件搜索系统
World File Search System通过微生物对铀的富集和修复
铀是核能工业的关键原料,预计到2040年的核能产生将增加一倍,以满足不断增长的能源需求,这使得铀供应成为能源安全的问题。此外,铀是一种重金属,既具有趋化性和放射性毒性,又严重危害了人类健康和环境安全。铀资源利用的增长增长了铀向环境的释放。因此,高度需要对铀污染和从非规定方法中回收的铀。微生物具有固定铀的高潜力。本综述总结了微生物将铀从水溶液和废水中固定在微生物物种,性能,富集和修复机制以及适用环境方面的能力。
IDT高通量杂交捕获术的长基因组片段的富集证明了协议(RUO23-2352_001 09/23)
图1。高通量杂交捕获量的长基因组片段工作流程。 (a)高分子量(HMW)基因组DNA需要长片段的制备和富集。 (b)使用高通量兼容的G管将HMW DNA碎片至〜10 kb。 (c)使用特殊准备的尺寸选择珠通过尺寸选择去除多余的较小片段。 (d)尺寸选定的片段被最终修复(ER)A-Tail(AT),并将适配器连接到适配器序列,并带有样品识别条形码序列。 (e)样品池与XGEN自定义HYB面板杂交,并捕获并富集。 (f)富集的目标片段通过远程PCR扩增。 (g)放大的富集片段是第二次尺寸选择的或清理以进行最佳测序读取长度。 然后,富集和条形码的样品池接受所需的第三代测序工作流程。高通量杂交捕获量的长基因组片段工作流程。(a)高分子量(HMW)基因组DNA需要长片段的制备和富集。(b)使用高通量兼容的G管将HMW DNA碎片至〜10 kb。(c)使用特殊准备的尺寸选择珠通过尺寸选择去除多余的较小片段。(d)尺寸选定的片段被最终修复(ER)A-Tail(AT),并将适配器连接到适配器序列,并带有样品识别条形码序列。(e)样品池与XGEN自定义HYB面板杂交,并捕获并富集。(f)富集的目标片段通过远程PCR扩增。(g)放大的富集片段是第二次尺寸选择的或清理以进行最佳测序读取长度。富集和条形码的样品池接受所需的第三代测序工作流程。
基于MBS富集策略和抗体-DNA介导的CHA扩增的SERS传感平台对CyFrA21-1的高度敏感检测
喉癌(LC)是头部和颈部第二常见的恶性肿瘤。由于其阴险的性质,大多数患者在被诊断出来时已发展到中期和晚期,缺少最佳治疗期。因此,早期检测,诊断和治疗对于改善LC的预后和提高患者的生活质量至关重要。在这项研究中,通过结合磁珠(MBS)富集策略和抗体-DNA介导的催化发夹自组装(CHA)信号放大效果技术来开发表面增强的拉曼(SERS)传感平台。4-在纳米塔时,将胃苯苯甲酸(4-MBA)和发夹DNA 1(HPDNA1)(hpDNA1)修饰到金纳米果仁酰胺(GNBPS)的表面上。发夹DNA 2(HPDNA2)修饰的MB用作捕获纳米探针。在CHA和磁体诱导的MBS富集的作用下,GNBP可以组装在MB的表面上,形成高密度的“热点”,以增强SERS信号。结果表明,SERS传感平台具有高灵敏度,高特异性和高可重现性的优势,其检测极限(LOD)低至Pg/mL水平。SERS感应平台成功地检测了LC患者血清和健康对照组中CYFRA21-1的表达水平。通过酶连接的免疫吸附测定(ELISA)验证了SERS结果的准确性。因此,该SERS传感器可用于在血清中检测CYFRA21-1,为早期诊断LC提供了一种简单可靠的新方法。
Illumina DNA准备外观2.0加富集
本文档及其内容与Illumina,Inc。及其分支机构(“ Illumina”)专有,仅用于与本文中所述的产品的使用,无目的。本文件及其内容不得用于任何其他目的和/或未经Illumina事先书面同意以任何方式传达,披露或复制的任何其他目的。Illumina不根据其专利,商标,版权或普通法权利或本文件的任何第三方的类似权利传达任何许可证。
富集
nRICH 儿童健康领域的最新研究和建议 - 旨在为您带来儿科学领域一些突破性进展的摘要,这些摘要是从全球知名期刊中精心挑选出来的。专家评论将评估文章的重要性和相关性,并讨论其在印度环境中的应用。nRICH 将涵盖儿科的所有不同亚专业,从新生儿学、胃肠病学、血液学、青少年医学、过敏和免疫学到泌尿学、神经病学、疫苗学等。每期都将以简明的摘要开始,并代表原文中的主要观点和想法。然后是来自不同亚专业的印度专家的深思熟虑和博学的评论,他们将深入介绍如何阅读和分析这些文章。我相信学生、从业者和所有有兴趣了解儿童健康领域最新研究和建议的人都将从这项将于每周一在线发布的努力中受益匪浅。
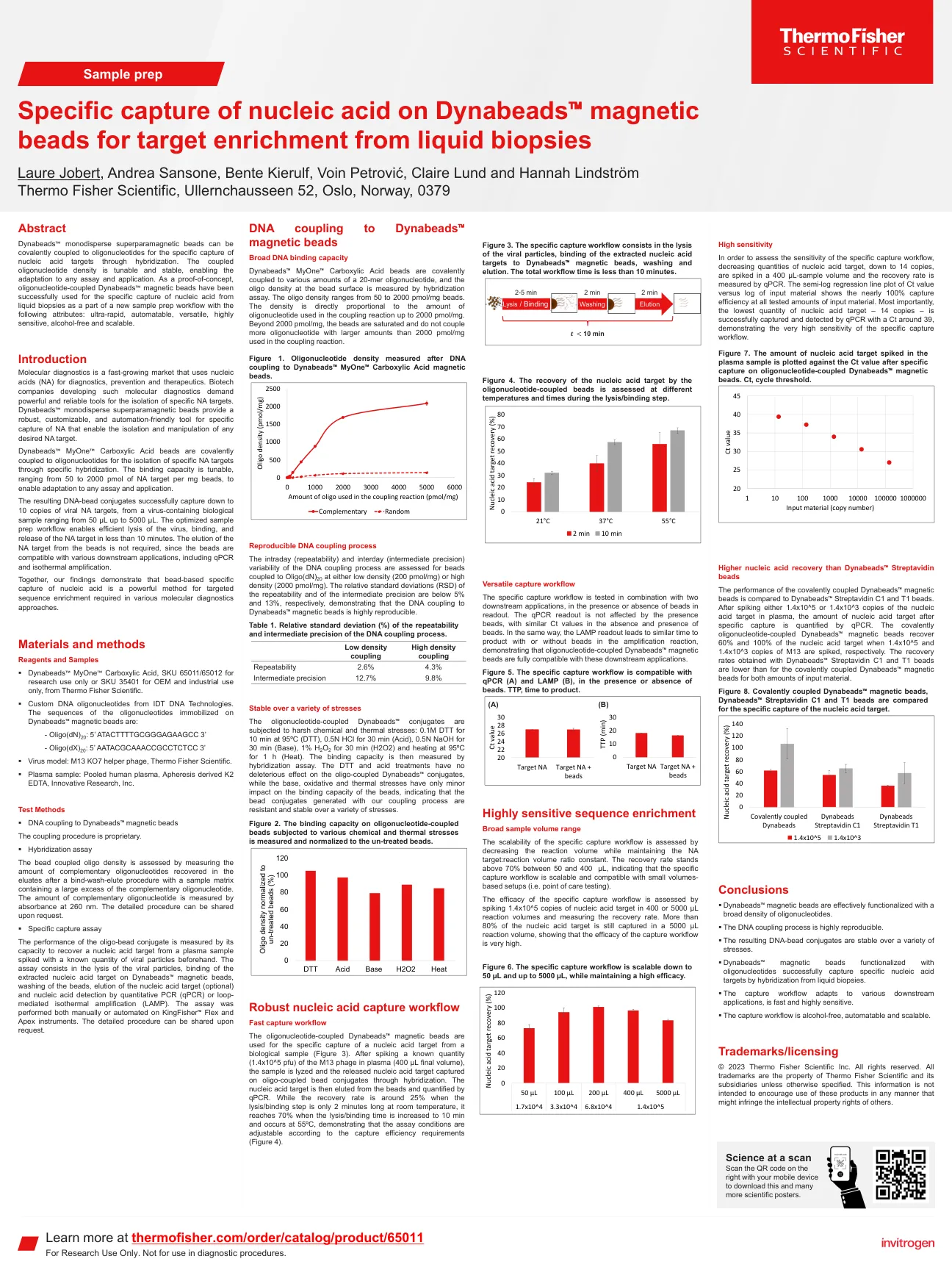
特异性捕获核酸在dynabeads™磁珠上的特异性捕获液体活检的目标富集
寡核苷酸耦合的dynabeads™磁珠用于从生物样品中特异性捕获核酸靶标(图3)。在等离子体(400 µL最终体积)中峰于M13噬菌体的已知量(1.4x10^5 pfu)后,样品被液化,并通过杂交在寡聚偶联的珠子偶联物上捕获的释放的核酸靶标。然后从珠子中洗脱核酸靶标,并通过qPCR定量。当裂解/结合步骤仅在室温下长2分钟时,回收率约为25%,但是当裂解/结合时间增加到10分钟并在55ºC处发生时,恢复速率达到70%,表明根据捕获效率要求,测定条件可以调节(图4)。
在空气 - 水接口上富集糖
理由。有机物在海面积聚。在本文中,我们提供了对持续性白泡沫中溶解糖的富集进行的首次定量评估,并将这种富集与涉及植物浮游生物的9天中型体验中的9天间中型实验中的海面微层层(SSML)进行比较。方法论。游离单糖,在轻度酸水解后确定总糖,并且将寡糖/多糖成分挖掘为挖掘,因为总和单糖之间的差异。结果。总糖水贡献了很大一部分的溶解有机碳(DOC),占海水中DOC的13%,在SSML中占27%,在泡沫中占31%。中值富集因子(EFS),计算为糖的浓度相对于SSML或SSML中的钠浓度与海水的浓度比,在SSML中为1.7至6.4,泡沫中的含量为1.7至6.4。基于EFS,木糖醇,甘露醇,葡萄糖,半乳糖,甘露糖,木糖,木糖,富藻糖,鼠李糖和核糖的中位数比SSML更富集。讨论。糖的最大EFS与较高的叶绿素水平相吻合,表明在浮游植物盛开期间,海面富集糖表面富集。SSML上海泡沫中有机物的富集较高,表明表面活性有机化合物越来越丰富在持续的气泡膜表面上。这些发现有助于解释海洋有机物如何高度富集在海洋表面上的气泡产生的海洋喷雾剂中。
靶标富集纳米孔测序和从头组装揭示了 CRISPR-Cas9 在人类细胞中诱导的复杂靶标基因组重排同时发生
CRISPR-Cas9 系统被广泛用于通过双向导 RNA 永久删除基因组区域。CRISPR-Cas9 可能会引起基因组重排,但持续的技术发展使得表征复杂的靶向效应成为可能。我们将创新的基于液滴的靶向富集方法与长读测序相结合,并将其与定制的从头序列组装相结合。这种方法使我们能够在靶向基因组位点内以千碱基规模剖析序列内容。我们在此描述了 Cas9 造成的广泛基因组破坏,包括靶区域基因组重复和倒位的等位基因共存,以及外源 DNA 的整合和聚集的染色体间 DNA 片段重排。此外,我们发现这些基因组改变导致功能异常的 DNA 片段并可改变细胞增殖。我们的发现拓宽了 Cas9 删除系统的结果范围,强调了细致的基因组验证的必要性,并引入了一种数据驱动的工作流程,能够以更高的分辨率详细剖析目标序列内容。
靶标富集纳米孔测序和从头组装揭示了由 CRISPR-Cas9 诱导的 1 个复杂的靶基因组重排同时发生
靶标富集的纳米孔测序和从头组装揭示了 CRISPR-Cas9 在人类细胞中诱导的 1 个复杂的靶基因组重排的共现 2 3 4 5 Keyi Geng 1、Lara G. Merino 1、Linda Wedemann 1、Aniek Martens 1、Małgorzata Sobota 1、6 Yerma P. Sanchez 1、Jonas Nørskov Søndergaard 1、Robert J. White 2、Claudia Kutter 1 * 7 8 1 瑞典卡罗琳斯卡医学院生命科学实验室微生物学、肿瘤和细胞生物学系 10 2 约克大学生物系,英国约克 11 * 通讯作者。电话:+46 (0) 70 4933896。电子邮件:12 claudia.kutter@ki.se 13 14 15 标题:Xdrop-LRS 揭示 CRISPR-Cas9 的靶向效应 16 17 18 关键词 19 意外的 CRISPR-Cas9 编辑、组合基因组复制-倒置-整合、20 基于液滴的靶向富集、长读测序、从头序列组装 21 22 23 摘要 24 CRISPR-Cas9 系统被广泛用于通过双 25 向导 RNA 永久删除基因组区域。CRISPR-Cas9 可能会引起基因组重排,但持续的技术发展使得表征复杂的靶向效应成为可能。我们将创新的基于液滴的靶向富集方法与长读测序相结合,并将其与定制的从头序列组装相结合。这种方法使我们能够在靶基因组位点内以千碱基规模剖析序列内容。我们在此描述了 Cas9 造成的广泛基因组破坏,包括靶区域基因组重复和倒置的等位基因共现,以及外源 DNA 的整合和聚集的染色体间 DNA 片段重排。此外,我们发现这些基因组改变导致功能异常的 DNA 片段,并可能改变细胞增殖。我们的研究结果拓宽了 Cas9 删除系统的结果范围,强调了细致的基因组验证的必要性,并引入了数据驱动的工作流程,从而能够以卓越的分辨率详细剖析靶序列内容。
通过无标记富集和重组基因工程位点 (MERGE) 实现快速、可扩展的组合基因组工程
‡ 通信地址:aashiq.kachroo@concordia.ca 关键词:基因组工程、CRISPR-Cas9、人源化酵母、蛋白酶体 缩写:CFU、菌落形成单位;DSB、双链断裂;HDR、同源定向 DNA 修复;HR、同源重组;CELECT、基于 CRISPR-Cas9 的选择以丰富基因型;MERGE、无标记富集和重组基因工程位点;SGA、合成遗传阵列