XiaoMi-AI文件搜索系统
World File Search System利用双主编辑和位点特异性重组酶进行可编程大片段 DNA 删除、替换、整合和反转
。CC-BY-NC 4.0 国际许可下可用(未经同行评审认证)是作者/资助者,他已授予 bioRxiv 永久展示预印本的许可。它是此预印本的版权持有者此版本于 2021 年 11 月 2 日发布。;https://doi.org/10.1101/2021.11.01.466790 doi:bioRxiv 预印本
片段的光电探测器效率提高了局部激光诱导的结晶
摘要:金属 - 半导体 - 金属等离激元纳米结构可以通过增强局部静电式和光学效果来实现芯片效果的操纵和超快光电检测。后者是通过使用纳米结构的薄膜(GE)等离子体 - 波导指导光电遗传学来实现的。虽然它们的大小和位置可以在纳米化过程中准确控制,但由于沉积的无定形性质,检测器的官能显着降低。我们证明,通过空间控制激光诱导的GE结晶,可以显着提高波导积分GE等离子光电探测器的效率(超过2个数量级)。我们研究了经过800 nm激光处理的自由空间和波导综合的GE光电探测器,通过拉曼光谱监测结晶程度,并通过检测电信辐射来证明效率提高。可以在各种纳米仪设备中使用所证明的局部后处理技术,以实现有效和超快的芯片辐射监测和检测,从而显着改善了检测器特性,而不会危及其他组件的性能。关键字:表面等离子体偏振子,等离激元波导,片上光反检测,激光诱导的结晶
ICOM 的修改 - 业余无线电部分和片段
Error 500 (Server Error)!!1500.That’s an error.There was an error. Please try again later.That’s all we know.
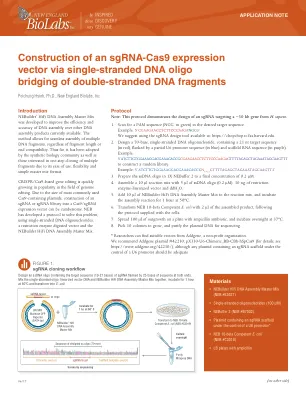
通过双链DNA片段的单链DNA寡桥构建SGRNA-CAS9表达载体
总结本申请说明证明了将SGRNA序列插入靶向DNA组件的9.5 kb载体中的便利性。与传统的克隆方法不同,必须合成和重新弥补两个寡核体,该新协议提供了一种设计寡核的简单方法,并与所需的向量组装。Nebuilder HIFI DNA组装主混合物对传统方法的实质性改进,特别是节省时间,易用性和成本。
片段 - 印第安纳大学印第安纳波利斯分校 ScholarWorks
癌症将直接影响超过三分之一人口的生活。DNA损伤反应 (DDR) 是一个复杂的系统,涉及损伤识别、细胞周期调控、DNA 修复以及最终的细胞命运决定,在癌症病因和治疗中发挥着核心作用。涉及 DDR 靶向的两种主要治疗方法包括:采用抗癌基因毒性剂的组合疗法;以及合成致死,利用散发性 DDR 缺陷作为癌症特异性治疗的机制。尽管许多 DDR 蛋白已被证明“无法用药”,但基于片段和结构的药物发现 (FBDD、SBDD) 已推进了治疗剂的鉴定和开发。FBDD 已促成 4 种药物(另有约 50 种药物处于临床前和临床开发阶段),而据估计,SBDD 已促成 200 多种 FDA 批准药物的开发。基于蛋白质 X 射线晶体学的片段库筛选,特别是针对难以捉摸或“无法用药”的靶标,可以同时生成命中结果以及蛋白质-配体相互作用和结合位点(正构或变构)的详细信息,从而为化学可处理性、下游生物学和知识产权提供信息。使用一种新型的高通量基于晶体学的片段库筛选平台,我们筛选了五种不同的蛋白质,命中率约为 2 e 8%,晶体结构约为 1.8 至 3.2 Å。我们考虑了当前的 FBDD/SBDD 方法和一些设计针对 DDR 核酸酶减数分裂重组 11(MRE11,又名 MRE11A)、无嘌呤/无嘧啶核酸内切酶 1(APE1,又名 APEX1)和 flap 核酸内切酶 1(FEN1)的抑制剂的示例性结果。© 2020 作者。由 Elsevier Ltd. 出版。这是一篇根据 CC BY-NC-ND 许可协议 ( http://creativecommons.org/licenses/by-nc-nd/4.0/ ) 开放获取的文章。
生活片段 - 阿罗尔森档案
这份教育和方法材料专门讲述了纳粹在俄罗斯苏维埃联邦社会主义共和国(前苏联的一部分,现为俄罗斯联邦)占领区犯下的罪行的历史。对苏联的灭绝战争是纳粹德国最严重的罪行之一。阿罗尔森档案馆收藏了大量有关俄罗斯纳粹迫害受害者的文件。关于那些被迫害、被驱逐到德国、被杀害或被强迫劳动的人的传记资料提供了对当时事件和受害者个人故事的深入了解。该教材以阿罗尔森档案馆关于各俄罗斯受害者群体的传记资料为基础,与俄罗斯大屠杀研究与教育中心合作编写,旨在用于历史教学。该材料包含个人文件,可阐明
生活片段 - 阿罗尔森档案
本教育和方法材料专门介绍了纳粹在俄罗斯苏维埃联邦社会主义共和国(前苏联的一部分,现为俄罗斯联邦)被占领土上犯下的罪行的历史。对苏联的歼灭战是纳粹德国最大的罪行之一。阿罗尔森档案馆包含大量有关纳粹迫害的俄罗斯受害者的文件。关于那些遭受迫害、被驱赶到德国、被杀害或在强迫劳动中被剥削的人的传记资料,让人们能够深入了解当时的事件和受害者的个人故事。该教育材料以阿罗尔森档案馆有关俄罗斯各受害者群体的传记资料为基础,与俄罗斯大屠杀研究和教育中心合作编写,旨在用于历史教学。该材料包含可阐明
基于片段的共价配体发现
受最近批准用于癌症治疗的共价激酶抑制剂 (TKI) 的启发,共价探针和药物的开发经历了一场复兴,现在吸引了工业界和学术界的浓厚兴趣,包括针对 EGFR 的抑制剂:阿法替尼 (Gilotrif) 和奥希替尼 (Tagrisso) 或 BTK:阿卡替尼 (Calquence) 和伊布替尼 (Imbruvica)。1–4 与暂时靶向保守底物和/或变构结合位点的非共价小分子不同,共价抑制剂通常在效力、选择性、药代动力学和药效学方面表现出差异化的药理学,因为它们能够与靶蛋白形成不可逆的共价键。 5,6 尽管有这些优点,许多人仍然对共价抑制剂持怀疑态度,因为它们会产生能够引发特异性免疫反应和过敏/超敏反应的蛋白质加合物。7,8 从历史上看,共价药物的发现
嘧啶抑制剂靶向基孔肯尼亚病毒NSP3大域的基于片段的药物设计
NSP3(NSP3MD)的大域在甲虫病毒中高度保守,而Chikungunya病毒(CHIKV)NSP3MD的ADP-核糖基水合酶活性对于Chikv病毒复制和毒力至关重要。迄今已确定针对CHIKV NSP3的小分子药物。在这里,我们报告了与NSP3MD结合的小片段,这些片段实际上是通过筛选片段和X射线晶体学来解决的。这些鉴定出的片段具有相似的支架,2-吡啶酮-4-羧酸,并特别结合了NSP3MD的ADP-核糖结合位点。Among the fragments, 2-oxo-5,6-benzo- pyrimidine-4-carboxylic acid showed anti-CHIKV activity with an IC 50 of 23 μ M. Our frag- ment-based drug discovery approach provides valuable information to further develop a specific and potent nsP3 inhibitor of CHIKV viral replication based on the 2-pyrimidone-4- carboxylic acid scaffold.表明,这种嘧啶支架也可以与其他α病毒和冠状病毒的大域结合,因此具有潜在的抗病毒活性。
CRISPR-CAS13酶学在临床环境中迅速检测到SARS-COV-2片段
Wahab A. Khan 1,2 Rachael E. Barney 1,Gregory J. Tsongalis 1,2 * 1)病理学和实验室医学系,Audrey and Theodore Geisel医学院,达特茅斯学院,美国新罕布什尔州,美国新罕布什尔州03755,美国。 2)美国NH 03756,Dartmouth Hitchcock Medical Center,Dartmouth Hitchcock Medical Center的病理与实验室医学系临床基因组学和先进技术实验室; * 相应的。gregory.j.tsongalis@hitchcock.org