XiaoMi-AI文件搜索系统
World File Search System视觉特征前额编码的个体差异
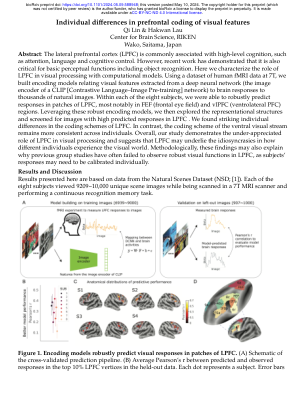
摘要:外侧前额叶皮层 (LPFC) 通常与高级认知有关,例如注意力、语言和认知控制。然而,最近的研究表明,它对于包括物体识别在内的基本感知功能也至关重要。在这里,我们用计算模型描述了 LPFC 在视觉处理中的作用。使用 7T 的人类 fMRI 数据数据集,我们建立了编码模型,将从深度神经网络(CLIP [对比语言-图像预训练] 网络的图像编码器)中提取的视觉特征与大脑对数千张自然图像的反应联系起来。在八个受试者中,我们能够稳健地预测 LPFC 斑块中的反应,最明显的是 FEF(额叶眼场)和 vlPFC(腹外侧 PFC)区域。利用这些强大的编码模型,我们随后探索了表征结构并筛选了 LPFC 中具有高预测反应的图像。我们发现 LPFC 的编码方案存在显著的个体差异。相比之下,腹侧视觉流的编码方案在个体之间保持更一致。总体而言,我们的研究证明了 LPFC 在视觉处理中的作用未被充分重视,并表明 LPFC 可能是不同个体体验视觉世界的独特之处的基础。从方法论上讲,这些发现也可能解释了为什么以前的团体研究往往未能观察到 LPFC 中强大的视觉功能,因为受试者的反应可能需要单独校准。
使用纵向变分自动编码器对图像数据进行进展模型
摘要。疾病进展模型对于理解退行性疾病至关重要。混合效应模型一直用于模拟临床评估或从医学图像中提取的生物标志物,允许在任何时间点进行缺失数据的填补和预测。然而,这种进展模型很少用于整个医学图像。在这项工作中,变分自动编码器与时间线性混合效应模型相结合,以学习数据的潜在表示,使得各个轨迹随时间遵循直线,并以一些可解释的参数为特征。设计了一个蒙特卡罗估计器来迭代优化网络和统计模型。我们将此方法应用于合成数据集,以说明时间依赖性变化与受试者间变异性之间的分离,以及该方法的预测能力。然后,我们将其应用于来自阿尔茨海默病神经影像计划 (ADNI) 的 3D MRI 和 FDG-PET 数据,以恢复大脑结构和代谢改变的详细模式。
Andrew V. Sutherland 稀疏的自动编码器用于增强学习模型中的可解释性
美国专利US-6609117-B2和US-6349292-B1,“通过公共网络分配邮资的系统和方法,可以有效地打印邮政指示,并在要邮寄的项目上进行邮寄并进行身份验证,” A.V.Sutherland,M.R。Klugerman,F.M。 d'ippolito,2002和2003。Klugerman,F.M。d'ippolito,2002和2003。
2025 Premier的编码挑战 - 课程对齐与...
数字技术中的学习重点是进一步发展计算思维中的理解和技能,例如对程序进行分类和概述;并越来越了解如何在家中,在学校和当地社区中使用数字系统并可以使用数字系统。到4年底,学生将有机会创建一系列数字解决方案,例如涉及用户选择的互动冒险,对简化的现实世界系统建模和简单的猜测游戏。在第3年和第4年,学生根据其组件探索数字系统,以及数字显微镜,相机和交互式白板等外围设备。他们收集,操纵和解释数据,发展对数据特征及其表示的理解。使用抽象的概念,学生使用诸如总结事实来推断结论等技术来定义简单的问题。他们通过文本和图表记录了简单的解决方案,并发展其设计技能,从最初遵循准备的算法到描述自己的支持分支(选项选择)和用户输入。他们的解决方案是使用适当的软件(包括使用图形元素而不是文本说明的视觉编程语言)实现的。,他们一般而言解释了他们的解决方案如何满足特定需求,并考虑社会如何使用数字系统以环境可持续的方式满足需求。在教师指导的情况下,学生识别并列出完成任务或项目所需的主要步骤。在在线环境中分享想法并进行交流时,他们对为什么要考虑受众的感受并运用班级同意的安全实践和社会协议的理解,以表现出尊重的行为。
基于相变材料光激活的可重构编码超表面用于太赫兹光束操控
图 2. 所提出的光控编码元件的设计和特性。a) 元原子编码元件的详细结构,在 SiO 2 基板上构建了 1 μm 厚的金方块和 1 μm 厚的 GeTe 方块图案。b) 编码元件两种状态的示意图:状态“0”表示 GeTe 的非晶态(绝缘态),状态“1”表示 GeTe 的晶体(导电)态。c) 和 d) 两种状态下编码元件的相应反射特性(c 幅度和 d 相位)。e) GeTe 层表面电阻随温度的变化(双探针测量),显示两种状态下的电特性相差六个数量级以上,并且冷却至室温时晶体状态具有非挥发性行为。 f) 有限元模拟 GeTe 层在具有不同能量密度的 35 纳秒长单脉冲紫外激光照射下的温度上升情况:单脉冲的通量为 90 mJ/cm 2,将使最初为非晶态的 GeTe 的温度升至其结晶温度 ( TC ) 以上,而随后的 190 mJ/cm 2 激光脉冲将使 GeTe 的温度升至其局部熔化温度 TM 以上,并将材料熔化淬火回非晶态。下图是拟议的 1 比特元原子的配置和示意图
Edna元编码时间抽样策略对沿海生物多样性检测的影响
生物时间序列观测对于更好地理解生态过程并确定人类对海洋的影响至关重要(Ducklow等,2009;BáLint等,2018; Takahashi等,2023)。有效进行了有效的海洋监测计划,有时使用数十年来收集的时间序列(Fontaine and Rynearson,2023年)。环境DNA(EDNA)从水样品中进行的元法编码越来越多地用于监测沿海生物多样性并检测随着时间的推移生物群落的变化(Deiner等,2017; Mathieu等,2020)。现在,通过使用EDNA METABARCODING或其他生物分子技术(https://obon-ocean.orgean.org/about/),建立了诸如海洋生物分子观测网络(OBON)之类的程序,以通过全球规模的合作和长期研究来增强海洋生物监测。为了确定在不同的时间尺度和环境条件上是否存在稳定的,复发的EDNA检测,对环境中的埃德娜(Edna)如何随物种物候(例如,生命阶段,生殖和代谢)和物理过程(例如水动力学,温度,uv)(seymour,uv)(Seymour,2019; des souza; de 22; eve and and and 2016; eve; et e and; et e and and;这种知识对于对长期EDNA数据趋势的有意义解释也至关重要。越来越多的研究报告了EDNA检测峰在短季节内的窗口中,并将这种模式归因于生物学因素(Laramie等,2015; Sigsgaard等,2017; Stoeckle等,2017; Handley等,2019; Handley等,2019; 2019; Troth et al。,2021; 2021; Sevellec et al。虽然有几项研究报道了用埃德娜(Edna)检测到的社区的显着年度变化(Closek等,2019; Laporte等,2021; di Capua等,2021; Carvalho等,2024),2024年),很少有短期变量(Kelly et al。,2018 al。等人,2024年)以及自然的短期可变性如何影响我们解释沿海EDNA数据以评估社区结构随时间变化的能力。水的时间系列edna metabarcoding提供了沿海北极生物监测的重要潜力。北极海洋正经历着由物理转变驱动的深刻气候和相关的生物变化,包括海冰熔化,海温升高和运输活动增加(Garcia-Soto等,2021; Murray等,2024)。尽管对北极生物群进行测量的后勤挑战,其中许多是地方性的,但已经记录了海洋社区的快速变化(Post等,2009; Koenigstein,2020)。Edna Metabarcoding跨多个营养水平检测生物的能力使其成为这个广阔而偏远地区的宝贵工具(Lacoursière-Roussel等,2018; Leduc等,2019; Sevellec等,Sevellec等,2021; Geraldi等,Geraldi等,2024)。这种非侵入性方法也是生物监测海洋社区的最伦理方法之一,使其在敏感的北极地区特别有价值。为了充分表征生物多样性中的长期闪烁,我们仍然需要理解北极地区海洋生物多样性的季节性和季节性季节性模式。在这里,我们比较了使用加拿大北极丘吉尔港作为案例研究的不同时间抽样策略,以监测埃德娜的后生社区,目的是
正交表示用于估计因果量的学习 生成的AI和创造性工作:叙事,价值观和影响 triffid:提高第一响应者效率的自动机器人辅助 微守流:边缘设备的复杂性自我注意力低的视觉变压器 covid- ... 之间的不可思议和争议性溢出 arxiv:2502.10437v1 [q-bio.pe] 2025年2月9日 对表示漏洞和工程强大的视觉变形金刚的机械理解 arxiv:2502.05610v1 [CS.CL] 2025年2月8日 Goproteingnn:利用蛋白质知识图蛋白表示学习 台式4Merge:综合基准,用于合并逼真的密集交通与微交互式车辆 arxiv:2502.08704v1 [cond-mat.supr-con] 2025年2月12日 机器人引发的久坐行为的社会控制 增强数字阴影的稳健性,用于使用增强岩石物理建模的二氧化碳存储监控 arxiv:2502.07964v1 [cs.lg] 2025年2月11日 对石墨烯中振荡大声电效应的相干检测 arxiv:2502.09316v1 [CS.CL] 2025年2月13日 将基于物理的建模与锂离子电池的机器学习相结合 Trellis BMA:DNA存储的IDS通道上的编码跟踪重建 arxiv:2502.07650v1 [stat.ml] 2025年2月11日 与Soc- ... 的多间隔能量储备合作
表示学习被广泛用于观察数据的因果量(例如,有条件的平均治疗效应)。尽管现有的表示学习方法具有允许端到端学习的好处,但他们没有Neyman-Ottrol-ottrodenal学习者的理论特性,例如Double Ro-Busberness和Quasi-Oracle效率。此外,这种表示的学习方法通常采用诸如平衡之类的规范约束,甚至可能导致估计不一致。在本文中,我们提出了一类新型的Neyman-Ottrodonal学习者,以在代表水平上定义的因果数量,我们称之为或称为校友。我们的旅行者具有几个实际的优势:它们允许基于任何学习的表示形式对因果量进行一致的估计,同时提供了有利的理论属性,包括双重鲁棒性和准门的效率。在多个实验中,我们表明,在某些规律性条件下,我们的或学习者改善了现有的表示学习方法并实现最先进的绩效。据我们所知,我们的或学习者是第一批提供代表学习方法的统一框架,而Neyman-ottrol-ottrodenal学习者进行因果量估计。
方面能源管理系统中的硬编码凭证
abb不提供明示或暗示的保修,包括针对本文档中包含的信息的适销性和适用性的保证,并且对本文档中可能出现的任何错误都不承担任何责任。在任何情况下,ABB或其任何供应商均不应对使用本文档的使用或使用本文档中所述的任何硬件或软件的使用,即使ABB或其供应商已被告知有此类损害的可能性,也对本文档中所述的任何硬件或软件的使用,或使用本文档中描述的任何硬件或软件的使用,对任何性质或类似的损害均不承担任何责任。
通过 LHCII 编码基因的多重基因组编辑揭示类囊体基粒堆积
HAL 是一个多学科开放存取档案库,用于存放和传播科学研究文献,无论这些文献是否已出版。这些文献可能来自法国或国外的教学和研究机构,也可能来自公共或私人研究中心。
衰老心力衰竭的非编码和编码机制,具有甲状腺功能障碍的射血分数
(未通过同行评审认证)是作者/资助者。保留所有权利。未经许可就不允许重复使用。此预印本版本的版权持有人于2025年2月8日发布。 https://doi.org/10.1101/2025.02.04.636476 doi:Biorxiv Preprint






![正交表示用于估计因果量的学习
生成的AI和创造性工作:叙事,价值观和影响
triffid:提高第一响应者效率的自动机器人辅助
微守流:边缘设备的复杂性自我注意力低的视觉变压器
covid- ... 之间的不可思议和争议性溢出
arxiv:2502.10437v1 [q-bio.pe] 2025年2月9日
对表示漏洞和工程强大的视觉变形金刚的机械理解
arxiv:2502.05610v1 [CS.CL] 2025年2月8日
Goproteingnn:利用蛋白质知识图蛋白表示学习
台式4Merge:综合基准,用于合并逼真的密集交通与微交互式车辆
arxiv:2502.08704v1 [cond-mat.supr-con] 2025年2月12日
机器人引发的久坐行为的社会控制
增强数字阴影的稳健性,用于使用增强岩石物理建模的二氧化碳存储监控
arxiv:2502.07964v1 [cs.lg] 2025年2月11日
对石墨烯中振荡大声电效应的相干检测
arxiv:2502.09316v1 [CS.CL] 2025年2月13日
将基于物理的建模与锂离子电池的机器学习相结合
Trellis BMA:DNA存储的IDS通道上的编码跟踪重建
arxiv:2502.07650v1 [stat.ml] 2025年2月11日
与Soc- ... 的多间隔能量储备合作](/simg/3/371e53cea55d57b21dc3cec4490fd2d843578c28.webp)


