XiaoMi-AI文件搜索系统
World File Search System发件人:海军记录修正委员会主席 收件人:海军部长 主题:海军记录 ICO 审查,USN,XXX-XX- 参考:(a) 第 10 章 USC § 1552 (b) OPNAVINST 7220.14,2005 年 12 月 24 日 (c) DoD 7000.14-R FMR 第 7A 卷,第 18 章 附件:(1) DD 表格 149 及其附件 (2) OCNO 的咨询意见(N130C3),2023 年 3 月 2 日 (3) 主体的海军记录 1. 根据参考(a)的规定,主体(以下简称请愿人)向海军记录修正委员会(委员会)提交了附件(1),要求更正其海军记录,以表明请愿人在 2021 年 7 月 8 日至2022 年 9 月 30 日。 2. 委员会由 、 和 组成,于 2023 年 3 月 9 日审查了申诉人的错误和不公正指控,并根据其规定,确定应根据现有的记录证据采取下文指示的纠正措施。委员会考虑的文件材料包括附件、申诉人海军记录的相关部分以及适用的法规、条例和政策。 3. 在向委员会提出申请之前,申诉人已用尽海军部现行法律和法规规定的所有行政补救措施。委员会审查了与申诉人的错误和不公正指控有关的所有记录事实后,发现如下:a. 2017 年 1 月 18 日,申诉人开始服现役。b. 2018 年 5 月 21 日,申请人在驻扎期间收到正式的变更任务命令(BUPERS 命令:1418),离开日期为 2018 年 6 月。申请人的最终活动是执行任务,抵达日期为 2018 年 7 月 9 日,预计轮换日期 (PRD) 为 2022 年 7 月。c. 2018 年 6 月 21 日,申请人从 调至 ,并于 2018 年 7 月 8 日抵达 执行任务,并开始领取职业海上工资。
发件人:海军记录修正委员会主席 收件人:海军部长 主题:海军记录 ICO 审查,USN,XXX-XX- 参考:(a) 第 10 章 USC § 1552 (b) OPNAVINST 7220.14,2005 年 12 月 24 日 (c) DoD 7000.14-R FMR 第 7A 卷,第 18 章 附件:(1) DD 表格 149 及其附件 (2) OCNO 的咨询意见(N130C3),2023 年 3 月 2 日 (3) 主体的海军记录 1. 根据参考(a)的规定,主体(以下简称请愿人)向海军记录修正委员会(委员会)提交了附件(1),要求更正其海军记录,以表明请愿人在 2021 年 7 月 8 日至2022 年 9 月 30 日。 2. 委员会由 、 和 组成,于 2023 年 3 月 9 日审查了申诉人的错误和不公正指控,并根据其规定,确定应根据现有的记录证据采取下文指示的纠正措施。委员会考虑的文件材料包括附件、申诉人海军记录的相关部分以及适用的法规、条例和政策。 3. 在向委员会提出申请之前,申诉人已用尽海军部现行法律和法规规定的所有行政补救措施。委员会审查了与申诉人的错误和不公正指控有关的所有记录事实后,发现如下:a. 2017 年 1 月 18 日,申诉人开始服现役。b. 2018 年 5 月 21 日,申请人在驻扎期间收到正式的变更任务命令(BUPERS 命令:1418),离开日期为 2018 年 6 月。申请人的最终活动是执行任务,抵达日期为 2018 年 7 月 9 日,预计轮换日期 (PRD) 为 2022 年 7 月。c. 2018 年 6 月 21 日,申请人从 调至 ,并于 2018 年 7 月 8 日抵达 执行任务,并开始领取职业海上工资。
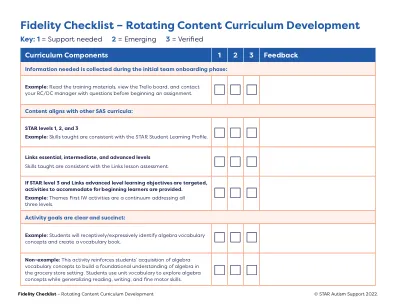
保真度检查表 – 轮换内容课程开发
非示例:此活动强化学生对代数词汇概念的掌握,以在杂货店环境中建立对代数的基本理解。学生使用单位词汇探索代数概念,同时概括阅读、写作和精细运动技能。
慢性粒细胞白血病模型显示,轮换使用普纳替尼和伊马替尼可暂时提高伊马替尼的疗效
慢性粒细胞白血病 (CML) 的靶向疗法有效,但很少能治愈。患者通常需要无限期治疗,这为药物耐药性的产生提供了充足的时间。耐药性问题是 CML 导致死亡的主要原因之一,因此任何预防耐药性的方法都很重要。药物轮换,即定期在不同药物之间切换治疗,就是这样一种选择,理论上可以延缓耐药性的发生。药物轮换疗法的体外测试是将其应用于动物或人体试验的第一步。我们开发了一种在 CML 细胞系中测试药物轮换方案的方法,该方法基于用适量的抑制剂培养细胞,中间穿插清洗程序和药物交换。在 CML 特异性细胞系 KCL-22 中评估了伊马替尼和普纳替尼的药物轮换。药物轮换最初减少了 KCL-22 细胞的生长,但细胞最终适应了该方案。我们的结果表明,在药物轮换中,普纳替尼会暂时使细胞对伊马替尼敏感,但这种效果是短暂的,经过几个治疗周期后最终会消失。本文讨论了这一观察结果的可能解释。
通过站点轮换实现学生学习个性化
我们的行动理论融合了个性化学习的四个基本要素(如上所述),这些要素必须到位。具体来说,必须有适当的技术设备;与课程保持一致的数字学习材料,让学生有更大的控制权;形成性评估数据;以及帮助教学人员使用这些资源的培训。这些投入是站点轮换模式的基础,旨在推动积极成果。与站点轮换相关的短期成果包括增加差异化教学,从而推动内容和技能的学习改进。中期成果包括更有动力和参与度的学生、更高的教师满意度和更好的课堂管理。成功的站点轮换模式的长期成果包括提高学生成绩和教师保留率,减少学生行为问题。(完整的行动理论见附录 A。)
CRUK 剑桥中心 MRes 轮换项目
轮换项目名称 使用 100 万个可诱导 DNA 条形码进行原位谱系追踪实验室主任 (PI) 姓名 Jamie Blundell 第二位指导老师(如适用) N/A 项目早期检测指导老师电子邮件 jrb75@cam.ac.uk 实验室位置 哈奇森 MRC 研究中心项目概要目的和目标维持血液、皮肤、肠道和其他组织的干细胞处于不断更新的状态,从而积累基因改变,其中一些导致克隆扩增和癌症 [1]。理解这一点需要能够测量组织维持期间发生的群体动态。在此,我们建议构建一个原位谱系追踪工具,该工具可以诱导生成数百万个 DNA 条形码组合,从而允许人们使用下一代测序以精确度并行追踪数百万个细胞谱系。与以前的半定量方法 [2] 不同,这项技术将能够定量追踪与体内组织维持相关的克隆动态,并深入了解如何实现体内平衡以及它在癌症早期阶段如何崩溃。我们之前在酿酒酵母中的工作已经证明,基于 cre-lox 系统的位点特异性 DNA 条形码和谱系动态的定量追踪可用于深入了解突变如何在大量细胞群体中产生、扩展和竞争 [3]。我们与长期合作伙伴 Sasha Levy 进一步开发了这项技术,现在可以原位生成条形码多样性,而无需转化质粒文库。这项改进的技术将利用 3 个串联 loxP“着陆垫”,每个“着陆垫”(在 Cre 诱导后)可以不可逆地整合存储在基因组其他地方的三个独立串联阵列中的约 100 个独特条形码序列中的一个。对于这个 MRes 轮换项目,我们计划扩大这项技术的规模,以在酵母中稳健地生成 100 万个独特的条形码组合。这将证明该技术能够以单细胞精度追踪体内细胞谱系,从而为干细胞生物学和癌症发病中的主要未解问题提供参考。实验计划 学生将首先构建由 loxP 位点分隔的约 100 个条形码组成的长串联阵列构建体,并使用标准同源重组将此构建体整合到已包含 cre-lox 着陆垫的酵母菌株的基因组中。然后,学生将研究此构建体可诱导的条形码多样性如何取决于串联阵列的诱导条件和基因组位置。优化后,学生将整合另外两个串联阵列,并尝试实现超过 100 万个独特条形码的多样性,将使用定制设计的 2 步 PCR 协议进行仔细量化,该协议使用唯一分子标识符 (UMI) 来标记单个 DNA 分子。