XiaoMi-AI文件搜索系统
World File Search System引文:Pietra D、Brisci A、Rumi E、Boggi S、Elena C、Pietrelli A、Bordoni R、Ferrari M、Passamonti F、De Bellis G、Cremonesi L 和 Cazzola M. Dee
对于 HRM 检测,采用补充表 S1 中报告的优化内含子引物。PCR 在 20 μ L 中进行,其中包含 100 ng DNA、0.5 单位 HotStart Taq 聚合酶以及 1x 缓冲液(Qiagen,德国希尔登)、1.5 mM MgCl 2、800 μ M dNTP、300 nM 每种引物和 1x EvaGreen(Idaho Technologies,犹他州盐湖城)作为插入染料。循环和 HRM 分析在 Rotor-Gene ™ 6000 实时分析仪上进行,采用以下热方案:95°C 持续 15 分钟(一个循环);95°C 持续 30 秒,55°C 持续 30 秒,72°C 持续 30 秒(50 个循环);72°C 持续 10 分钟(一个循环);熔化温度从 85°C 升至 95°C,每秒上升 0.1°C。使用相关的 Rotor-Gene ™ 6000 系列软件 (v1.7.87) 分析数据。标准化条在前导范围的 88°C 和 88.5°C 之间,在尾随范围的 92.5°C 和 93°C 之间,置信阈值为 90%:如果 HRM 图超出了指定参考基因型的置信范围,则软件会将样本识别为变异。图 S1A 显示了健康受试者和 3 名 MPN 患者的 DNA 样本的 HRM 图谱,这些样本先前已通过微电子微芯片分析进行了基因分型(未显示数据)。患者 PV02_113 为 MPL (W515K) 纯合子(TGG>AAG 转换),其 HRM 曲线相对于野生型序列向左移向较低温度,这与纯合变体导致熔解温度 (Tm) 降低的预期一致。患者 PV04_494 为 MPL (W515A) 纯合子(TGG>GCG 转换)等位基因
evolu&on Sweden 2025
亲爱的会议par+cipant,我们期待您在2025年1月13日至15日在Linköping举行的瑞典会议上欢迎您参加Evolu+!此PDF包含会议的摘要(口头和海报),这是一个初步的(希望最终,但任何更改都将在Google Docs excelle文件上更新并发送给所有人)。该会议将在林克平大学的Valla(Main)校园的C构建中举行。会议将在C4演讲剧院举行,海报在C4外的门厅区设置。registra+On于1月13日在演讲厅外9.30 Un+L 13.00。在大学餐厅Kårallen(请参阅下一页的地图)有一个午餐场地,该餐厅位于下一栋楼,距离C建造(距演讲厅两分钟步行路程)。我们已经为所有那些表示想要储层+的人预订了一个Reserva+(很可能会容纳任何原谅或改变主意的人)。午餐的费用是每个人自费。在Addi+On上,我们将在Fimba Breaks中提供茶,咖啡和饼干,并由Qiagen慷慨地提供(在会议上停留在他们的摊位上,打招呼!)。我们还要感谢Scilifelab慷慨的全体扬声器,以及HOS+NG A DDLS扬声器会议。我们在演讲剧院外的第13届和14日举行了海报会议,还强调了在镇上(O'Leary's)的会议酒吧。这是因为它的尺寸而选择的,但是我们没有任何实际的储层+ons(但内部效果很大)。它位于主广场(Storatorget),其他几家酒吧也位于主广场或下一街(Ågatan)上,供那些明智的人在晚上见面喝酒的人。我们希望大家度过一个愉快的圣诞节和新的一年,并期待所有的兴趣+NG的演讲,并在新的一年中与所有人见面。最好的Evolu+On在瑞典Linköping组织Commivee Dominic Wright(主席),Rie Henriksen,Per Jensen,Jensen,Urban Friberg和Krzysztof Bartoszek
使用第三代测序优化从鼻衬液中提取DNA,以评估鼻微生物组
背景:通过鼻吸附对鼻衬液(NLF)采样最少侵入性且耐受性良好,但是使用此技术评估鼻微生物组的可行性尚不清楚。但是,低生物量使气道样品特别容易受到与污染物DNA有关的问题。在这项研究中,我们评估了使用方法学对低生物量呼吸样品分离的DNA的适用性,并评估了与传统的拭子采样方法相比,通过鼻吸附收集的衬里液的衬里如何捕获鼻微生物的多样性和组成。方法:从成年志愿者那里收集鼻拭子和NLF。DNA。评估DNA的质量和数量,并进行了短阅读16S rRNA测序,以评估可行性和提取偏见。然后使用优化的提取方法从NLF和鼻拭子中提取DNA,并且进行了全长16S rRNA测序,以比较NLF和鼻拭子之间的微生物谱。使用NF核/Ampliseq管道,PacificBiosciences/PB-16S-NF管道或软件EMU分类分类法分类,并使用R Packages Temages and Mixomics进行下游分析。结果:所有提取方法均从模拟群落中恢复了DNA,但仅基于降水的方法从NLF产生了足够的DNA。提取方法显着影响微生物谱,需要机械裂解以最大程度地减少针对特定属的偏差。曲线与长读测序相当。结论:我们的发现证明了使用通过鼻吸附收集的NLF分析鼻微生物组的可行性,并验证了两种提取方法,作为适合全长的16S rRNA测序的低生物量呼吸类样品的RRNA测序。我们的数据证明了在低生物量呼吸样品中无偏DNA提取方法的重要性,以及随后DNA提取对观察到的微生物谱的影响。此外,我们证明了NLF可能是使用16S rRNA测序评估鼻拭子的适当替代样品。
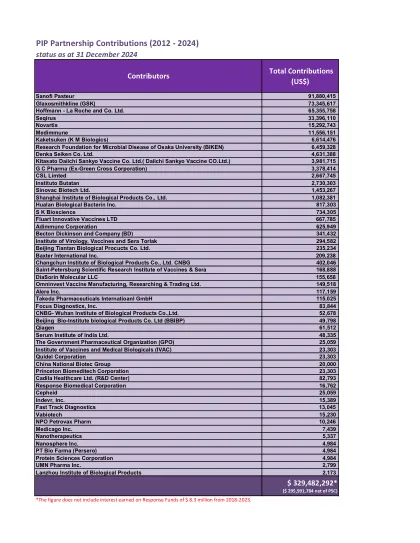
PIP 合作伙伴贡献(2012 - 2024)
赛诺菲巴斯德 91,880,415 葛兰素史克(GSK) 73,345,617 霍夫曼-罗氏公司 65,355,758 Seqirus 33,396,110 诺华 15,292,743 Medimmune 11,556,151 Kaketsuken(KM Biologics) 6,614,476 大阪大学微生物疾病研究基金会(BIKEN) 6,459,328 Denka Seiken Co. Ltd. 4,631,388 北里第一三共疫苗有限公司(Daiichi Sankyo Vaccine CO.Ltd.) 3,981,715 GC Pharma(前绿十字公司) 3,378,414 CSL Limted 2,667,745布塔坦研究所 2,730,303 科兴生物制品有限公司 1,453,267 上海生物制品研究所有限公司 1,082,381 华兰生物疫苗股份有限公司 817,303 SK Bioscience 734,305 Fluart Innovative Vaccines LTD 667,785 Adimmune Corporation 625,949 Becton Dickinson and Company (BD) 341,432 病毒、疫苗和血清研究所 Torlak 294,582 北京天坛生物制品股份有限公司 235,234 百特国际公司 209,238 长春生物制品研究所有限公司 CNBG 402,046 圣彼得堡疫苗和血清科学研究所 168,888 DiaSorin Molecular LLC 155,658 Omninvest Vaccine Manufacturing, Researching & Trading Ltd. 149,518 Alere Inc. 117,159 Takeda Pharmaceuticals Internatioanl GmbH 115,025 Focus Diagnostics, Inc. 83,844 CNBG-武汉生物制品研究所有限公司52,678 北京生物研究所生物制品有限公司(BBIBP) 49,798 Qiagen 61,512 印度血清研究所有限公司 48,335 政府制药组织(GPO) 25,059 印度疫苗和医学生物制品研究所(IVAC) 23,303 Quidel Corporation 23,303 中国生物技术集团 20,000 Princeton Biomeditech Corporation 23,303 卡迪拉医疗保健有限公司(研发中心) 82,793 Response Biomedical Corporation 16,762 Cepheid 25,059 Indevr, Inc. 15,389 Fast Track Diagnostics 13,045 Vabiotech 15,230 NPO Petrovax Pharm 10,246 Medicago Inc. 7,439 Nanotherapeutics 5,337 Nanosphere Inc. 4,984 PT Bio Farma (Persero) 4,984 Protein Sciences Corporation 4,984 UMN Pharma Inc. 2,799 兰州生物制品研究所 2,173
混合轻型态在拓扑超导体中与空腔光子搭配的
细菌“ candidatus nardonella dyophthoridicola”是一种革兰氏阴性的gam- maproteotototototabterial tocyobterial tocytobiont(图。1)。特别是,它是与象鼻虫相关的细胞内义务共同主义者(1)。通过向其宿主供应酪氨酸,细菌在表皮中起着至关重要的作用(2)。与第二个象鼻虫相关的符号不同,“ candidatus sodalis pierantonius”,它在宿主的整个生命周期中保持在功能性细菌中(3-5)。我们使用长阅读测序来研究“ Ca.nardonella dryophthoridicola”菌株nardrf,与意大利人种群相关的Rhynchophorus ferrugineus。2017年,昆虫宿主是从卡塔尼亚地区的一棵棕榈树中取样的。p在25°C,黑暗的24小时内,直到分成人。剖析了十个新出现的成年人以提取其细菌。然后按照制造商的动物组织提取说明,使用Dneasy血液和组织试剂盒(意大利Qiagen,意大利)合并细菌以进行DNA提取。在90V时通过0.8%琼脂糖凝胶电泳对DNA完整性进行了1H的验证。用纳米体100分光光度计(意大利的Thermo Fisher Scienti)和Qubit双链DNA(DSDNA)高敏化测定试剂盒测量了DNA纯度和浓度。使用R9.5流单元在奴才MK1B设备上进行了长阅读测序。使用Minknow V18.03.1进行测序48小时。读取量超过500 bp进行后续分析。重点识别为“ Ca.用于图书馆制备,使用1D连接测序试剂盒(SQK-LSK 108)原始Col使用了2.5 m g的非大量和非大小选择的总基因组DNA。然后,将最终DNA的0.5 m g加载到流动细胞上。基本调用,具有高准确性算法,质量截止值为7。所有工具均使用默认参数运行,除非另有说明。使用min-iasm(7)组装了元基因组fastq读取(主机和共生体)。nardonella dyophthoridicola”,以ncbi非冗余(NR)数据库进行鉴定。提取这些概念并用于重新填充组件。重叠群用于映射和提取“ Ca.nardonella dryophthoridicola”使用minimap2 v2.17(8)。然后使用Flye v2.8.1(9)重新组装836,116读。使用Circlator v1.5.5(10)与选项进行了循环 - Merge_Min_ID 85和 - Merge_breaklen 1000,如牛津Nanopore读取。使用公开的Illumina简短读数(SRA登录
使用...对混合样本进行 DNA 元条形码编码 - Aguirre Lab
牛津纳米孔 Flongle 简介:本方案描述了我们使用纳米孔 Flongle 进行 DNA 元条形码编码的方法。它涵盖了纳米孔测序和 DNA 元条形码编码的简要背景、我们为元条形码编码设计的引物、我们的 PCR 方法、纳米孔文库制备和样品加载以及使用 Ontbarcoder 应用程序进行的数据分析。牛津纳米孔测序:牛津纳米孔测序仪 1 是第三代实时长读测序仪,越来越受欢迎。它相对便宜(起价 1000 美元),小巧便携,可生成长读长(1000 个碱基对),并实时测序,这意味着您可以在测序反应进行时下载和分析序列数据。纳米孔测序的工作原理是检测 DNA 穿过纳米孔时流动池上纳米孔中电荷的变化。DNA 核苷酸(A、C、T、G)在穿过纳米孔时会以不同的方式改变电荷,因此机器可以根据孔电荷的变化确定 DNA 链的序列。 Flongle:纳米孔流动槽有两种类型,常规流动槽适用于大型项目(成本约为 1,000 美元),Flongle 2 流动槽适用于小型实验(每个流动槽成本约为 90 美元)。虽然 Flongle 流动槽成本不算太高,但对单个样本进行测序还是太贵了。常规 Sanger 测序每个样本的成本为 2-6 美元!因此,必须将样本汇集在一起进行测序,也就是说,将几个或多个样本一起装入单个 Flongle 流动槽中。为了稍后分离样本,需要用条形码标记样本,以便识别它们。DNA 宏条形码:DNA 条形码是使用参考序列来识别物种。对指定的条形码基因(传统上是线粒体 COI 基因)进行测序,然后将获得的序列与条形码序列数据库进行比较。DNA 宏条形码是指在单个测序反应中汇集许多个体,以使用 DNA 条形码识别物种。 Nanopore 测序仪可用于 DNA 宏条形码,并在一次测序运行中生成多个样本的序列。我们实验室中的 DNA 宏条形码:在我们的实验室中,我们使用带有 Flongle 流动槽的 Nanopore 测序仪进行 DNA 宏条形码。使用苯酚-氯仿 3 、Qiagen 4 甚至 Chelex 5(昆虫)方案提取 DNA。然后我们进行 PCR 以扩增 COI DNA 条形码基因(也可以使用其他基因,如 12S 和 16S 6 ),在琼脂糖凝胶上运行产物以查看如何
限制摘要和琼脂糖凝胶电泳
T ECHNIQUES I N M OLECULAR B IOLOGY – R ESTRICTION D IGEST AND A GAROSE G EL E LECTROPHORESIS This lab will introduce you to DNA modification by restriction enzymes using the purified plasmids you prepared from your transformation.我们还将使用水平凝胶电泳对纯化的质粒和消化进行分析。您将对限制酶进行切割之前和之后对纯化的质粒进行凝胶分析。您将执行质粒的模拟(控制)摘要,一种具有两种不同限制酶和双重摘要的单个摘要。从中,您应该能够确定Qiagen微型制备质粒DNA的纯度,以及在PQE60载体中的WGMDH插入物。一定要阅读有关限制摘要(教科书读数)和琼脂糖凝胶(教科书和讲义)的信息,以了解每个概念的理论,并了解执行成功实验所需的细节。必需的视频 - 新英格兰Biolabs准备的网页上的限制酶链接。观看所需的最小视频是(具有限制性酶的消化,限制酶消化的标准协议和NEB限制酶Double Digigest协议视频)。在进行实验之前,您还应该查看其他几个。https://www.neb.com/applications/cloning-‐and-‐synthetic-‐ biology/dna-‐preparation/restriction-‐enzyme-‐digestion Practical notes on Restriction Endonucleases (RE) and Their Use Enzymatic Unit definition: 1 Unit = amount of enzyme necessary to digest 1 µg DNA in 1 hr (37°C, with适当的缓冲区)。glycerol Re Digests。确保您使用的是正确的缓冲液,酶与底物的正确比率(DNA和正确消化的条件)。Rextion缓冲每个限制酶具有一个缓冲液,其中最高活性(通常为10倍浓缩物)。某些酶具有共同的缓冲液,而其他酶则需要与独特的缓冲液一起使用以进行最佳活动。几家公司(以新英格兰的Biolabs为例)具有与多种酶合作的共同缓冲液。您的网站上有一个链接 - 探索几家公司以了解缓冲液和酶。存储条件RE通过反复暴露于较高的温度来对活动丧失敏感;使用库存以长期存储保持在-20°C,在使用时在〜0°C(在冰上)处于〜0°C(在冰上)。进行消化时,温育几个小时后,某些酶的活性就会损失。由于RE昂贵,因此必须谨慎处理库存。酶应在-20°C时不断存储在非冻结冰箱中。(无霜的冰柜定期在冰点上加热以限制冰的积累。)也最好将酶放在冰箱中的绝缘容器中,该酶在打开冰柜时会限制温度变化(如果存储在霜冻的冰柜中,这一点尤其重要)。为防止在-20°C下冻结,RE库存在含有50%甘油的溶液中。由于在存在> 5%甘油的情况下可以抑制或改变RE活性,因此应库存不超过10%的最终RE消化反应混合物。[A 1:10 RE的稀释度将50%的甘油含量为5%。]在不正确的缓冲液条件下或在存在> 5%甘油的情况下,RE的恒星(*)活性可以显示出改变的DNA裂解特异性,称为“恒星活性”。在这种情况下,酶可以识别出其正常6 bp识别位点的4个基对子集,因此将在比预期的更多位点上切割DNA。例如,熟悉的酶EcoRI因其在低离子强度溶液中的恒星活性而臭名昭著。
鉴定人类细胞中复制应力反应的关键因素Louise M.E. Janssen 1,Empar Baltasar Perez 1,Chantal Vaartin
方法在补充了10%FCS,1%谷歌补充剂(Gibco),100 U/ml青霉素和100μg/ml链霉菌素的IMDM(Gibco)中培养了衍生成近单倍型HAP1细胞的细胞培养。siRNA转染是根据制造商的指南使用Rnaimax(Invitrogen)进行的。在这项研究中使用了以下siRNA:Sinon-targetable(Dharmacon),Sipolg2(地平线,TargetPlus,SmartPool),SIMRPL23(Horizon,Targetplus,TargetPlus,Smartpool)。将所有药物(Aphidicolin,Hu,Olaparib,Rad51i(B02),DNA-PKI(NU74441)和寡霉素A)溶解在DMSO中,并以指示浓度使用。细胞使用具有137CS源的γ提取器(最佳疗法)进行γ辐射。生长测定HAP1细胞以1500个细胞/孔的密度将HAP1细胞铺在96孔板中,并被视为5天。5天后,使用100%甲醇固定细胞,并在室温下使用Crystal Violet染色2H。随后,将晶体紫溶解在10%乙酸中,并使用Biotek Epoch Epoch分光光度计在595 nm处测量强度。使用非线性拟合,sigmoidal,4pl,x是log(浓度),将这些测量值用于棱镜中的IC50计算。在9mm玻璃盖上生长免疫荧光细胞,并在室温下以4%甲醛和0.2%Triton X-100固定10分钟。使用了以下抗体:人类抗克雷斯特(Cortex Biochem,CS1058),兔抗PH3SER10(Campro,#07-081),小鼠抗ERCC6L(PICH)(ABNOVA,ABNOVA,000548421-B01P)。所有初级抗体在4°C的夜间孵育。使用固定缓冲液I(BD生物科学)固定细胞。细胞。二级抗体(分子探针,Invitrogen)和DAPI在室温下孵育2小时。使用延长金(Invitrogen)安装盖玻片。使用具有60倍1.40 Na油目标的Deltavision Deonvolution显微镜(Applied Precision)获取图像。SoftWorx(应用精度),ImageJ,Adobe Photoshop和Illustrator CS6用于处理获得的图像。单倍体插入诱变筛选基因对用APH或HU处理的HAP1细胞的存活至关重要,如先前所述35,使用单倍体插入诱变筛查鉴定。诱变的HAP1细胞是从Brummelkamp实验室获得的。简短地,获得HAP1细胞的诱变如下:在HEK293T细胞中产生了基因陷阱逆转录病毒。每天两次收获逆转录病毒至少三天,并通过离心(使用SW28转子进行2小时,21,000 rpm,4°C,4°C)进行沉淀。在8μg/ml硫酸素硫酸素的存在下,在T175烧瓶中至少连续两天,在8μg/ml硫酸素的存在下,将大约4000万个HAP1细胞通过浓缩基因陷阱病毒的转导而被诱变。在包含10%DMSO和10%FCS的IMDM培养基中冷冻诱变细胞。解冻后,在存在27.5 nm adphidicolin或100μmHu的情况下,将诱变的HAP1细胞转移了10天。传递后,通过胰蛋白酶-EDTA收集细胞,然后进行沉淀。为了最大程度地减少潜在地含有杂合突变的二倍体细胞的混杂,用DAPI染色固定的细胞,以允许使用Astrios Moflo对G1单倍体DNA含量进行分类。将3000万个排序的细胞在56°C下裂解过夜,以使使用DNA迷你试剂盒(QIAGEN)进行基因组DNA分离。插入位点映射基因陷阱插入位点通过LAM-PCR放大,然后进行捕获,ssDNA接头连接和指数放大,并在测序之前使用含有Illumina适配器的引物,如前所述,如前所述35。映射和插入位点的分析以前描述了78。简短地,在对HISEQ 2000或HISEQ 2500(Illumina)进行测序之后,将插入位点映射到人类基因组(H19),允许一个不匹配,并与RefSeq坐标相交,以将插入位点分配给基因。基因区域在相对链上重叠的基因区域没有考虑进行分析,而对于在相同链基因名称上重叠的基因是串联的。对于每种复制和两种药物治疗(APH或HU)基因的必要性都是通过二项式检验确定的。合成致死性。一个基因通过所有Fisher的测试,其p值截止为0.05,效应大小至少为0.12(减法比率wt sense比率 - 复制应力条件感官比率)。
ORS 2025 年会论文第 220 号
ssouth@uoregon.edu 披露:Sanique South (N)、Yan Carlos Pacheco (N)、Levi Wood (N)、Nicholas Hannebut (N)、Cindy Brawner (N)、Matlock Jeffries (N)、Nick Willett (N) 简介:全球有数百万人患有创伤后骨关节炎 (PTOA),它是美国导致残疾的主要原因之一。此外,目前尚无已知的治愈方法或疾病改良疗法来阻止 PTOA 进展。细胞疗法在临床前研究中通常显示出巨大的潜力;然而,临床试验显示结果差异很大。这种差异被认为部分来自供体之间细胞效力的高度异质性以及宿主环境的多变性。了解供体人类间充质细胞 (hMSCs) 的可靠性和效力是确保 PTOA 获得一致和优化的治疗结果的关键步骤。 DNA 甲基化和去甲基化在调节 MSC 再生和免疫调节中发挥作用。然而,甲基化在 MSC 调节中的确切作用,以及基线表观遗传模式是否有助于预测关键治疗特性尚不完全清楚。为了弥补这些知识空白,本研究旨在基于基线表观遗传特征和结构结果建立供体 hMSC 治疗效力的预测模型,以研究可修改的细胞靶点,确保细胞治疗获得更好且一致的治疗结果。我们假设,与预测的治疗效果较差的 hMSC 相比,预测的治疗性 hMSC 将表现出独特的表观遗传特征。方法:体外研究:从 RoosterBio 和 Lonza 购买骨髓衍生的 hMSC。将来自 12 位供体的 hMSC 培养 24 小时(RoosterNourish TM -MSC 培养基,RoosterBio;MSCGM™ 间充质干细胞生长培养基,Lonza)。收获细胞并使用 Qiagen DNEasy 试剂盒提取 DNA。DNA 经过亚硫酸盐转化(每个样本 500ng,Zymo EZ DNA 甲基化试剂盒),然后加载到 Illumina Infinium HumanMethylation EPIC 阵列上,该阵列可以量化整个基因组中的 >850,000 个 CpG 位点,包括外显子、内含子和基因间区域。使用 R(v. 4.4.0)进行统计分析。使用 ChAMP 包(v.3.14)加载和处理原始 .IDAT 文件。首先加载原始阵列数据,并将 CpG 位点甲基化数据转换为 beta 值(0-1 甲基化值估计值表示给定 CpG 位点甲基化与未甲基化探针强度之比)。然后使用默认选项的 champ.norm 函数使用 beta 混合分位数归一化程序对 beta 值进行归一化。排除以下情况:(1)检测 P ≥0.01 的探针、针对非 CpG 位点的探针、位于性染色体上的探针,以及在CpG 探针 3' 端 5bp 范围内具有已知单核苷酸多态性的探针,其次要等位基因频率≥1% [1] (N=158,841)。对于模型开发,使用具有自动特征选择的 glmnet 包 (v. 2.0-16) 开发了弹性网络正则化广义逻辑模型。通过 3 倍内部交叉验证调整模型,并记录性能特征。由于发现几个 CpG 位点是再生能力的完美预测因子,我们随后执行了逐步减少数据集的方法,其中,在每一轮开发之后,从数据集中删除最终模型中包含的特征并重新进行开发,总共 50 轮开发周期。所有 50 轮中的所有模型都表现完美(AUC=1.0),可能是因为样本量相对较小而过度拟合。使用在 MATLAB(Mathworks)中生成的 PLSDA 和 PLSR 模型来识别治疗性 hMSC,并使用分泌的细胞因子水平读数作为独立变量,以不同的 hMSC 供体/治疗作为二元结果变量,对来自初始体外研究的 z 分数数据进行训练。使用已建立的内侧半月板横断面 (MMT) 临床前大鼠模型,在 PTOA 的体内临床前模型中验证了预测的治疗性 hMSC(图 1A)。结果:初步研究的数据用于训练 PLSR 预测统计模型。预测模型预测前瞻性地揭示了沿 LV 轴 1 分离的大约六个供体的 hMSC,预测与治疗效果相关,从而预测治疗效果较差和治疗效果较强的供体;因此,6 个样本被指定为可能的“反应者”,6 个被指定为可能的“无反应者”(图 1B)。在甲基化分析中,我们发现在 50 轮开发周期中选定了 119 个 CpG 位点。所有位点均存在显著差异甲基化(P 值 7.5E-8 至 4.1E-4)。与无反应者相比,应答者中大约一半的 CpG 为高甲基化(n=45),其余为低甲基化(n=43)。应答者与无反应者之间平均甲基化值差异最大(Δβ 最高)的 CpG 位点包括 cg14705220(Δβ=0.25 应答者-无反应者 [应答者高甲基化],P =4E-4)和 cg09382002(Δβ=-0.23,P =3E-4 [应答者低甲基化]),图 2。然后,我们对与这些差异甲基化位置相关的基因进行了通路分析。 119 个 CpG 定位到 88 个已知基因。这些基因在 T 细胞信号转导(IL-7 信号转导通路,P =2.27E-3)、吞噬细胞:NK 细胞相互作用(IL-15 产生,P =8.13E-3)和 B 细胞信号转导(April 介导信号转导 P =8.69E-3、B 细胞活化因子信号转导 P =9.09E-3)中的重要通路中富集。有趣的是,差异甲基化基因组位置中富集程度最高的基因网络集中在几个已知的 OA 效应物周围,包括 NFkB 复合物、组蛋白去乙酰化酶 (HDAC) 和机械感受器 (TRPV1) 等 (图 3)。讨论:甲基化数据结果支持了我们的假设,即预测的治疗性 hMSC 将表现出独特的表观遗传特征。我们的数据表明,基于来自 hMSC 的混合细胞 DNA 甲基化数据的模型可以很容易地区分可提供治疗益处的细胞产品和不会提供治疗益处的细胞产品。这些差异甲基化模式中涉及的基因在先前在 OA 中描述的途径中富集。意义/临床意义:DNA 甲基化分析可能有助于在膝关节 OA 关节内注射前筛选 hMSC 供体,以最大限度地提高临床益处。此外,进一步研究我们发现的驱动表观遗传差异的个体细胞亚群可能会揭示出可用于开发未来膝关节 OA 疗法的新途径。致谢:本研究得到了俄勒冈州吴仔人类表现联盟的支持。