XiaoMi-AI文件搜索系统
World File Search System使用自适应克隆选择根据 sMRI 数据对阿尔茨海默病阶段进行分类
摘要 — 结构磁共振成像 (sMRI) 已研究了多种神经系统疾病,并且已将其映射到大脑的不健康区域。必须尽快确定阿尔茨海默病 (AD) 患者,以便开始治疗。最近的研究集中于应用机器学习 (ML) 技术来分割大脑结构并对 AD 进行分类。克隆选择 (CS) 理论有效地实现了分类和优化的目标。自适应克隆选择 (ACS) 技术用于将 sMRI 扫描分为多个类别,例如认知正常 (CN)、轻度认知障碍 (MCI) 和纯 AD 类别。提出的 ACS 描述了免疫反应的基本特征。这为抗原只能在接收它的细胞子集内成熟而不是在身体其他部分成熟的假设提供了支持。与依赖突变的进化计算相比,这种方法擅长关注克隆扩增和亲和力的发展。所提出的 ACS 技术从克隆扩增概念中引入了基本标准,有助于创建用于识别上述 CN、MCI 和 AD 模板匹配的高效策略。所提出的 ACS 方法在分类和检测准确度方面比最先进的方法高出约 99%。关键词 — 阿尔茨海默病 (AD)、磁共振成像 (sMRI)、人工免疫系统 (AIS)、增强模糊 K 最近邻 (EFKNN)、自适应神经模糊推理系统 (ANFIS)
SF9单细胞克隆用于重组蛋白的产生。
1。Hong,M。Et。 al。,杆状病毒insect细胞系统的基因工程,以改善蛋白质的产生。 正面。 Bioeng。 Biotechnol。,2022。 2。 MA,H。等。 al。,Spodoptera frugiperda SF9细胞系是色夫病毒感染和病毒阴性细胞的异质种群:含有色齿病毒X基因变体和病毒阴性细胞Clone的细胞克隆的分离和表征。 病毒学,2019年。 3。 Brogee,P。,朝向C31INIT具有能力的SF9细胞系的发展。 UWSpace,2018年。 4。 Zitzmann,J。等。 al。,单细胞克隆可以选择更有生产力的果蝇Melanogaster S2细胞以进行重组蛋白表达。 生物技术。 REP。,2018。Hong,M。Et。al。,杆状病毒insect细胞系统的基因工程,以改善蛋白质的产生。正面。Bioeng。Biotechnol。,2022。2。MA,H。等。 al。,Spodoptera frugiperda SF9细胞系是色夫病毒感染和病毒阴性细胞的异质种群:含有色齿病毒X基因变体和病毒阴性细胞Clone的细胞克隆的分离和表征。 病毒学,2019年。 3。 Brogee,P。,朝向C31INIT具有能力的SF9细胞系的发展。 UWSpace,2018年。 4。 Zitzmann,J。等。 al。,单细胞克隆可以选择更有生产力的果蝇Melanogaster S2细胞以进行重组蛋白表达。 生物技术。 REP。,2018。MA,H。等。al。,Spodoptera frugiperda SF9细胞系是色夫病毒感染和病毒阴性细胞的异质种群:含有色齿病毒X基因变体和病毒阴性细胞Clone的细胞克隆的分离和表征。病毒学,2019年。3。Brogee,P。,朝向C31INIT具有能力的SF9细胞系的发展。UWSpace,2018年。4。Zitzmann,J。等。al。,单细胞克隆可以选择更有生产力的果蝇Melanogaster S2细胞以进行重组蛋白表达。生物技术。REP。,2018。REP。,2018。
揭开 mAb43——革命性的单克隆抗体药物
mAb43 代表了医学领域的范式转变,提供有针对性的精准治疗,有可能彻底改变多种疾病的治疗格局。凭借其高特异性、良好的药代动力学和多方面的作用机制,mAb43 有望成为一种多功能治疗剂,能够满足尚未满足的医疗需求并改善患者预后。随着正在进行的研究和临床试验继续阐明 mAb43 的全部潜力,医学的未来更加光明,有望为全球患者提供个性化、有效的治疗。
开发了针对单纯疱疹病毒的高效组合单克隆抗体治疗
©作者2024。Open Access本文是根据Creative Commons Attribution 4.0 International许可获得许可的,该许可允许以任何媒介或格式使用,共享,适应,分发和复制,只要您对原始作者和来源提供适当的信誉,请提供与创意共享许可证的链接,并指出是否进行了更改。本文中的图像或其他第三方材料包含在文章的创意共享许可中,除非在信用额度中另有说明。如果本文的创意共享许可中未包含材料,并且您的预期用途不受法定法规的允许或超过允许的用途,则您需要直接从版权所有者那里获得许可。要查看此许可证的副本,请访问http://creativecommons.org/licenses/4.0/。Creative Commons公共领域奉献豁免(http://creativecom- mons.org/publicdomain/zero/zero/1.0/)适用于本文中提供的数据,除非在信用额度中另有说明。
疫苗诱导的对PFRH5的人单克隆抗体显示出对恶性疟原虫临床分离株的广泛中和活性
疫苗对恶性疟原虫网状细胞结合蛋白同源物5(PFRH5)的靶向寄生虫生命周期的血液阶段。pFRH5有可能触发菌株转移抗体的产生,并在临床前和早期临床研究中证明了其功效。疫苗诱导的单克隆抗体(mAb)对PFRH5表现出对来自不同地理区域的恶性疟原虫实验室菌株的有希望的结果。在这里,我们评估了疫苗诱导的抗PFRH5 mAb对遗传多样的恶性疟原虫临床分离株的功能影响。我们使用了先前从英国成年PFRH5疫苗的单个B细胞中分离出来的mAB,并使用了前体内生长抑制活性(GIA)测定法来评估其针对恶性疟原虫临床分离株的功效。下一代测序(NGS)用于评估恶性疟原虫临床分离株中遗传多样性的广度,并推断抗体易感性涉及的基因型/表型关系。我们显示了三个主要GIA组的临床分离株的剂量依赖性抑制:高,中和低。除一个分离株外,我们的数据显示,恶性疟原虫临床分离株和3D7参考菌株之间的mAb GIA谱没有显着差异,该菌株携带了疫苗等位基因。我们还观察到了MAB组合的添加剂关系,因此GIA-LOW和GIA-MEDIUM抗体的组合导致GIA增加,对多克隆IgG反应中特定克隆的贡献具有重要意义。虽然我们的NGS分析显示了PFRH5基因中新型突变的发生,但预计这些突变对已知MAB的抗原结构或识别几乎没有功能影响。我们目前的发现补充了关于抗PFRH5 mAb的菌株超然潜力的早期报道,据我们所知,这是关于恶性疟原虫临床分离株易感性的第一份报告,从自然感染对疫苗诱导的人类MAB对PFRH5的敏感性。
自愿公告重组完全人类抗βklotho单克隆抗体药物(JMT202)获得临床试验批准
该产物是FGFR1C/βKlotho受体激动剂,它通过与βKlotho蛋白结合并特别激活FGFR1C/βKlotho受体复合物,从而调节糖脂代谢,从而模仿其天然叶子,FGFF21蛋白质的作用。该产品可能可用于治疗代谢相关疾病,例如血脂异常,非酒精性脂肪性肝炎(NASH),2型糖尿病和肥胖症。此外,它具有与调节糖脂代谢的其他靶向药物(例如GLP-1)结合使用的潜力。这项临床试验批准的指示是高甘油三酸酯患者的甘油三酸酯水平降低。临床前研究表明,该产品具有良好的安全性,长时间的时间间隔,甘油三酸酯水平的显着降低以及对肝脏的降低症的影响,从而提供了有希望的临床发育价值。
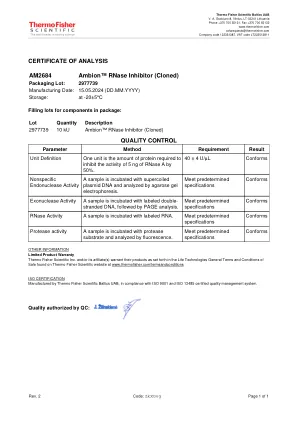
AM2684 Ambion™RNase抑制剂(克隆)
其他信息有限的产品保修Thermo Fisher Scientific Inc.和/或其关联公司保证其产品在Life Technologies的一般销售条款和销售条件中所列出的产品在Thermo Fisher Scientific网站上找到的一般销售条款和条件,网址为www.thermofisher.com/termsandconditions。
先前存在的干细胞异质性决定克隆对癌症驱动突变的采集的反应
2。ICREA,加泰罗尼亚研究与高级研究机构巴塞罗那,西班牙10号加泰罗尼亚。11 12 *这些作者对这项工作也同样贡献13#铅接触:alejo.rodriguez-fraticelli@irbbarcelona.org 14 15摘要:16 17癌细胞,即使患有相同18个突变的患者,癌细胞也显示出广泛的表型变化。原始细胞的差异提供了潜在的解释,但是这些19种测定在传统上依赖于表面标记,缺乏克隆分辨率来区分20个茎和祖细胞的异质子集。为了应对这一21个挑战,我们开发了Strack,这是一个无偏的框架,纵向追踪22个克隆基因表达和膨胀动力学,并在获得23个癌症突变之前和之后。我们研究了两个不同的白血病驱动突变,即DNMT3A-24 R882H和NPM1CA,发现对这两种突变的响应在不同的干细胞状态下均为25个变量。具体而言,通常随时间越来越多的分化26个偏置干细胞可以有效地随两个突变扩展27。npm1c突变令人惊讶地逆转了克隆蛋白的内在偏置28,茎偏置的克隆会引起更加成熟的恶性29个州。我们提出了一个克隆的“反应规范”,其中预先存在的克隆状态30决定了不同的癌症表型潜力。31 32关键字:单细胞,癌症开始,原始细胞,谱系跟踪,DNMT3A,33 NPM1C,克隆造血症,髓样恶性肿瘤34 35 36亮点:37-单细胞在克隆级别的癌症开始(strack)。43 4438-离体扩展文化维持内在和可遗传的HSC异质性。39-预启示性突变增强了高输出干细胞的自我更新,40增加了其生存概率。41-转化突变重编程低输出干细胞命运到更成熟的42个恶性状态。