XiaoMi-AI文件搜索系统
World File Search Systemnlme:线性和非线性混合效应模型
ACF....................................................................................................................................................................................................................................8 ACF.gls....................................................................................................................................................................................................................................9 ACF.lme....................................................................................................................................................................................................................9 ACF.lme.................................................................................................................................................................................................................... . . . . . . . . . . . . . . . . . . . 10 苜蓿 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 11 allCoef . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . .................................................................................................................................................................................................................................................................................................................................................................................................................. 13 anova.lme .................................................................................................................................................................................................................................................................................................................................................................................................................................................. 15 as.matrix.corStruct .................................................................................................................................................................................................................................................................................................................................................................................................................................................. 15 as.matrix.corStruct .................................................................................................................................................................................................................................................................................................................................................................................................................................. . 18 as.matrix.pdMat . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 21 测定. . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 21 表. . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 23 平衡分组. . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 25 bdf. . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ................. ... . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ..................................................................................................................................................................................................................................................32 coef.lmList.......................................................................................................................................................................................................................................................................................33 coef.modelStruct.......................................................................................................................................................................................................................................................................................................................35 coef.pdMat....................................................................................................................................................... ..................................................................................................................................................................................................................................................................................................................................................................................................................36 coef.reStruct ..................................................................................................................................................................................................................................................................................................................................................................................................................................37 coef.varFunc .................................................................................................................................................................................................................................................................................................................................................................................................................................................................................. . ... ....................................................................................................................42 比较预测........................................................................................................................................................................................................................................................................................... . . 43 corAR1 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ... . ... ................. ... . . . . . . 58 corMatrix.corStruct . . . . . . . . . . . . . . . . 59 corMatrix.pdMat . . . . . . . . . . . . . . . . . . . . . 61 corMatrix.reStruct . . . . . . . . . . . . . . . . . ...
印度和美国关于贸易政策论坛的联合声明
双方同意继续从美国进口饲料。美国打算尽快完成将芒果和石榴辐照预先清关计划/监管监督移交给印度当局的工作。美国和印度还期待签署关于印度向美国出口石榴籽的系统方法操作工作计划。印度打算完成植物检疫工作以允许进口美国樱桃,印度打算完成植物检疫认证以允许进口美国苜蓿干草作为动物饲料。此外,美国同意努力完成印度关于鲜食葡萄进入美国的申请,印度同意努力完成双方同意的出口证书以允许进口美国猪肉和猪肉产品。
引用纸浆版本(APA):Griffiths,M.,Delory,B.M.,Jawahir,V.,Wong,K.M.,Bagnall,G.C.,Dowd,T.G.,Nusinow,D.A.,Miller,Miller,A.J
图2在单场试验中生长的覆盖作物物种的表型性状评估。(a)植物表型特征的主要成分分析由植物质量分数和杂草严重程度的家族聚集,这是对PC1和总生物量的最大贡献者,对PC2的贡献最大。(b - g)箱形图显示了每个覆盖作物物种的单个表型特征评分。苜蓿(Medicago sativa),Dundale Pea(trifolium incarnatum),Milkvetch(Astragalus spp。),深红色三叶草(Pisum sativum),毛茸茸的vetch(vicia villosa),芥末酱(Brassica juncea),大麦(大麦(Hordeum vulgare)),小麦(triticum aestivum),冬季rye(secale cereale)(secale cereale)和diliticale(x triticosecale)[×Triticosecale)[
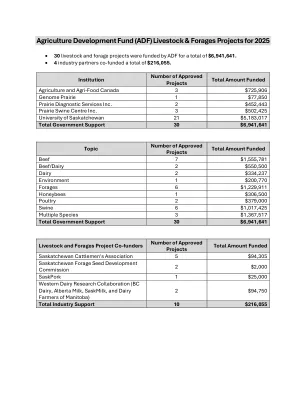
农业发展基金(ADF)牲畜和饲料项目2025
•相对于放牧系统(连续与旋转放牧系统),在放牧期间量化了牛和小牛的生长,以及放牧季节结束时的繁殖率。•测量牛的肠甲烷相对于放牧系统的产生。•表征牛的粪便微生物组,并将其与肠ch4产量相关联并建立肠ch 4的生物标志物。•增强肠甲烷对遗传潜力的基因组预测,以在放牧时选择减少排放的牛。•将牛水平的数据与土壤,植被,温室气体排放和社会经济结果相结合。ADF资金:$ 314,801苜蓿中的气候变化弹性,以增强牛肉和乳制品生产的盈利能力和可持续性。(20240701)首席研究员:Stacy Singer,农业和加拿大农业食品
kuzbass药植物的植物化学组成
burdock(tomentosum磨坊,根),苜蓿(Medicago sativa l.,叶子和茎),普通肺部(肺部官方L.,叶子和茎),常见的Yarrow(achillea millefium l.根),Sweetvetch(Hedysarum neteclect Ledeb。,根)和牛parsnip(Heracleum sibiricum L.,花序,叶子和茎)。要提取类黄酮,我们以40%,55、60、70和75%的浓度使用乙醇。分光光度法用于确定总类黄酮,而高性能液相色谱法被用来研究提取物的定性和定量组成。在sibiricum叶片中发现了类黄酮的最高收益率(除70%以外的所有浓度下),其次是55%和70%乙醇的乙醇提取物,以及75%的乙醇乙醇提取物。因此,这些植物在药物中使用最大的潜力。高性能液相色谱显示
报告名称:农业生物技术年鉴 - 2023
印度仍未决定是否将转基因 (GE) 作物和生物技术 (biotech) 衍生产品用于食品和饲料。Bt 棉花 (苏云金芽孢杆菌) 仍然是唯一获得完全批准用于商业种植的生物技术衍生作物,尽管监管机构现在也已授予生物安全授权,允许在环境中释放转基因茄子和芥菜。来自部分转基因大豆和油菜籽的大豆油和菜籽油,以及一些来自微生物生物技术的食品成分已获准进口。2021 年 8 月,印度商务部允许进口 120 万公吨 (MMT) 的转基因大豆压碎脱油豆饼(即豆粕),作为非 LMO 转基因产品进口。然而,印度在类似产品的市场准入问题上依然拖拖拉拉,例如来自转基因作物(即玉米和大豆)的干酒糟及可溶物和豆粕,以及转基因苜蓿干草。
报告名称:农业生物技术年鉴
自 2018 年 5 月以来,墨西哥尚未正式报告任何用于食品和饲料的转基因 (GE) 农产品的批准。此外,墨西哥以预防原则为由,自 2019 年以来未批准任何转基因作物(棉花和苜蓿)种植许可申请。棉花是墨西哥种植的唯一一种转基因作物,虽然产量在 2018/2019 销售年度创下了历史新高,但由于缺乏更新的种子供应和其他挑战,产量和质量显著降低。2021 年 1 月 1 日,墨西哥总统令生效,根据该令,将撤销对转基因玉米的现有授权,并禁止新的授权,直至 2024 年 1 月 31 日转基因玉米完全淘汰。2021 年,墨西哥进口的转基因玉米和大豆价值分别约为 50 亿美元和 30 亿美元,主要来自美国。
B 部分:将列入公共登记册的释放申请信息
田间试验中要研究的转基因植物是春大麦(Hordeum vulgare)品种 Golden Promise,其中参与感知和定植的基因之一已通过 CRISPR/Cas9 系统丧失功能或通过过度表达进行修改。该研究旨在进行田间试验,以检查 AMF 接种对低磷和富磷土壤中共生途径基因编辑和过表达基因修饰的春大麦品种 Golden Promise 的生物量和产量的影响。大麦品种 Golden Promise 的六个基因,即 SYMRK 、 CCamK 、 Cyclops 、 RAM1 、 NSP1 和 NSP2 已使用 CRISPR-Cas9 介导的基因编辑系统分别进行编辑。在实验室条件下,这些基因的基因编辑导致 AMF 定植失败或显著减少。此外,其中一个基因,即大麦NSP2 (HvNSP2) 及其来自蒺藜苜蓿 (MtNSP2) 的直系同源物,通过过度表达 (OX) 进行修改,从而促进了实验室环境中 AMF 的定植。