XiaoMi-AI文件搜索系统
World File Search System纳米IMPACT电化学检测单DNA折纸
人类肠道中包含大量的微生物,其代谢产物和潜在的有害食品抗原。肠上皮通过表达各种因素将各种因素组装成物理和化学屏障,将免疫细胞位于腔微生物中分离。除了上皮细胞外,免疫细胞对于通过产生炎症和抗炎性介质的生产而对粘膜屏障至关重要。肠道微生物群,由生物微生物的肠生态群落代表,影响宿主免疫系统的成熟和稳态,并有助于维持上皮完整性,并从其代谢中得出的小分子,称为代谢,称为代谢物。反过来,免疫细胞从微生物群中接收信号,并且可能在维持健康的细菌组成和增强上皮屏障功能方面起关键作用,从而导致宿主 - 细菌互助的建立。在包括炎症性肠道疾病在内的各种疾病的患者中,观察到微生物群和代谢组的改变。在这篇综述中,我们将讨论微生物及其代谢物在调节宿主免疫系统中的生理功能,并增强上皮屏障功能。对这些过程的进一步了解将有助于鉴定新的治疗靶标,并随后在一系列慢性炎性疾病中发展治疗干预措施。
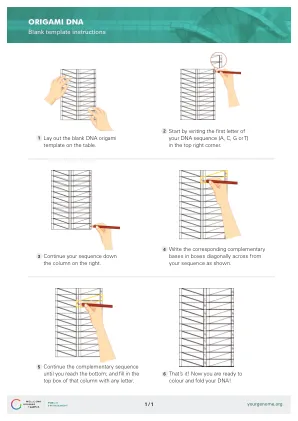
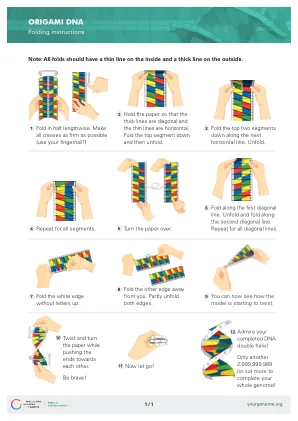
利用 DNA 折纸技术设计配体-药物偶联物用于靶向药物输送
在用于治疗复发性霍奇金淋巴瘤 [5,6] 的 Brentuximab vedotin (Adcetris) 和用于治疗 HER2 + 转移性乳腺癌 [7,8] 的 T-DM1 (Kadcyla) 获得美国食品药品管理局 (FDA) 临床批准的背景下。所谓的“魔弹”最初由 Paul Ehrlich 构想 [9],旨在将小分子药物的毒性与抗体的靶向能力结合起来,以提高总体疗效和治疗指数。[10–15] 尽管概念简单,但 ADC 的开发面临着若干挑战,包括可控的毒性、均质结合和有限的药物有效载荷能力。对于 ADC 来说,药物抗体比 (DAR) 和靶向能力之间的平衡是必需的,以降低候选药物的损耗率。DAR 非常高的 ADC 可能会降低对靶抗原的识别。 [16–19] 因此,开发具有高最大耐受剂量和高选择性的 ADC 是非常有必要的。[20–22]