XiaoMi-AI文件搜索系统
World File Search System更新合格的安全推定列表(QP)
开发了安全性(QP)方法的合格推定,以提供定期更新的对微生物安全性的通用预评估,该预测旨在用于食品或饲料链中,以支持EFSA科学面板的工作。QPS方法是基于对每个代理商的分类学身份,相关知识和安全问题的主体的评估。在可能的情况下,针对分类单元(TU)确定的安全问题是在物种/应变或产品水平上确认的,并由“合格”反映。在此声明所涵盖的时期中,发现新信息导致了芽孢杆菌Velezensis的“缺乏氨基糖苷生产能力”的资格。帕拉利希菌素的质量变为“缺乏杆菌蛋白生产能力”。对于另一个tu,没有发现会改变先前推荐的QPS TU的状态的新信息。在2022年4月至2022年9月之间对EFSA的52种微生物(包含在内),未评估48种微生物,因为:7种是炎性真菌,3个是粪肠球菌,其中2个是大肠杆菌的肠球菌,其中1个是肠splosecces spp。在这一时期内提到的其他四个tus,并且以前被重新分类的一个以前作为一种不同的物种进行了评估。不建议使用QPS状态链球菌。ogataea polymorpha提出了QPS状态,其质量为“仅出于生产目的”。无法评估,因为它没有被识别为物种水平。 QPS状态建议使用QPS状态的地理曲杆菌,并具有“缺乏毒素活性”的质量。lactiplantibacillus argentoratensis(新物种)包括在QPS列表中。
在计算机分析中揭示了推定的Mitovires的新型进化谱系
抽象的Mitoviruses(Mitoviridae家族)是在真菌和植物的线粒体中代表的小无衣壳RNA病毒。迄今为止,唯一的真实的动物米托病毒被鉴定为Lutzomyia longipalpis mitovirus 1(Lulmv1)。来自几种动物的转录组研究的公共数据库可能是识别经常错过的Mitovires的好来源。因此,在NCBI转录组shot弹枪组装(TSA)库中搜索类似于Mitovirus的转录本,以及对先前在NCBI非冗余(NR)蛋白质序列库中记录的Mito-病毒的搜索,以识别与动物相关的类似Mitovirus序列。在TSA数据库中总共确定了10个新的推定中病毒,在NR Pro-te-te-te-Te-Te-Teperin数据库中总共确定了5个推定的Mitovires。据我们所知,这些结果代表了与Poriferan,Cnidarians,echinoderms,Crustaceans,Myriapods和Arachnids相关的推定线病毒的第一个证据。根据使用最大似然法的不同系统发育推论,这18种推定的线索病毒与LULMV1(唯一已知的动物感染线虫病毒)形成了强大的单系谱系。基于计算机程序中的这些发现,证明了与动物相关的一系列推定的mitovirus的有力证据,这些枝条被临时命名为“ kvinmitovirus”。
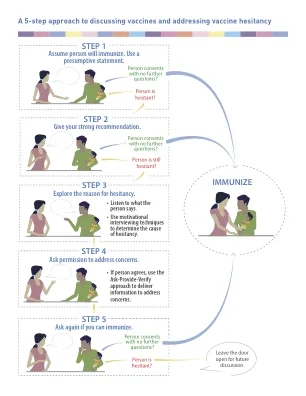
假设人会免疫。使用推定语句。
听取了该人的担忧后,请允许解决他们的担忧。如果该人同意,那么正确提供信息以解决他们的疑虑很重要。做到这一点的一种方法是使用询问验证方法。在这种方法中,医疗保健提供者将询问他们对犹豫的特定原因的了解,提供信息以解决该问题,然后验证信息已被理解。提供信息并验证理解后,总结关键消息可能会有所帮助。