XiaoMi-AI文件搜索系统
World File Search System多重素数编辑和非病毒的传递...
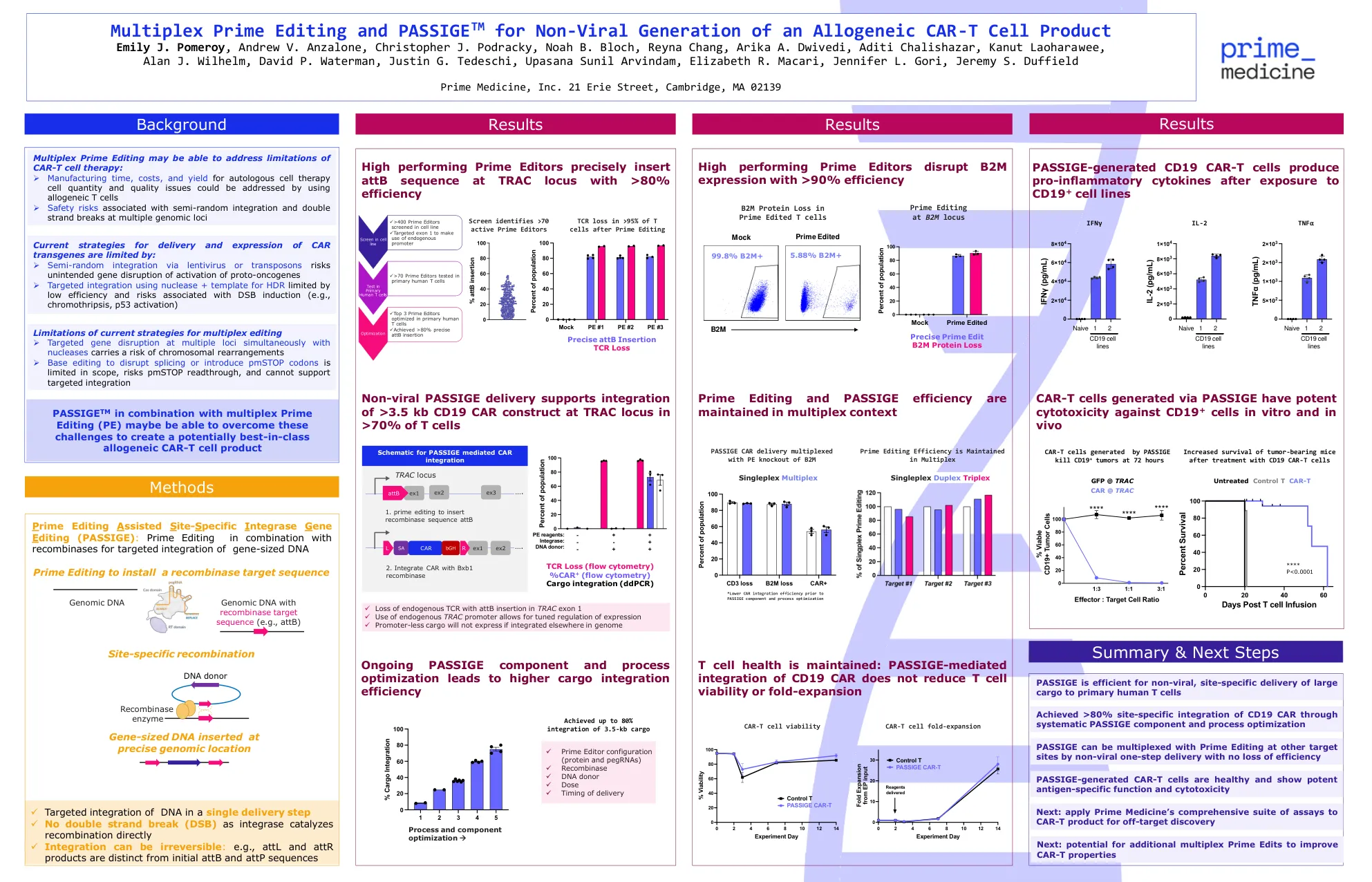
当前的CAR转基因输送和表达策略受到以下限制:➢通过慢病毒或转座子通过慢性病毒或转座的半随机整合危险,即在核酸酶 + to ndrate +限制与DSB诱导相关的HDR限制的核酸酶 +模板的核酸酶积分的靶向整合(例如/chromothips)
DNA 分子量标准简介凝胶电泳操作注意事项
A -1 DNA 降解 —— 避免核酸酶污染。 电泳缓冲液陈旧 —— 电泳缓冲液多次使用后,离子强度降低, pH 值上升,缓冲能力减弱,从而影响电泳效 果。建议经常更换电泳缓冲液。 所用电泳条件不合适 ——电泳时电压不应超过 10 V/cm ,温度小 于 30 ℃,核查所用电泳缓冲液是否有足够的 缓冲能力和凝胶浓度是否正确。 DNA 上样量过多 ——减少凝胶中 DNA 上样量,建议电泳样 品根据孔的宽度加样。 DNA 样含盐过高 ——电泳前通过乙醇沉淀去除过多的盐。 有蛋白污染 ——电泳前酚抽提去除蛋白。 琼脂糖质量 ——选用高质量的琼脂糖 (TIANGEN 公司 ) 。
CRISPR技术在植物基因组编辑中的应用
新型基因编辑技术中使用的核酸酶主要有四类,分别是:巨核酸酶、锌指核酸酶(ZFN);转录激活因子样效应核酸酶 (TALEN);以及成簇的规律间隔的短回文重复序列 (CRISPR) 相关 (Cas) (Gaj 等人,2016)。巨核酸酶是一种在特定区域切割 DNA 的内切核酸酶,可识别大于 12 bp(碱基对)的序列。 LAGLIDADG 巨核酸酶家族包含 I-CreI 和 I-SceI,它们是第一种用于基因编辑的酶。由于只有少数氨基酸残基与核苷酸接触,这些酶被设计用于在特定位点切割基因(Paques;Duchateau,2007)。此外,ZFN 是一种人工酶,也是最早用于诱导植物靶向突变的酶之一。这些酶是由锌指型结构域和限制性酶 Fok I 的结构域融合产生的。与基因编辑中使用的其他核酸酶一样,ZFN 会在需要修复的 DNA 特定位置插入双链断裂 (DSB),并且由于修复机制中的故障,可能会出现突变 (Carroll, 2011)。使用该系统的主要问题是这种酶的高毒性,以及它会产生许多脱靶效应(Cornu et al., 2008; Ramirez et al., 2008),这会损害不应改变功能的基因的功能(Zhang et al., 2015)。随着版本的合并
生命编辑基因编辑——随时随地进行编辑
双链断裂:双链断裂涉及核酸酶切割 DNA 的两条链,从而实现在切割位点的基因删除(敲除)或插入/修复(敲入)。我们的核酸酶的 PAM 多样性允许在基因组的几乎任何地方引入敲除或敲入编辑。碱基编辑:碱基编辑将一个核苷酸(碱基)转换为另一个核苷酸,而无需切割 DNA 的两条链。这是通过将经过修改以仅切割一条 DNA 链的核酸酶与编辑目标核苷酸的脱氨酶偶联来实现的。我们的模块化碱基编辑方法将我们的专有核酸酶和脱氨酶彼此偶联。
CRISPR 基因组编辑的最佳解决方案
CRISPR-Cas9技术是强大的第三代基因组编辑工具,比第一代ZFN(锌指核酸酶)和第二代TALEN(转录激活因子样效应核酸酶)更经济、更高效。 CRISPR-Cas9系统由精确识别并结合目标DNA的向导RNA和切割目标DNA的Cas9核酸酶组成,通过破坏和修复DNA双链的过程实现基因操作。 Bioneer 通过与基因组编辑技术领导者 ToolGen 合作推出的 AccuTool™ 提供了基因组编辑的整体解决方案,从 gRNA 设计到合成、Cas9 核酸酶和验证。
基因组编辑技术,机制和改善治疗性植物化学物质的产生:机会和前景
图1各种基因组编辑工具。(a)锌指核酸酶(ZFN)充当二聚体。每个单体由DNA结合结构域和核酸酶结构域组成。每个DNA结合结构域由3 - 6个锌指重复序列组成,识别9 - 18个核苷酸。核酸酶结构域由II型限制性核酸内切酶FOK1组成。(b)转录激活剂类似核酸酶(Talens):这些是类似于ZFN的二聚体酶。每个亚基由DNA结合结构域(高度保守的33 - 34个氨基酸序列)和FOK1核酸酶结构域组成。(c)CRISPR/CAS9:CAS9核酸内切酶由SGRNA(单引导RNA:CRRNA和TRACRRNA)引导,用于靶特定裂解。二十个核苷酸识别位点存在于原始基序(PAM)的上游(来自Arora&Narula,2017年)。版权所有©2017 Arora和Narula。这是根据Creative Commons归因许可(CC BY)的条款分发的开放访问文章。
第4章通过技术中的植物中的基因表达调制CRISPR/DCAS9
在近几十年内,涉及DNA精确操纵的核酸酶的技术已经发生了深刻的进步,成为了诱导音节突变的有希望的替代方法,并且对基因表达的薄而控制。是基因组编辑,例如核酸酶锌指(锌指核酸酶),具有转录本激活型效应的数字(Talens,英语转录本类核酸酶),以及最近的CRISPR/CAS技术(来自英语粘膜调节性调节性的短与核酶壳相关)。后者具有其革命性,尤其是为了缘故,普遍性和相对简单性(Pickar-Oliver; Gersbach,2019年)。此外,CRISPR/CAS是一种灵活的工具,需要进行修改,这有助于其持续的改进并多样化其在细胞功能和生物技术中的应用。
基因组医学:了解基因组工程,重点关注基因组编辑
基因组编辑的一种主要方法是将称为“工程核酸酶”的酶剪刀部署到 DNA 的目标区域。一旦工程核酸酶被引导到精确的位置,它就会结合并切割目标基因。此时,核酸酶通过修改、移除或替换有缺陷的基因对细胞的遗传密码进行永久性更改。根据细胞的类型,基因组编辑可能会在患者体内暂时或永久保留。
一个基因组还是数千个基因组?群体基因组学和脱靶风险
使用 SpCas9 核酸酶进行 ONE-seq 脱靶分析的结果 a,群图显示五个先前分析的 SpCas9 gRNA 的 ONE-seq 核酸酶分数。每个圆圈代表一个单独的 ONE-seq 文库成员。彩色圆圈代表先前确认的真正脱靶位点。未显示 ONE-seq 核酸酶分数低于 0.001 的位点。n/a,未在先前发表的 CIRCLE-seq 研究中进行验证。b,维恩图比较了 ONE-seq、CIRCLE-seq 和 Digenome-seq(空心彩色圆圈)提名先前由 GUIDE-seq(实心紫色圆圈)验证的真正脱靶位点的能力。所有被视为由 ONE-seq 验证的位点的 ONE-seq 核酸酶分数均 >0.01。
新一代基因编辑工具研究进展
摘要 以核酸酶为主要成分的基因编辑工具目前已能对哺乳动物基因组实现可编程的定点突变或插入或删除。从锌指核酸酶(ZFNs)、转录激活因子样效应核酸酶(TALENs)、CRISPR/Cas系统到更安全、更精准的Cas9融合蛋白基因编辑工具以及其他核酸酶基因编辑工具,本文系统地阐述了基因编辑的发展与演变,详细介绍了下一代基因编辑工具的开发与优化,并对基因编辑工具的临床应用与挑战进行了展望。 关键词 基因编辑;CRISPR/Cas9;碱基编辑;先导编辑;双链断裂;进展