XiaoMi-AI文件搜索系统
World File Search System使用工程化的 CRISPR RNA 引导胞苷脱氨酶对结核分枝杆菌进行可编程碱基编辑
耐多药结核分枝杆菌 ( Mtb ) 感染严重危害全球人类健康,迫切需要新的治疗策略。高效的基因组编辑工具有助于识别参与细菌生理、发病机制和耐药机制的关键基因和途径,从而有助于开发耐药结核病的新疗法。在这里,我们报告了一个双质粒系统 MtbCBE,用于灭活基因并在 Mtb 中引入点突变。在该系统中,辅助质粒 pRecX-NucS E107A 表达 RecX 和 NucS E107A 以抑制 RecA 依赖性和 NucS 依赖性的 DNA 修复系统,碱基编辑质粒 pCBE 表达结合胞苷脱氨酶 APOBEC1、Cas9 切口酶 (nCas9) 和尿嘧啶 DNA 糖基化酶抑制剂 (UGI) 的融合蛋白。这两个质粒共同实现了结核分枝杆菌基因组中所需位点处 G:C 到 A:T 碱基对的有效转换。碱基编辑系统的成功开发将有助于阐明结核分枝杆菌致病机理和耐药性的分子机制,并为开发其他微生物的碱基编辑工具提供重要启发。
碱基编辑酶 APOBEC3A 以低效率和高选择性催化 RNA 中的胞嘧啶脱氨
A3A 和 eA3A 表达。A3A 表达构建体 (Addgene #109231) 之前已有描述,可用于纯化 A3A 作为融合蛋白 (MBP-A3A-His),可进一步加工以生成分离的 A3A 结构域。32,33 对于 eA3A (A3A-N57G),N57G 突变是通过 Q5 定点诱变 (New England Biolabs, NEB) 引入的。A3A 和 eA3A 构建体的细菌表达之前已有详细描述。 33 将纯化的 MBP-A3A-His、MBP-eA3A-His 或分离的 A3A 在 50 mM Tris-Cl(pH 7.5)、50 mM NaCl、10% 甘油、0.5 mM DTT 和 0.01% Tween-20 中透析过夜,并使用 BSA 标准曲线确定蛋白质浓度。基于 SwaI 的脱氨酶对 ssDNA 和嵌合底物的活性。5'-荧光素 (FAM) 荧光标记的底物 S35-dC 或具有单个靶核糖胞嘧啶的匹配底物(在其他 DNA 骨架中)(S35-rC)由 Integrated DNA Technologies (IDT) 合成,以及相关产品对照(S35-dU 和 S35-rU)。在最佳 A3A 反应条件(最终为 20 mM 琥珀酸:NaH 2 PO 4:甘氨酸 (SPG) 缓冲液 pH 5.5,0.1% Tween-20)下,用 6 倍稀释的未标记 A3A(从 1 µM 到 4 pM)处理 100 µM 寡核苷酸。反应在 37 ˚C 下进行 30 分钟,然后终止(95 ˚C,10 分钟)。然后加入 200 nM 互补链并退火。加入 SwaI (NEB),在室温下消化过夜。加入甲酰胺上样缓冲液,样品加热变性(95 ˚C,20 分钟),然后在 50 ˚C 下在 20% 变性 TBE/尿素聚丙烯酰胺凝胶上运行。使用 Typhoon 成像仪(GE Healthcare)上的 FAM 滤光片对凝胶进行成像。使用 ImageJ 中的面积量化工具进行定量分析。720 碱基对 ssDNA 底物的合成。为了生成 ssDNA,使用 720 bp gBlock 基因片段 (IDT) 作为模板 (补充图 2a),并使用 Taq 聚合酶 (NEB) 进行扩增,采用指数后线性 (LATE) PCR 反应方案,该方案采用相对于磷酸化的反向引物过量的正向引物。32 对反应物进行纯化 (NucleoSpin、Fisher),然后在 37 ˚C 下用 核酸外切酶处理 1 小时以降解磷酸化链,然后进行热失活 (90 ˚C,10 分钟)。然后将产物在 2% 琼脂糖凝胶上运行,并使用凝胶 DNA 回收试剂盒 (Zymoclean) 回收 ssDNA。通过乙醇沉淀进一步纯化 ssDNA,并使用 Qubit ® 荧光计 (ThermoFisher) 测量其浓度。对于一个重复,ssDNA 以大分子寡核苷酸 (IDT) 的形式获得,并通过乙醇沉淀进一步纯化。720 聚体 RNA 底物的合成。使用 720 bp 基因块 (IDT) dsDNA 作为模板,在推荐条件下使用 TranscriptAid Enzyme Mix (ThermoFisher) 通过体外转录生成 RNA,并在 37 ˚C 下孵育两小时。然后通过苯酚-氯仿提取和乙醇沉淀纯化 RNA。将样品重新悬浮在无核酸酶的水中,并进一步用 MspI、XbaI 和 AclI 限制性酶 (NEB) 处理以消化任何剩余的 DNA 模板。在 37 ˚C 下孵育 1 小时后,使用 RNA Clean and Concentrator-5 试剂盒 (Zymo Research) 纯化 RNA。为了进一步确保完全去除模板 DNA,在 37 ˚C 下用 DNase I (Ambion) 处理 RNA 30 分钟。重复纯化 (RNA Clean and Concentrator-5),并使用 Qubit ® 荧光计测量纯化 RNA 的浓度。通过“预测二级结构网络”预测 720 聚体中几个中尺度区域的二级结构
SARS-CoV-2 基因组中的短序列基序动态表明胞嘧啶脱氨在 CpG 减少中起着作用
。CC-BY-NC-ND 4.0 国际许可下可用(未经同行评审认证)是作者/资助者,他已授予 bioRxiv 永久展示预印本的许可。它是此预印本的版权持有者此版本于 2021 年 1 月 5 日发布。;https://doi.org/10.1101/2020.06.19.161687 doi:bioRxiv 预印本
用脱氨酶替换 SpCas9 HNH 结构域...
预印本(未经同行评审认证)是作者/资助者。保留所有权利。未经许可不得重复使用。此版本的版权所有者于 2020 年 11 月 9 日发布。;https://doi.org/10.1101/2020.11.09.371237 doi:bioRxiv 预印本
编辑的作用是什么?了解腺苷脱氨酶作用于 RNA 的 A-to-I RNA 编辑的生理功能
腺苷到肌苷 (A-to-I) 编辑是一种 RNA 转录后修饰,可改变其序列、编码潜力和二级结构。在作用于 RNA 的腺苷脱氨酶 (ADAR) 蛋白 ADAR1 和 ADAR2 的催化下,A-to-I 编辑发生在小鼠的约 50 000 – 150 000 个位点上,而人类的位点则达到数百万个。绝大多数 A-to-I 编辑发生在重复元素中,这解释了物种间位点总数的差异。ADAR1 在哺乳动物中编辑的主要物种保守作用是抑制未编辑的细胞来源的内源性 RNA 引起的先天免疫激活。在没有编辑的情况下,反向配对序列(例如 Alu 元素)被认为会形成稳定的双链 RNA (dsRNA) 结构,从而触发 dsRNA 传感器(例如 MDA5)的激活。一小部分编辑位点位于编码序列内,并且在后生动物中进化保守。已证明 ADAR2 编辑对于大脑神经递质受体的重新编码具有生理重要性。此外,RNA 编辑的变化与各种病理状态有关,从严重的自身免疫性疾病 Aicardi-Goutières 综合征到各种神经发育和精神疾病以及癌症。但是,检测到编辑位点是否意味着功能重要性?人类和转基因小鼠模型中的遗传学研究以及进化基因组学已开始阐明 A-to-I 编辑在体内的作用。此外,最近的发展表明在癌症等病理条件下编辑可能具有不同的功能。
使用 Cas9 - 腺嘌呤脱氨酶融合在细菌中进行可编程腺嘌呤脱氨†
系统已被探索作为有效的选择剂来消除未编辑的细胞,从而大大简化了细菌中的基因操作过程。9尽管基于 CRISPR/Cas 的基因组编辑方法简单且高效,但它们仍然依赖于细菌中的 HR 来实现精确的基因操作,因此难以在某些缺乏强大 HR 系统的细菌(如结核分枝杆菌)中建立。最近,脱氨酶介导的碱基编辑系统的发展为生物学中的精确基因操作提供了新策略。10 – 12碱基编辑系统使用脱氨反应和随后的 DNA 复制过程直接转换目标碱基,而不是前面提到的基于 CRISPR/Cas 的基因组编辑方法中所利用的 HR。已经建立了两种主要类型的碱基编辑系统:胞嘧啶碱基编辑器(CBE)10,11 和腺嘌呤碱基编辑器(ABE)。 12,13 CBE 已广泛用于各种生物体(包括真核生物 10,11,14 - 17 和一些细菌物种 17 - 22)中的可编程胞嘧啶到胸腺嘧啶的转化,而 ABE 主要在真核生物中建立,例如哺乳动物细胞 12,23 和植物 24,25,用于精确的腺嘌呤到鸟嘌呤的转化。最近,在链霉菌中开发了一种名为 CRISPR-aBEST 的 ABE 系统。13 此外,还开发了可编程的腺苷到肌苷和胞苷到尿苷的 RNA 编辑器。26,27
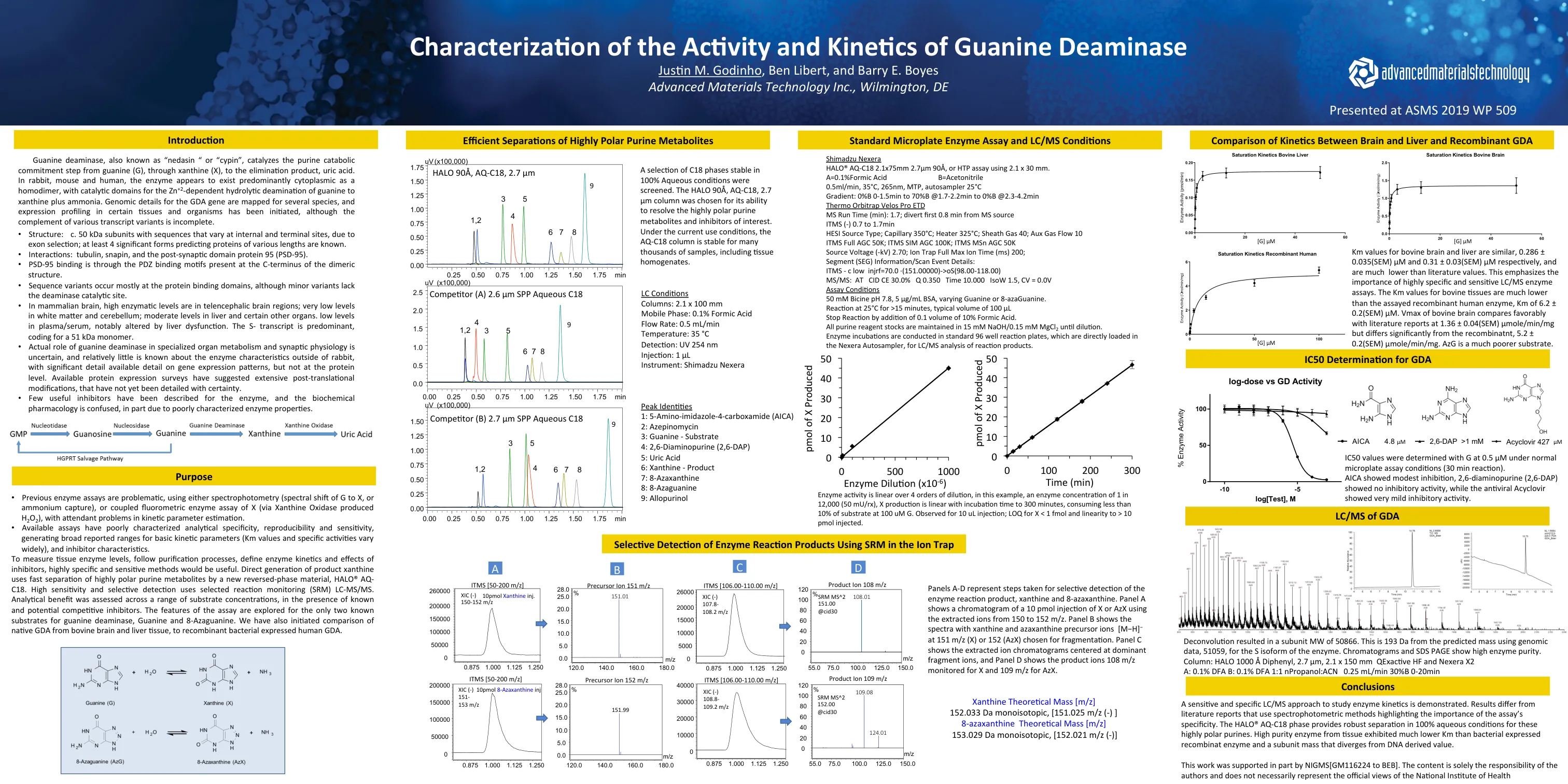
鸟嘌呤脱氨酶动力学的表征
属 要测量 直接属 高sensi 分析 对于仅有两个已知的鸟嘌呤脱氨酶,鸟嘌呤和8-亚瓜氨酸的已知底物探索了测定的特征。 我们还对来自牛脑和肝脏的Na要测量 直接属 高sensi 分析 对于仅有两个已知的鸟嘌呤脱氨酶,鸟嘌呤和8-亚瓜氨酸的已知底物探索了测定的特征。 我们还对来自牛脑和肝脏的Na直接属 高sensi 分析 对于仅有两个已知的鸟嘌呤脱氨酶,鸟嘌呤和8-亚瓜氨酸的已知底物探索了测定的特征。 我们还对来自牛脑和肝脏的Na高sensi 分析 对于仅有两个已知的鸟嘌呤脱氨酶,鸟嘌呤和8-亚瓜氨酸的已知底物探索了测定的特征。 我们还对来自牛脑和肝脏的Na分析 对于仅有两个已知的鸟嘌呤脱氨酶,鸟嘌呤和8-亚瓜氨酸的已知底物探索了测定的特征。 我们还对来自牛脑和肝脏的Na对于仅有两个已知的鸟嘌呤脱氨酶,鸟嘌呤和8-亚瓜氨酸的已知底物探索了测定的特征。我们还对来自牛脑和肝脏的Na