XiaoMi-AI文件搜索系统
World File Search SystemEdit-R™ CRISPR 设计工具
图 1. Edit-R™ CRISPR 设计工具如何选择针对人类基因 PPIB 的 20 个核苷酸序列的示例。目标序列可以位于基因组 DNA 的任一链上,只要它处于 5' 到 3' 方向,并且该链的 3' 端有一个 NGG PAM(使用默认的 S. pyogenes Cas9 时)。Cas9 核酸酶将在 NGG PAM 上游三个核苷酸的位置切割 DNA 的两条链。建议选择完全位于编码基因早期组成外显子内的靶位点,但 Edit-R™ CRISPR 设计工具将返回整个编码区域的结果,因此如果需要,可以靶向特定的外显子或蛋白质结构域。
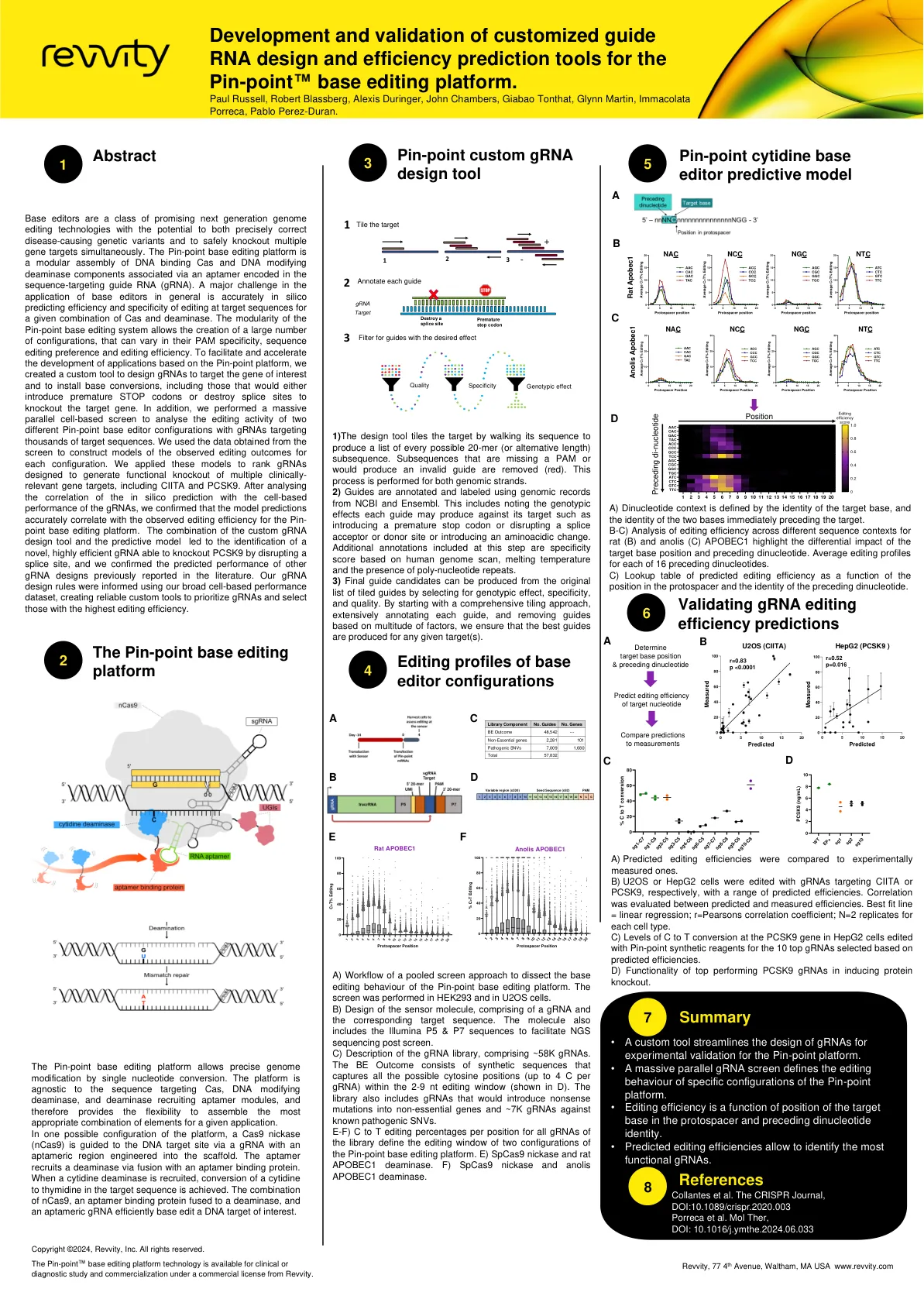
精准定制 gRNA 设计工具
碱基编辑器是一类有前途的下一代基因组编辑技术,既可以精确纠正致病的遗传变异,又可以同时安全地敲除多个基因靶标。Pin-point 碱基编辑平台是一个模块化组装的 DNA 结合 Cas 和 DNA 修饰脱氨酶组件,它们通过序列靶向向导 RNA (gRNA) 中编码的适体连接。碱基编辑器应用中的一个主要挑战是准确地通过计算机预测给定 Cas 和脱氨酶组合在目标序列上的编辑效率和特异性。Pin-point 碱基编辑系统的模块化允许创建大量配置,这些配置的 PAM 特异性、序列编辑偏好和编辑效率可能有所不同。为了促进和加速基于 Pin-point 平台的应用程序开发,我们创建了一个自定义工具来设计 gRNA 以靶向感兴趣的基因并安装碱基转换,包括那些会引入过早的终止密码子或破坏剪接位点以敲除目标基因的转换。此外,我们进行了大规模并行细胞筛选,以分析两种不同的 Pin-point 碱基编辑器配置的编辑活动,其中 gRNA 靶向数千个目标序列。我们使用从筛选中获得的数据构建了每种配置观察到的编辑结果的模型。我们应用这些模型对设计用于产生多个临床相关基因靶标(包括 CIITA 和 PCSK9)功能性敲除的 gRNA 进行排序。在分析了计算机预测与 gRNA 的细胞性能之间的相关性后,我们确认模型预测与 Pin-point 碱基编辑平台观察到的编辑效率准确相关。自定义 gRNA 设计工具和预测模型的结合导致识别出一种新型、高效的 gRNA,它能够通过破坏剪接位点来敲除 PCSK9,并且我们确认了文献中先前报道的其他 gRNA 设计的预测性能。我们的 gRNA 设计规则是使用我们广泛的基于细胞的性能数据集得出的,从而创建了可靠的自定义工具来优先考虑 gRNA 并选择具有最高编辑效率的 gRNA。
未来的人机设计工具:从各个层面...
设计师需要什么?设计,作为一个动词,是关于构思和创造的。任何系统的设计者都需要表示系统的组件以及它们如何组合在一起。对于社会技术系统,这些组件既指机器,又指人。在设计执行某种功能的东西时,设计师还需要表示该功能所需的过程或活动流程。在创造新事物时,很难甚至不可能预测设计选择的所有后果,但这种困难不应阻止设计师寻求帮助他们进行此类预测的工具。对于分布式、多方系统(所有社会技术系统都是如此),预测性能的关键能力是识别和理解工作过程中的因果关系。因此,人机设计工具应根据它们如何很好地表示人和机器、正在执行的工作以及整个工作过程中人与机器之间的关系来评估。
针点定制GRNA设计工具
荧光素酶测定允许研究转录基因表达,病毒生命周期,细胞活力和生化过程,使其成为药物开发的重要工具。您是否正在寻找记者基因,ATP检测,我们的荧光素酶发光测定选项以便利的微板格式提供了高灵敏度。
为AI生物设计工具开发护栏
人工智能(AI)与生命科学的融合有可能为社会带来巨大的好处,但是AI生物设计工具(BDTS)的进步也带来了可能造成重大伤害的风险,并带有潜在的全球后果。几乎没有护栏,以确保安全安全地使用BDT。本报告基于与各种生物安全专家,AI专家和BDT开发人员的访谈和讨论,确定了可能的内置护栏以及管理BDT访问访问BDT以促进访问权限的选择,同时又可以防止滥用滥用。在本报告中,“护栏”一词是指与模型本身相关的减轻风险措施,从模型的概念和开发到其部署或释放。该报告还确定了潜在的试点项目,以启动这些护栏的开发,探索可行性和挑战,并扩大工具包以保护BDT。
核酸线框纳米结构的设计工具
在核酸纳米技术中,纳米级结构是由DNA或RNA的合理设计的链自组装的(1,2)。核酸的碱基配对特性使它们成为可编程的可编程材料,它可以使结构具有高精度和复杂性的组装,其中包括目前多达数万个核苷酸。DNA和RNA折纸(3,4)是两个强大的,广泛的设计范式,可以指导如何通过精心构成的辅助链或kisterifs sistaple staple strands-spaple-staple-staple-staple-staple-staple-staple-staple-staple-staple-staple-staple-staple-staple-staple-staple-staple-staple-staple-staple strander-s in dna s in dna s in of dna procrant-s s of dna staple strands s in dna s in''' RNA折纸中的主题)。两种方法都已用于设计各种2D形状和3D结构(5,6)。大多数当前的3D折纸设计遵循在彼此顶部包装几层螺旋螺旋或螺旋束的方法,和 /或弯曲的螺旋束如最初建议的< / div>
Alt-R HDR 设计工具和 HDR 供体寡核苷酸
图 1.与其他修饰相比,具有 Alt-R HDR 修饰的供体寡核苷酸表现出更高的 HDR 效率。 图 1.与其他修饰相比,具有 Alt-R HDR 修饰的供体寡核苷酸表现出更高的 HDR 效率。 (A)供体寡核苷酸修改的示意图。 (B)每次修改的 HDR 效率。使用 4D-Nucleofector™ 系统(Lonza)将四个靶向基因位点的 RNP 复合物(2 µM)与 0.5 µM 单链 HDR 供体寡核苷酸通过电穿孔共转染到 Jurkat 和 HeLa 细胞中。使用的 RNP 复合物是 Alt-R Sp HiFi Cas9 Nuclease V3,以及 Alt-R CRISPR-Cas9 crRNA 和 tracrRNA。 使用了三种类型的供体寡核苷酸:未经任何修饰的寡核苷酸(未修饰的)、具有硫代磷酸酯键的寡核苷酸(PS 修饰的)和具有 Alt-R HDR 修饰的寡核苷酸(Alt-R HDR 修饰的)。 电穿孔后 48 小时 (HeLa) 或 72 小时 (Jurkat) 提取基因组 DNA。通过在 Illumina™ MiSeq™ 系统 (v2 化学、150 bp 双端读取) 上进行扩增子测序来测量 HDR 效率。
dnaforge:核酸线框纳米结构的设计工具
strands”在DNA折纸中,接吻环和RNA折纸中的其他连接器图案)。两种方法都已用于设计各种2D形状和3D结构(5,6)。大多数当前的3D折纸设计遵循在彼此顶部包装几层二维螺旋或螺旋束的方法,和/或弯曲的螺旋束如(7,8)中最初建议。3D设计的替代路径是创建一个线框结构,该结构仅包含3D模型的边界边缘和顶点。在这个方向上有几个值得注意的前虫前旅行(9,10),但是随着柔性且坚固的折纸技术的发展,它大多开始获得追随者(6,11)。与螺旋装箱相比,线框设计的一些优势包括使用链的经济,这允许建造较大的结构,并在低盐条件下更好地折叠。一些挑战是结构的刚性较低,尤其是对于大型的单螺旋边缘设计(可以通过使用多螺旋边缘来减轻,以增加链的使用来缓解)和大型复杂设计的产量低。已经存在几种核酸纳米结构设计工具(8、12、13、14、15、16、17、18、19、20、21)。Most of these however address helix-packing designs, with the more recent ones oriented towards wireframe structures including vHelix (14), DAEDALUS (15) and ATHENA (18) for 3D DNA wireframes, Sterna (20) for single-stranded 3D RNA wireframes and PyDAEDALUS (21) for 3D RNA/DNA hybrid wireframes.这些工具主要支持一种特定的设计方法,每个工具也都处于离线状态,需要一个单独的过程来安装工具及其辅助库,有时可能很难找到或在最坏的情况下弃用。
PTPT:通过多物理设计工具参数调整...
摘要 — 在先进的集成电路设计中,与电子设计自动化 (EDA) 工具相关的物理设计流程起着至关重要的作用。大多数情况下,输入物理设计工具的参数主要是基于专家的领域知识手动挑选的。然而,由于技术节点的不断缩小以及参数组合所跨越的设计空间的复杂性,甚至再加上耗时的仿真过程,这种对物理设计工具参数配置的手动探索变得极其费力。在设计流程参数调整领域存在一些工作。然而,非常有限的现有技术探索了多个感兴趣的结果质量 (QoR) 指标(例如延迟、功率和面积)之间的复杂相关性并同时明确优化这些目标。为了克服这些弱点并寻求物理设计工具的有效参数设置,在本文中,我们提出了一个多目标贝叶斯优化 (BO) 框架,以多任务高斯模型作为替代模型。采用基于信息增益的获取函数依次选择工具模拟候选对象,以有效逼近帕累托最优参数配置。在 7 纳米技术节点下的三个工业基准上的实验结果证明了所提出的框架相对于前沿作品的优越性。
用于快速评估航空航天部件结构的 AM 设计工具
航空航天界最近对可消耗/消耗性技术的关注凸显了对新的、简化的设计工具的需求,以及对当前设计要求的潜在重新评估。增材制造 (AM) 技术提供了设计灵活性和创新潜力;然而,对于 AM 材料,内部空隙和表面特征会导致组件故障,并在应力寿命 (S-N) 数据中产生显著的分散。AM 分散减少通常通过工艺优化来实现;然而,这种方法需要特定的 AM 工艺知识和大量的材料数据。缺陷知情设计方法可确定特定的检查要求,最大限度地减少表征材料性能的数据量,并减少大量工艺开发以最大限度地减少缺陷的需要,因为缺陷的影响已明确包含在设计中。因此,缺陷知情设计可有效降低 AM 工艺开发的成本。当前的工作利用小裂纹扩展模型和实验缺陷观察来开发缺陷信息 S-N 等效模型,该模型可以根据起始缺陷应力强度因子 (K) 准确预测组件寿命。这种 K-N 方法减少了在 S-N 数据中观察到的散度,并最大限度地减少了模型所需的训练数据。这项研究的结果是用于 AM 应用的多功能设计工具,以及对不确定性量化的材料测试要求的定量理解。在这里发现的见解揭示了改进可消耗/消耗品领域设计的机会,并促进了 AM 供应链中的成本节约,从而使各行各业受益。