XiaoMi-AI文件搜索系统
World File Search System2023-2024-2英语教授的本科课程...
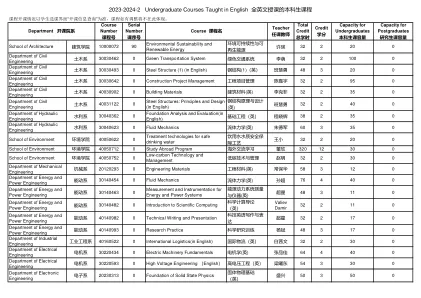
Department of Physics 物理系 20430225 2 Fundamentals of Physics (1) 基础物理学 (1) 蒋硕 96 5 150 0 Department of Physics 物理系 20430234 1 Fundamentals of Physics (2) 基础物理学 (2) 蒋硕 64 4 102 0 Department of Physics 物理系 20430265 3 Fundamentals of Physics (3) 基础物理学 (3) Kim Kihwan 80 5 130 0 School of Life Sciences 生命学院 30450203 1 Biochemistry(1)(in English) 生物化学 (1)( 英文 ) 刘栋 48 3 175 0 School of Life Sciences 生命学院 30450203 2 Biochemistry(1)(in English) 生物化学 (1)( 英文 ) 刘栋 48 3 180 0 School of Life Sciences 生命学院 30450263 0 Microbiology(in English) 微生物学 ( 英文 ) 陈国强 48 3 80 0 School of Life Sciences 生命学院 30450303 1 Genetics(in English) 遗传学(英文) 王田 48 3 90 0 School of Life Sciences 生命学院 30450303 2 Genetics(in English) 遗传学(英文) 王田 48 3 100 0 School of Life Sciences 生命学院 30450453 0 Molecular Biology(in English) 分子生物学 ( 英 ) 杨扬 48 3 150 0
降量药物的全脑激活特征
目的:有效的抗肥胖治疗剂的发展在很大程度上取决于靶向控制体重的特定脑稳态和享乐机制的能力。为了进一步了解抗肥胖药物治疗招募的神经循环,本研究旨在确定六种六种不同体重的药物类别的全脑激活特征。方法:食用C57BL/6J小鼠(每组n¼8)接受Lorcaserin(7 mg/kg; I.P.),rimonabant(10 mg/kg; i.p.),溴张素(10 mg/kg; i.p.),sibutramine(10 mg/kg; p.o.),semaglutide(0.04 mg/kg; s.c。)或setMelanotide(4 mg/kg; s.c。)。给药后两个小时对大脑进行采样,并使用C-FOS免疫his型化学和自动定量三维(3D)成像在单细胞分辨率下分析全脑神经元激活模式。结果:整个脑分析包括308个地图集的小鼠脑区域。为了启用快速和有效的数据挖掘,开发了基于Web的3D成像数据查看器。所有降级药物均表现出脑部范围的反应,在C-FOS表达特征中具有显着相似之处。在位于背面迷走神经复合物和下丘脑中的离散稳态和非室内喂养中心中检测到重叠的C-FOS反应,并同时激活多个边缘结构以及多巴胺能系统。2021作者。由Elsevier GmbH出版。这是CC BY-NC-ND许可证(http://creativecommons.org/licenses/by-nc-nd/4.0/)下的开放访问文章。结论:各种降级药物类别的全脑C-FOS表达特征指向一组离散的大脑区域和神经信号,这些区域可能代表未来抗肥胖疗法的关键神经解剖靶标。
基于新能源发电改进型移相全桥变换器研究
中图分类号 : TM561 Analysis of Improved Phase-shift Full-bridge Converter for New Energy Generation ZENG Zhihui 1, 2 LIU Yunpeng 1, 2 ZHANG Linmei 1, 2 YANG Ming 1, 2
用于耦合全阶和降阶模型的多保真计算
混合物理-机器学习模型越来越多地用于传输过程的模拟。许多与科学和工程应用相关的复杂多物理系统包括多个时空尺度,并包含一个多保真度问题,该问题在各种公式或异构计算实体之间共享一个接口。为此,我们提出了一种强大的混合分析和建模方法,结合基于物理的全阶模型 (FOM) 和数据驱动的降阶模型 (ROM),形成混合保真度描述中面向预测数字孪生技术的集成方法的构建块。在界面上,我们引入了一个长短期记忆网络,以各种形式的界面误差校正或延长来桥接这些高保真度和低保真度模型。所提出的界面学习方法被测试为一种解决 ROM-FOM 耦合问题的新方法,使用双保真度设置解决非线性平流扩散流情况,该设置可以捕捉广泛传输过程的本质。
非洲绿猴(African Green Monkey)全基因组CRISPR sgRNA ...
目的基因 sgRNA 数目: 64853 ;阴性对照 sgRNA 数目: 2000 ; sgRNA 大小: 20bp
全基因组长阅读测序降采样及其对变化精度和回忆
高通量基因分型能够对种群基因组学和全基因组关联研究中的遗传多样性进行大规模分析,这些研究结合了大量加入的基因型和表型表征。基于测序的基因分型方法由于较低的确定性偏差而逐渐替换传统的基因分型方法。然而,基于测序的全基因分型在具有较大基因组和高比例的重复性DNA的物种中变得昂贵。在这里,我们描述了CRISPR-CAS9技术在3.76-Gb基因组(镜头Culinaris)中耗尽重复元素,84%由重复序列组成,从而将测序数据集中在编码和调节区域(单子拷贝区域)上。我们设计了一组566,766个GRNA,旨在重复2.9英镑,排除了基于ATACSQ数据的重复区域重复的注释基因和推定的调节元素。新颖的耗竭方法去除了〜40%的读取映射到重复序列,从而将这些映射到单拷贝区域增加了约2.6倍。在分析2500万个片段时,与非部位的文库相比,测序数据中的重复对单个拷贝偏移增加了约10倍。在相同的条件下,我们还能够鉴定单拷贝区域中的遗传变异量增加了12倍,并通过挽救杂合变体的特征来提高基因分型精度,否则由于覆盖范围较低,否则会遗漏这些变体。该方法的执行方式类似,无论多路复用水平,文库类型或基因型,包括不同的品种和密切相关的物种(L. Orientalis)。我们的结果表明,CRISPR-CAS9驱动的重复耗竭将测序数据集中在单拷贝区域上,从而改善了大型和重复的基因组中的高密度和全基因组基因分型。
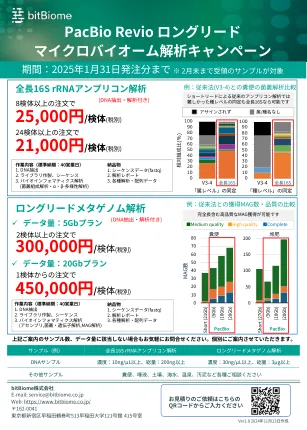
全长 16S rRNA 扩增子分析
bitBiome Inc. 电子邮件:service@bitbiome.co.jp 网站:https://www.bitbiome.co.jp/ 日本东京新宿区早稻田鹤卷町 513 号 162-0041 早稻田大学第 121 栋 415 室