XiaoMi-AI文件搜索系统
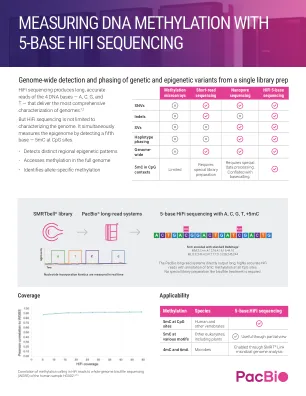
World File Search System用5-碱基HIFI测序测量DNA甲基化
仅研究使用。不适用于诊断程序。©2023加利福尼亚州的太平洋生物科学(“ PACBIO”)。保留所有权利。此文档中的信息如有更改,恕不另行通知。PACBIO对本文档中的任何错误或遗漏不承担任何责任。某些通知,条款,条件和/或使用限制可能与您使用PACBIO产品和/或第三方产品有关。请参阅适用的PACBIO销售条款和条件以及PACB.com/license的适用许可条款。Pacific Biosciences,PACBIO徽标,PACBIO,Circulomics,Omniome,Smrt,Smrtbell,Iso-Seq,Seqel,Seqel,Nanobind,SBB,Revio,Revio,Onso,Apton,Apton和Kinnex是PacBio的商标。
腺嘌呤碱基编辑器介导的剪接重塑激活非规范剪接位点
、闫彤 1 、陈浩然 1 、王嘉华 1 、王英怡 4 、杨叶琴 5 、项略 1 、池在龙 1 、任开群 2 、林斌 6 、林戈 7,8 、李劲松 3,4 、刘勇 1,* 和顾锋 1,2,9,* 来自 1 温州医科大学附属眼视光学院、卫生部视觉科学国家重点实验室、卫生部重点实验室和浙江省眼视光重点实验室,浙江省温州;2 湖南师范大学医学院、湖南省模式动物与干细胞生物学重点实验室、生殖与转化医学湖南省工程研究中心,长沙,中国; 3 中国科学院上海生物化学与细胞生物学研究所、上海分子男科学重点实验室、细胞生物学国家重点实验室、分子细胞科学卓越中心,上海,中国;4 上海科技大学生命科学与技术学院,上海,中国;5 浙江中医药大学护理学院,浙江杭州,中国;6 香港理工大学眼科视光学院,香港,中国;7 中信湘雅生殖与遗传医院,湖南省生殖与遗传临床研究中心,长沙,中国;8 中南大学基础医学院生殖与干细胞工程研究所,长沙,中国;9 湖南师范大学附属广秀医院(湖南广秀医院),长沙,中国
穿梭肽在体内将碱基编辑器 RNP 递送至恒河猴气道上皮细胞
我们在此报告了首次证明穿梭肽在恒河猴模型中将蛋白质和 ABE8e-Cas9 RNP 递送至呼吸道上皮的转化潜力。在单次气雾剂给药后,我们成功地将荧光标记的蛋白质货物递送至大气道和小气道的上皮细胞以及一些肺泡上皮。使用 S315 穿梭肽进行 ABE8e-Cas9 RNP 递送,我们在使用支气管刷回收的细胞中实现了 CCR5 基因座的显著 A 到 G 编辑。从气管和近端气道收获的上皮中 CCR5 位点的编辑效率达到 5.3%。在具有 R553X 突变的人类 CF 气道上皮中应用这种递送方法实现了类似的编辑水平并赋予 CFTR 功能的部分恢复。
细菌中无约束的碱基编辑
。cc-by-nc-nd 4.0国际许可证。根据作者/资助者提供了预印本(未经同行评审认证)提供的,他已授予Biorxiv的许可证,以在2023年11月12日发布的此版本中在版权所有者中显示预印本。 https://doi.org/10.1101/2023.04.16.537106 doi:Biorxiv Preprint
体内腺嘌呤碱基编辑纠正新生小鼠 Hurler 综合征模型
© 作者 2023。开放存取 本文根据知识共享署名 4.0 国际许可协议进行授权,允许以任何媒体或格式使用、共享、改编、分发和复制,只要您给予原作者和来源适当的信任,提供知识共享许可协议的链接,并指明是否做出了更改。 本文中的图片或其他第三方资料包含在文章的知识共享许可协议中,除非资料的致谢中另有说明。 如果资料未包含在文章的知识共享许可协议中,且您的预期用途不被法定规定允许或超出了允许的用途,则需要直接从版权所有者处获得许可。 要查看此许可证的副本,请访问 http://creat iveco mmons. org/licen ses/ by/4. 0/。
Transformer 碱基编辑器在哺乳动物细胞和小鼠中的设计与应用
将载脂蛋白 B mRNA 编辑酶、催化性多肽样胞苷脱氨酶与催化功能受损的 Cas 蛋白(例如 nCas9 或 dCas9)融合,提供了一种新型基因编辑技术,即碱基编辑,可高效地实现靶向碱基替换。然而,在碱基编辑中观察到全基因组和全转录组脱靶突变,这引发了对治疗应用的安全性担忧。之前,我们开发了一种新的碱基编辑系统,即 transformer 碱基编辑器 (tBE),可在哺乳动物细胞和小鼠中诱导高效编辑,且不会观察到全基因组或全转录组脱靶突变。这里我们描述了设计和应用 tBE 的详细方案。本方案包括设计单向导 RNA (sgRNA) 和辅助 sgRNA 对、构建构建体、确定全基因组和转录组范围的脱靶突变、生产含有 tBE 的腺相关病毒、将腺相关病毒递送到小鼠体内以及检查体内编辑效果的步骤。使用 sgRNA-辅助 sgRNA 对,tBE 的高精度碱基编辑可以在 2-3 周内(在哺乳动物细胞中)或 6-8 周内(在小鼠中)完成。整个过程可以由研究人员使用分子生物学、生物信息学和小鼠饲养的标准技术共同完成。
利用随机多路复用 sgRNA 组装碱基编辑器进行定向进化水稻基因
摘要 基于 CRISPR 的定向进化是一种有效的育种生物技术,可改善植物的农艺性状。然而,使用单个单向导 RNA 其基因多样化仍然有限。我们在这里描述了一种多重正交碱基编辑器 (MoBE) 和一种随机多重 sgRNA 组装策略,以最大化基因多样化。MoBE 可以在不同的靶标上有效诱导正交 ABE (< 36.6%)、CBE (< 36.0%) 和 A&CBE (< 37.6%),而 sgRNA 组装策略将各种靶标上的碱基编辑事件随机化。对于水稻乙酰辅酶 A 羧化酶 (OsACC) 第 34 外显子的每一条链上的 130 个和 84 个靶标,我们在随机双 sgRNA 和随机三重 sgRNA 文库中观察到多达 27 294 种靶标-支架组合类型。我们进一步利用MoBE和随机双sgRNA文库对水稻中的OsACC进行了定向进化,获得了更强的除草剂抗性的单突变或连锁突变。这些策略可用于功能基因的原位定向进化,并可能加速水稻性状改良。
联合基因型和表型结果建模改进了碱基编辑变异效应的量化
是作者/资助者,已授予 medRxiv 永久展示预印本的许可。 (未经同行评审认证)预印本此版本的版权持有者于 2023 年 9 月 10 日发布。;https://doi.org/10.1101/2023.09.08.23295253 doi:medRxiv 预印本
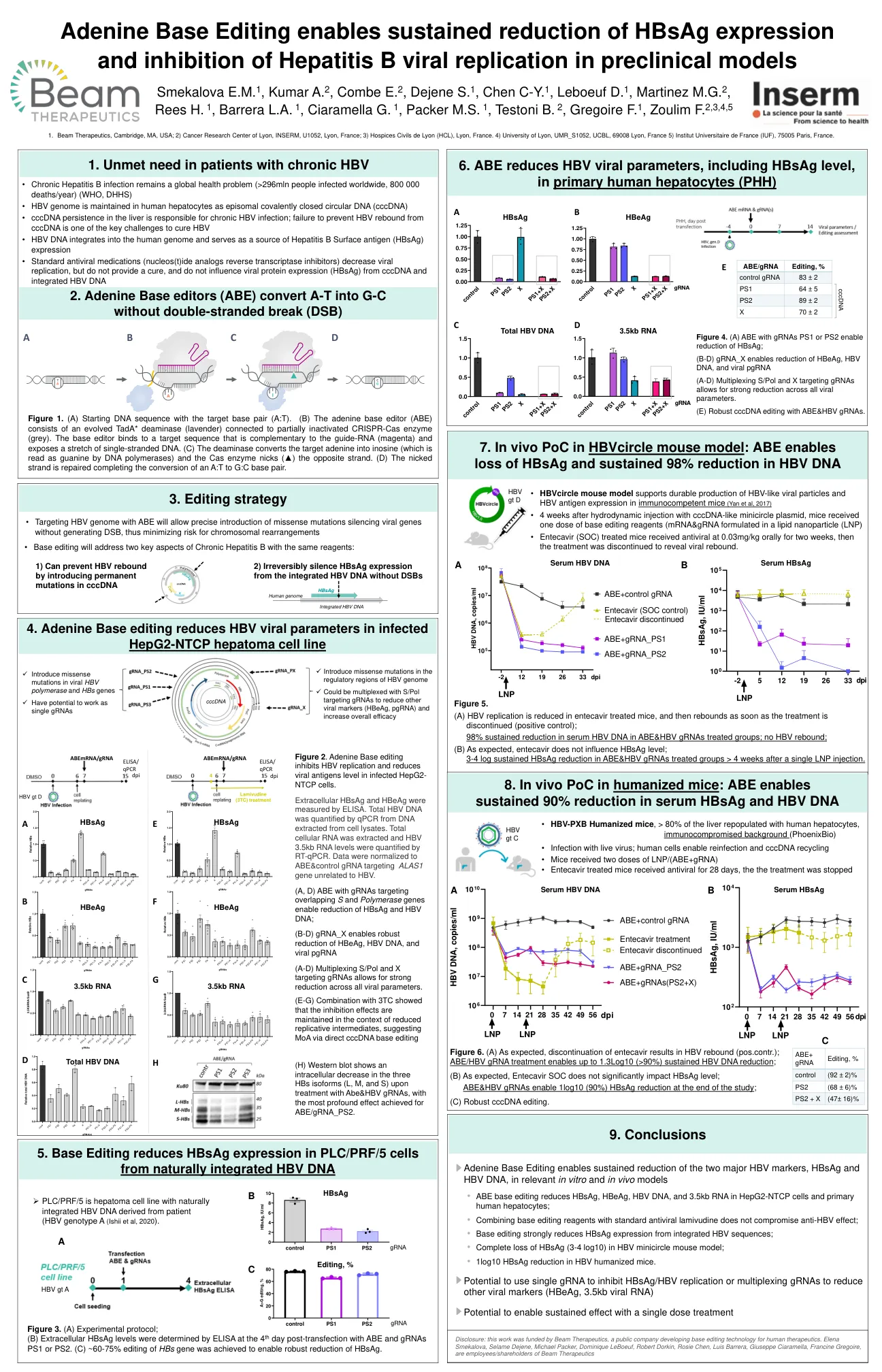
腺嘌呤碱基编辑能够在相关的体外和体内模型中持续降低两种主要的 HBV 标志物 HBsAg 和 HBV DNA
图 1. (A) 起始 DNA 序列,其中包含目标碱基对 (A:T)。(B) 腺嘌呤碱基编辑器 (ABE) 由进化的 TadA* 脱氨酶 (淡紫色) 和部分失活的 CRISPR-Cas 酶 (灰色) 组成。碱基编辑器与与向导 RNA (洋红色) 互补的目标序列结合,并暴露一段单链 DNA。(C) 脱氨酶将目标腺嘌呤转化为肌苷 (DNA 聚合酶将其读取为鸟嘌呤),Cas 酶切口 (▲) 另一条链。(D) 切口链被修复,完成从 A:T 到 G:C 碱基对的转换。
实现多千碱基长距离传输的方法和技术......
1 CRISP-HR Therapeutics,加利福尼亚州圣卡洛斯 94070 摘要 CRISPR 支持的细胞和基因疗法有可能彻底改变遗传医学领域。然而,由于当前工具和技术的局限性,绝大多数罕见病仍然无法治愈。到目前为止,大多数矫正治疗方法仅限于逐个突变的方法,其中 HDR 或较新的技术(如碱基编辑或主要编辑)一次重写小片段 DNA(~1-100 bp)。虽然这些方法很强大,但短编辑窗口(相对于人类基因的大小)在经济和/或技术上与大多数罕见疾病突变谱不兼容。在这里,我们首次证明 CRISPR/Cas9 可用于通过我们称为“长距离重写”的无选择过程同时“重写”人类基因组的 7kb+ 部分。长距离重写方法与多种核酸酶、细胞类型和基因组位点兼容,并且可以与基于双链断裂 (DSB) 和非 DSB 的方法一起使用。简介 CRISPR 和相关技术已被证明在从基础研发到工业以及最近的治疗应用的各个领域都非常有用。CRISPR/Cas9 系统最基本的用途是针对基因组中的特定 DNA 片段并创建小的插入或删除 (INDEL) 来破坏各种遗传元素(蛋白质编码序列、启动子/增强子、剪接位点等)。1–4 虽然这是一种强大的技术,但由于“破坏”基因可产生治疗益处的疾病非常有限,因此该方法在治疗应用方面受到限制。此外,人们越来越担心通过有害的双链断裂 (DSB) 介导的编辑产生的不良局部和全局副作用。5,6 因此,人们一直在努力开发工具和技术,以实现更可编程和更复杂的编辑,同时减少不良副作用。这些努力促成了各种具有扩展功能的 Cas9 融合蛋白的开发,例如 Base 和 Prime 编辑器 7,8 、CRISPRa 和 CRISPRi 9,10 、旨在操纵内源性修复途径选择的 Cas 蛋白工程 11-13 ,以及无数其他功能 14 。这些努力使得我们能够选择性地替换特定碱基、将任何碱基交换为另一个碱基、在不进行基因组修饰的情况下调节基因表达,以及操纵