XiaoMi-AI文件搜索系统
World File Search System使用深度学习来识别分子连接特征
图5。1 -like(浅蓝色)或2个(洋红色)痕迹的1D直方图由混合溶液测量的不同模型排序。基于使用0.4 nm片段作为输入的分类结果显示为实线。作为参考,使用剪切迹线作为输入的分类结果在此重现为阴影区域。(a)应用于0.4 nm片段的CNN模型会产生3066 1类和5216 2类样痕迹(与3406 1类似于3406 1 -like和4876 2-在使用完整迹线时喜欢痕迹)。(b)应用于0.4 nm片段的PC 1 /1DH模型产生6053 1类和2229 2类样痕迹(与4397 1-类似于4397 1 -like和3901 2 -like tlace时,使用完整的痕迹时)。(c)应用于0.4 nm片段的KMeans/2DH模型产生392 1-类似于7890 2-像痕迹(与5260 1 -like和3022 2 -2 -like Traces相比,当使用完整的迹线时)。(d)应用于0.4 nm片段的逻辑回归模型产生的4730 1类和3553 2类样痕迹(与4569 1-类似于4569 1的痕迹和3713 2 -2 -like tlace tlace时使用完整痕迹时)。
具有改良发光特性的高度非化化YAG陶瓷
图2。y 3+x al 5-x o 12(0≤x≤0.4)的结构演变得出了SXRD数据的分析。(a)Y 3.4 Al 4.6 O 12(R WP = 8.79%,χ= 1.16)的Rietveld细化具有高角度拟合插图的变焦。Blue tick marks indicate garnet reflections (99.77(2) wt.%), green tick marks indicate perovskite reflections (YAlO 3 , 0.33(2) wt.%) (b) The garnet structure of Y 3.4 Al 4.6 O 12 projected along (100), and a fragment projected along (111) showing the three different cation environments (orange atoms = Y 3+ ; dark blue octahedra = Alo 6;浅蓝色四面体= ALO 4)。(c)具有线性拟合覆盖(实线)的精制晶格参数A,并通过y 3+对16个位点的精制占用率,名义占用覆盖(虚线)。(d)在三种不同的阳离子环境中精制的金属氧距离(m-o)x,在y 3 al 5 o 12(m- o)0时标准化为其值。蓝色三角形=直接结晶样品;洋红色倒三角=玻璃结晶样品。错误栏对应于细化中的10x ESD。
基因组知识的圈养育种可以减少近亲抑郁症和动物园种群的遗传负荷
fi g u r e 1从单个粉红色鸽子的原始阅读中,粉红色依赖性耗竭(PPCADD)分数的每单核苷酸多态性(SNP)粉红色鸽子的产生管道。Snakemake(Mölder等,2021)管道用作输入主体个体的测序读数,受试者物种参考基因组以及CADD分数和参考基因组(即鸡肉,Chcadd分数(Groß,Bortoluzzi等,2020)和Galgal6参考基因组(Warren等,2017))。管道分为六个部分,对应于管道的部分(https://github。com/saspe ak/loadlift)。(1)(黄色)使用Phyluce从参考基因组中提取UCE。(2)(深蓝色)映射个体的测序读取到参考基因组,以指示10×Chromium读取数据(本文中使用)和Illumina读取数据的两种平行方法。(3)(浅蓝色)变体呼叫UCES中的SNP。(4)(浅灰色)创建链文件,用于从鸡基因组转化注释。(5)(深灰色)Chcadd得分转换为粉红色鸽子(主题物种)注释。(6)(绿色)床文件和UCE站点的交集到每个站点PPCADD(主题物种)分数(红色)。
癌细胞国际诱导基因表达
图1创建合成cAMP响应元件结合蛋白(CREB)响应启动子。(a)腺苷信号传导的描述。腺苷(红色球)结合腺苷受体A2AR/A2BR,该腺苷受体动员相关的G蛋白(绿色)激活腺苷酸环化酶(橙色受体),并将ATP转化为3'5'- 5'-循环腺苷单磷酸腺苷(Camp)。另外,福斯科蛋白(橙色球)可以直接激活腺苷循环酶。CAMP结合蛋白激酶A(PKA)与磷酸化的CREB,该CREB结合了Plindromic DNA基序“ TGACGTCA”,激活了基因表达。(b)启动子设计和筛选示意图。cAMP响应元件基序(CRE,突出显示的黄色)被克隆在3倍重复中,两侧是鸟嘌呤“ G”(带下划线),六个散布的填充核苷酸(N)。3x Cres(灰色正方形)放在核心启动子(蓝色箭头)上游的1-6个重复中。用高斯荧光素酶(GLUC)或绿色荧光蛋白(EGFP)定量启动子活性。(c,d)HEK293T细胞在96个井板中用指示的构建体(x轴)反向转染。转染后48小时,用车辆(DMSO,浅蓝色条)或20μm福斯科林(FSK,深蓝色条)将细胞介质更改为培养基。八个小时后,对培养基进行了采样并测试了GLUC活性(RLU)。条表示n = 3实验重复的平均值,误差线代表标准误差(SEM)。**通过方差分析(ANOVA)Tukey检验,与所有其他样本相比,表示P <0.01。(E,F)流式细胞仪启动子诱导。HEK293T细胞用96个井板中的指定构建体(x轴)反向转染。转染后48小时,细胞培养基被更改为未处理的培养基(浅蓝色条),或补充了0.750 m m m腺苷(ADO,深蓝色条)的培养基。八个小时后,将细胞胰蛋白酶胰蛋白酶进行胰蛋白酶,并将其重悬于FACS缓冲液中以进行流式细胞仪。y轴表示正向散射(FSC)单元的EGFP中位荧光强度。条代表n = 3实验重复的平均值,误差线代表SEM。(g)启动子对腺苷的剂量反应性。HEK293T细胞在96个井板上反向转染,并在传说中指示的构造,然后培养48小时。然后更改培养基以添加不同的腺苷浓度,在8小时后进行采样,并测试了GLUC活性(RLU)。**通过12倍-CRE_YB的ANOVA TUKEY测试代表P <0.01,与1 m m的所有其他样品相比。每个点表示n = 3实验重复的平均值,误差线为SEM。
Lou Barbe、Cendrine Mony、Benjamin W Abbott。人工智能通过视频游戏意外学习了生态学。《生态学与进化趋势》,2020 年,35 (7),第 557-560 页。�10.1016/j.tree.2020.04.006�。�hal-02635148�
星际争霸 II 中的经典对抗类型,由 AlphaStar 扮演,由两名玩家在资源有限的特定环境中相互对抗 — 参见“迷你地图”(图 I)。这代表两个个体之间的生态竞争,可以是种内竞争,也可以是种间竞争,这取决于玩家是否选择同一种族。地图显示了整个环境,但玩家的视野仅限于各自单位和建筑物周围较浅的圆形区域。资源是浅蓝色形状,深蓝色和红色形状是双方的建筑物和单位(Protoss 为蓝色,Terran 为红色)。可以通过这个迷你地图监控竞争的进展和结果,它显示了新资源斑块的殖民和开发、环境探索以及通常的生态崩溃。这张地图可用于监控更现实的生态模型。例如,几个玩家可以在更大且完全不可预测的环境中相互竞争,这将使我们能够研究人口和社区规模的生态过程。我们还可以设置场景,在游戏过程中人为地修改环境条件,然后评估对生态系统功能和生物多样性动态的影响。请注意,使用游戏参数可以轻松量化几个生态过程,如特征变化或权衡修改。图 I. 星际争霸 II 标准游戏的迷你地图。
发现新型药物类抗结核病靶向药物...
图 2 :(A) DZT 与 Dr DXPS 晶体结构(PDBID:2O1X)的顶级对接姿势。通过直接去除共结晶的 ThDP 制备 38 ApoDr DXPS,并在 Mg 2+ 存在下进行对接(本文其他对接操作相同)。(B)DZT 对 Dr DXPS 的抑制模式研究以及 DZT 对 Mt DXPS 和 H304A 突变体的剂量反应曲线。颜色代码:参考条件:紫色;变化的 [ThDP]:绿色;变化的 [PYR]:蓝色;变化的 [ D -GAP]:红色。(C)ThDP 和 DZT 的药效团视图。颜色代码:C 骨架:DZT:浅蓝色;ThDP:洋红色;His304(Dr DXPS):灰色;His296(Mt DXPS):棕色。表面:疏水位点:绿色;亲水位点:红色。(D)Dr DXPS (WT)、Dr DXPS (H304A) 和 Mt DXPS 的动力学表征(见图 S1 中的曲线)。(E)化合物 1 与 Dr DXPS 与 His304 相互作用的顶级对接姿势。(F)化合物 2 与 Dr DXPS 与 H304 相互作用的顶级对接姿势。本文中的所有对接研究均使用软件 LeadIT 44 进行;图 2C 使用 MOE 45 生成;图 2A、2E 和 2F 使用 Poseview 生成。46
Alt-R™ HDR ENHANCER V2 提高效率... - NET
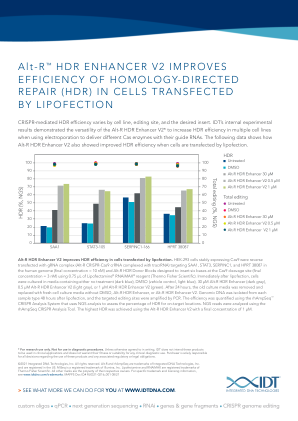
Alt-R HDR Enhancer V2 可提高脂质转染细胞的 HDR 效率。使用 0.75 μL Lipofectamine ® RNAiMAX ® 试剂(赛默飞世尔科技)将稳定表达 Cas9 的 HEK-293 细胞反向转染 gRNA 复合物(Alt-R CRISPR-Cas9 crRNA 与 tracrRNA 复合),靶向人类基因组中的 SAA1、STAT3、SERPINC1 和 HPRT 38087(最终浓度 = 10 nM)和设计用于在 Cas9 裂解位点插入六个碱基的 Alt-R HDR 供体块(最终浓度 = 3 nM)。脂质转染后,立即将细胞培养在含有无处理(深蓝色)、DMSO(载体对照,浅蓝色)、30 μM Alt-R HDR 增强剂(深灰色)、0.5 μM Alt-R HDR 增强剂 V2(浅灰色)或 1 μM Alt-R HDR 增强剂 V2(绿色)的培养基中。24 小时后,移除旧培养基,并用不含 DMSO、Alt-R HDR 增强剂或 Alt-R HDR 增强剂 V2 的新鲜细胞培养基替换。脂质转染 48 小时后从每种样本类型中分离基因组 DNA,并通过 PCR 扩增目标编辑位点。使用 rhAmpSeq ™ CRISPR 分析系统对效率进行量化,该系统使用 NGS 分析来评估目标位置的 HDR 百分比。使用 rhAmpSeq CRISPR 分析工具分析了 NGS 读数。使用最终浓度为 1 μM 的 Alt-R HDR 增强剂 V2 实现了最高的 HDR。
αkg介导的肉碱合成可通过组蛋白乙酰化促进同源重组
GSK864(IDH1I)和DNA破坏特工Olaparib(OLAP)或顺铂(CIS)单独或合并。%,并将其标准化为对照。d)在谷氨酰胺饥饿的条件下培养了指定的CCNE1-低(橙色)和 - 高(紫色)同源细胞,并单独或单独或单独或合并用DNA损害剂Olaparib(OLAP)或顺铂(Cisplatin(Cis)处理。%的细胞,并将其标准化为媒介物对照。e)将IP基因细胞注射到免疫功能低下的雌性小鼠(n = 8/组)中。表达空载体(ev)=橙色的单元格;表达CCNE1(CCNE1)=紫色的细胞。单独或组合使用媒介物,IDH1抑制剂GSK864(IDH1I)和Olaparib(OLAP)处理小鼠。在端点,通过计算腹膜肿瘤结节来计算肿瘤负担。f)仅用IDH1抑制剂GSK864(IDH1I)处理指示的CCNE1高细胞,单独使用DNA破坏药物Olaparib(OLAP)或顺铂(CIS)(CIS)(cis)(黄色)(黄色)(黄色)(黄色),并与细胞渗透性的A kg(绿色)或柠檬酸盐(蓝色)结合使用。%,并将其标准化为对照。g)在谷氨酰胺饥饿条件下培养了指定的CCNE1高细胞,并用DNA损伤剂Olaparib(OLAP)或顺铂(CIS)(CIS)(CIS)(黄色)和可渗透的细胞渗透kg(绿色)处理。%的细胞,并将其标准化为媒介物对照。h)依赖性二氧酶CRISPR KO屏幕的示意图。i)CRISPR KO屏幕的分析。所有图表示平均值±SD。显示为Log2折叠分数(CCNE1 + Olaparib vs. CCNE1)与(EV + Olaparib vs.EV)中的负分数的变化。J)在两个CCNE1高细胞系中5个负富集基因的Venn图。k)用SHGFP(Shcont-紫色)或两个靶向TMLHE的独立shRNA(SHTMLHE#1-浅蓝色,浅蓝色,SHTMLHE#2-深蓝色)转导指示的CCNE1高细胞,并用DNA损害剂Olaparib(Olap)用Cell-Cell-clip-carn的dna损害剂处理(la)或l-CARNIT(l-CARNIT)。%的细胞,并将其标准化为媒介物对照。l)单独使用肉碱合成抑制剂(Mildro)或单独使用DNA损伤剂Olaparib(OLAP)(紫色)和组合(黄色)处理指示的CCNE1高细胞。组合处理的细胞用可渗透的细胞A kg(绿色)或L- carnitine(L-Carn; Maroon)补充。%的细胞,并将其标准化为媒介物对照。m)用IDH1抑制剂GSK864(IDH1I)和单独的DNA损伤剂Olaparib(OLAP)(紫色)和组合(黄色)处理指示的CCNE1高细胞。组合处理的细胞用L-肉碱(L-Carn; Maroon)补充。%的细胞,并将其标准化为媒介物对照。n)在谷氨酰胺饥饿条件下(紫色)培养指示的CCNE1-高细胞,并单独用DNA损伤剂Olaparib(OLAP)(黄色)或补充L-Carnitine(L-Carn; Ma-Roon)。%,并将其标准化为对照。**** p <0.005,ns =不显着o)kg是tmlhe和carnitine上游的示意图。(A-D,F)显示的是来自每个等源性细胞系对中至少3个独立实验的代表性数据。(G,K-N)是来自每个等源性细胞系对中2个独立实验的代表性数据。
适应性癌症治疗中的阈值意识
图 1:EGT 模型中的确定性最优策略。(GLY-VOP-DEF) 三角形代表各个亚群所有可能的相对丰度。由于该策略是 bang-bang 策略,我们使用黄色背景(其中应以 MTD 速率使用药物)和蓝色背景(其中根本不应使用药物)来显示它。从初始状态 (q0,p0) = (0.26,0.665)(洋红色点)开始,子图显示 (a) 从真正确定性驱动的系统 (2.14) 中找到的最优轨迹,成本为 5.13;(b) 在确定性最优策略下生成的两个代表性样本路径,但受到随机适应度扰动的影响(较亮的一个成本为 3.33,而另一个成本为 6.23); (c) 使用 10 5 个随机模拟近似的累积成本 J 的 CDF。在 (a) 和 (b) 中,轨迹的绿色部分对应于不开药,轨迹的红色部分对应于以 MTD 率开药。在 (a) 中,确定性情况下的价值函数的水平集以浅蓝色显示。在 (c) 中,蓝色曲线是使用确定性最优策略 d ⋆ 生成的 CDF。其在成功条件下观察到的中位数和平均值分别为 4.95 和 4.91。棕色曲线是使用基于 MTD 的疗法生成的 CDF,在此示例中,它还最大限度地提高了“不受预算约束”的肿瘤稳定的机会。其在成功条件下观察到的中位数和平均值分别为 5.95 和 5.96。橙色和粉色曲线显示了两种不同的阈值感知策略的 CDF(分别为 ¯ s = 4 . 5 和 ¯ s = 5)。每个曲线上的大点表示不超过相应阈值的最大化概率。术语“阈值特定优势”是指在 ¯ s 时,d ¯ s ∗ 的 CDF 高于所有其他策略的 CDF。
Nirsevimab 2025
警报于2024年11月10日,澳大利亚政府宣布,根据2025年国家免疫计划(NIP),将向符合条件的孕妇提供预防RSV的ABRYSVO®疫苗。从2025年2月3日起,新的RSV母体疫苗Abrysvo®将在28至36周妊娠的孕妇国家免疫计划(NIP)中提供。从2025年3月17日开始,将为单克隆抗体Nirsevimab提供不受母体疫苗接种或患有严重RSV疾病的某些医疗危险因素保护的婴儿。由于国际供应,资格标准正在迅速变化,并取决于澳大利亚的基于州资助的计划。请参阅相关的部委/卫生部门,以获取您州或地区的最新资格信息。指示预防严重呼吸道合胞病毒(RSV)疾病动作长效重组中和中和人IgG单克隆抗体,该抗体与RSV蛋白的F蛋白的F1和F2亚基结合。提供了针对严重RSV感染的被动免疫。药物类型单克隆抗体商品名称Beyfortus演示1。Beyfortus 50mg在0.5毫升(100mg/ml)的单剂量预填充注射器(紫色柱塞杆)2。Beyfortus 100mg(100mg/ml)的单剂量预填充注射器(浅蓝色柱塞杆)剂量合格的婴儿 * *资格标准在各州各不相同。请参阅相关的部委/卫生部门,以获取有关您的州或地区的急诊资格信息。新南威尔士州NIRSEVIMAB的符合条件的婴儿新南威尔士州RSV预防计划2025将全年实施,而不仅仅是季节性。所有孕妇在妊娠28-36周之间,接受单剂量的RSV疫苗Abrysvo。