XiaoMi-AI文件搜索系统
World File Search SystemRNA编辑增加了SARS的核苷酸多样性-...
SARS-CoV-2 是一种正义单链 RNA 病毒,导致了 COVID-19 大流行。目前尚不清楚人类宿主细胞中的病毒是否以及在多大程度上经历了 RNA 编辑,这是一种主要的 RNA 修饰机制。在这里,我们对来自多个 COVID-19 患者支气管肺泡灌洗液样本的宏转录组数据进行了可靠的生物信息学分析,揭示了 SARS-CoV-2 中相当数量的 A-to-I RNA 编辑候选位点。我们通过评估 RNA 编辑特有的四个特征来确认这些候选位点上 A-to-I RNA 编辑信号的富集:推断的 RNA 编辑位点表现出 (i) 由基于 ADAR1 CLIP-seq 数据构建的深度学习模型预测的更强的 ADAR1 结合亲和力,(ii) ADAR1 抑制的人肺细胞中的编辑水平降低,(iii) 局部聚类模式,和 (iv) 更高的 RNA 二级结构倾向。我们的研究结果对于了解 SARS-CoV-2 的进化以及 COVID-19 研究(例如系统发育分析和疫苗开发)具有重要意义。
核糖核苷酸还原酶,淋病的新药物
抽象抗生素耐药酸酯(NG)是由于增加的多药耐药性(MDR)生物的增加而成为新兴的公共卫生威胁。我们确定了两个新型的口服活性抑制剂PTC-847和PTC-672,它们表现出狭窄的活性范围,包括NG,包括MDR分离株。通过选择对新型抑制剂有抗性的生物并测序其基因组,我们确定了一个新的治疗靶标,即IA核糖核苷酸还原酶(RNR)。在Ng MAP中分解突变与α亚基的N末端锥结构域,我们在这里显示的是在存在β亚基和变构效应子DatP的情况下形成抑制的α4β4状态。 酶测定确认PTC-847和PTC-672抑制NG RNR,并揭示了变构效应器DATP增强了抑制作用。 口服PTC-672的口服降低了小鼠模型中的NG感染,并且可能具有治疗对当前药物具有抗药性的治疗的治疗潜力。分解突变与α亚基的N末端锥结构域,我们在这里显示的是在存在β亚基和变构效应子DatP的情况下形成抑制的α4β4状态。酶测定确认PTC-847和PTC-672抑制NG RNR,并揭示了变构效应器DATP增强了抑制作用。口服PTC-672的口服降低了小鼠模型中的NG感染,并且可能具有治疗对当前药物具有抗药性的治疗的治疗潜力。口服PTC-672的口服降低了小鼠模型中的NG感染,并且可能具有治疗对当前药物具有抗药性的治疗的治疗潜力。
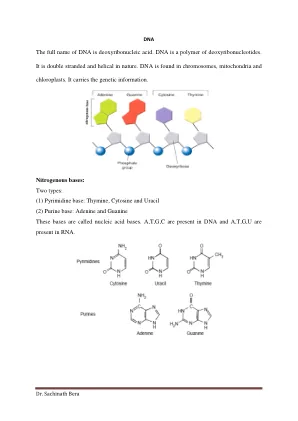
第一个自我复制分子是RNA核苷酸。
生命的起源;第一个自我复制分子是RNA核苷酸。K。Ohsaka Freelancer,CA USA上的抽象难以有效地合成RNA核苷酸,通过在模拟的益生元地球环境中加入其亚基在现代实验室中,这使我们提出了通过诸如矿物质的矿物质,当然是良好的猫症,并在良好的猫科动物等地上,通过交叉免费的自我复制来提出一个替代过程。该过程发生在具有循环环境变化的区域,例如由于潮汐的上升和下降,潮湿和潮湿的周期重复的潮湿和潮湿。核苷酸(单体)和多核苷酸(聚合物)的自我复制可被视为不断发展的生命的起源,也可以视为RNA遗传的原因。在聚合过程中自然建立了RNA的同R.。自我复制能够传递分子信息,并允许突变和自然选择,生命的基本进化过程。1。引言生活一直在通过自我复制,突变和自然选择过程发展。流行的思想表明,生命源于RNA核苷酸的聚合,这是通过间接证据和一些实验结果证实的,被称为RNA世界[1,2]。在现代实验室中,正在持续努力将RNA核苷酸与核碱基腺嘌呤(a短),尿嘧啶(U),鸟嘌呤(G)和胞嘧啶(C)合成,从简单的分子成分开始,可能是从可能存在于益生物土位上的简单分子成分开始的[3-7]。另外,某些中间产品可能起源于外太空并传递到地球。看来,整个过程导致RNA核苷酸的三个分子亚基,即核仁酶,核糖糖(S)和磷酸盐组(P)发生在益生元土中。在陨石中发现的证据表明这种可能性[8]。相比之下,最后一个过程,通过连接亚基来合成RNA核苷酸的合成很困难,因为必须将它们与适当的防治性和立体特异性构型一起连接在一起,并且需要克服高激活能量[9]。因此,必须有一个布置亚基并降低活化能以有效形成核苷酸的过程。一旦RNA核苷酸的浓度达到一定水平,就发生了聚合,并且在益生元土中合成了单链多核苷酸。在模拟的益生元条件下使用非生物催化剂的实验表明,单链多核苷酸可以长达50个核苷酸单位[10]。最大长度取决于多核苷酸的稳定性,后者不断受到解离(聚合物链破裂)。与已知的短函数RNA(约100个单位)的长度相比,最大长度很短。随着多核苷酸的长度,解离速率线性增加。为了进一步生长,必须在益生元土中进行多核苷酸稳定的过程。
cc-seq:Spo11 DNA的核苷酸分辨率映射...
。cc-by 4.0国际许可(未经Peer Review尚未获得认证)是作者/资助者,他已授予Biorxiv的许可证,以永久显示预印本。它是制作
酶促DNA合成的人工核苷酸密码子
核糖体将核酸中编码的遗传信息转化为蛋白质。即使将氨基酸逐一组装在一起,这种解码过程也需要mRNA上的三核苷酸密码子与同源氨基酰基-TRNA的相应反密码子之间的watson-Crick相互作用。遗传密码是退化的,由于序列柔韧性主要在第三核苷酸的水平上,因此由一个或多个TRNA识别。1,2另一方面,核酸的合成是由聚合酶介导的,并通过在生长链上组装单个单字母核苷酸来进行进行。由于机制的差异,这些基本生物聚合物的合成涉及的错误率大大差异从非常低的DNA复制到更容易出错的DNA转录到mRNA中,以及将mRNA转换为蛋白质的较小的忠诚度(分别为〜10 -8,〜10 -5,〜10 -5,〜10 -5,〜10 -10 -4,误差率将mRNA转换为蛋白质。3,4
酶促DNA合成的人工核苷酸密码子
1。S. Ye和J. Lehmann。 ,2022,50,4113-4 2。 F. V.支持和K. T. Hughes,Proc。 natl。 学院。 SCI。 U.S.A.,2017,114,4745-4750。 3。 K. Mohler和M. Ibba,Nat。 微生物。 ,2017,2,17117。 4。 J. M. M. Ogle和V. Ramakrishnan,Annu。 修订版 生物化学。 ,2005,74,129-1 5。 J. W. Chinese,A。Cropp,J。C. Anderson,M。 6。 M. A. Shandell,F。Cornish的太阳,2021,60,3455-3469。 7。 P. Ghosh,H。M. Cross,K。 am。 化学。 Soc。 ,2022,144,10556-1 8。 N. Freed,M。J。J. J. opine。 生物技术。 ,2022,74,129-1 9。 N. Freund,A。I。Taylor,St.Franklin,N。Subraman,S.-Y。 Peak-Chew,A。M. Whitaker,B。D. Freudental,M。Abramov,P。Holliger,Nat。 化学。 ,2023,15,91-1 10。 J. R. D. D. Freund,G。G。G. G. Dalwal,P。Holly和A. I. Taylor,RSC Chem。 大。 ,2022,3,1209-1 11。 C. Liu,C。Cozens,F。Jaziri,J。Rozenski,A。Marshal,St.Dumbre,V。 am。 化学。S. Ye和J. Lehmann。,2022,50,4113-42。F. V.支持和K. T. Hughes,Proc。natl。学院。SCI。 U.S.A.,2017,114,4745-4750。 3。 K. Mohler和M. Ibba,Nat。 微生物。 ,2017,2,17117。 4。 J. M. M. Ogle和V. Ramakrishnan,Annu。 修订版 生物化学。 ,2005,74,129-1 5。 J. W. Chinese,A。Cropp,J。C. Anderson,M。 6。 M. A. Shandell,F。Cornish的太阳,2021,60,3455-3469。 7。 P. Ghosh,H。M. Cross,K。 am。 化学。 Soc。 ,2022,144,10556-1 8。 N. Freed,M。J。J. J. opine。 生物技术。 ,2022,74,129-1 9。 N. Freund,A。I。Taylor,St.Franklin,N。Subraman,S.-Y。 Peak-Chew,A。M. Whitaker,B。D. Freudental,M。Abramov,P。Holliger,Nat。 化学。 ,2023,15,91-1 10。 J. R. D. D. Freund,G。G。G. G. Dalwal,P。Holly和A. I. Taylor,RSC Chem。 大。 ,2022,3,1209-1 11。 C. Liu,C。Cozens,F。Jaziri,J。Rozenski,A。Marshal,St.Dumbre,V。 am。 化学。SCI。U.S.A.,2017,114,4745-4750。 3。 K. Mohler和M. Ibba,Nat。 微生物。 ,2017,2,17117。 4。 J. M. M. Ogle和V. Ramakrishnan,Annu。 修订版 生物化学。 ,2005,74,129-1 5。 J. W. Chinese,A。Cropp,J。C. Anderson,M。 6。 M. A. Shandell,F。Cornish的太阳,2021,60,3455-3469。 7。 P. Ghosh,H。M. Cross,K。 am。 化学。 Soc。 ,2022,144,10556-1 8。 N. Freed,M。J。J. J. opine。 生物技术。 ,2022,74,129-1 9。 N. Freund,A。I。Taylor,St.Franklin,N。Subraman,S.-Y。 Peak-Chew,A。M. Whitaker,B。D. Freudental,M。Abramov,P。Holliger,Nat。 化学。 ,2023,15,91-1 10。 J. R. D. D. Freund,G。G。G. G. Dalwal,P。Holly和A. I. Taylor,RSC Chem。 大。 ,2022,3,1209-1 11。 C. Liu,C。Cozens,F。Jaziri,J。Rozenski,A。Marshal,St.Dumbre,V。 am。 化学。U.S.A.,2017,114,4745-4750。3。K. Mohler和M. Ibba,Nat。 微生物。 ,2017,2,17117。 4。 J. M. M. Ogle和V. Ramakrishnan,Annu。 修订版 生物化学。 ,2005,74,129-1 5。 J. W. Chinese,A。Cropp,J。C. Anderson,M。 6。 M. A. Shandell,F。Cornish的太阳,2021,60,3455-3469。 7。 P. Ghosh,H。M. Cross,K。 am。 化学。 Soc。 ,2022,144,10556-1 8。 N. Freed,M。J。J. J. opine。 生物技术。 ,2022,74,129-1 9。 N. Freund,A。I。Taylor,St.Franklin,N。Subraman,S.-Y。 Peak-Chew,A。M. Whitaker,B。D. Freudental,M。Abramov,P。Holliger,Nat。 化学。 ,2023,15,91-1 10。 J. R. D. D. Freund,G。G。G. G. Dalwal,P。Holly和A. I. Taylor,RSC Chem。 大。 ,2022,3,1209-1 11。 C. Liu,C。Cozens,F。Jaziri,J。Rozenski,A。Marshal,St.Dumbre,V。 am。 化学。K. Mohler和M. Ibba,Nat。微生物。,2017,2,17117。4。J. M. M. Ogle和V. Ramakrishnan,Annu。修订版生物化学。,2005,74,129-15。J. W. Chinese,A。Cropp,J。C. Anderson,M。 6。 M. A. Shandell,F。Cornish的太阳,2021,60,3455-3469。 7。 P. Ghosh,H。M. Cross,K。 am。 化学。 Soc。 ,2022,144,10556-1 8。 N. Freed,M。J。J. J. opine。 生物技术。 ,2022,74,129-1 9。 N. Freund,A。I。Taylor,St.Franklin,N。Subraman,S.-Y。 Peak-Chew,A。M. Whitaker,B。D. Freudental,M。Abramov,P。Holliger,Nat。 化学。 ,2023,15,91-1 10。 J. R. D. D. Freund,G。G。G. G. Dalwal,P。Holly和A. I. Taylor,RSC Chem。 大。 ,2022,3,1209-1 11。 C. Liu,C。Cozens,F。Jaziri,J。Rozenski,A。Marshal,St.Dumbre,V。 am。 化学。J. W. Chinese,A。Cropp,J。C. Anderson,M。6。M. A. Shandell,F。Cornish的太阳,2021,60,3455-3469。7。P. Ghosh,H。M. Cross,K。 am。 化学。 Soc。 ,2022,144,10556-1 8。 N. Freed,M。J。J. J. opine。 生物技术。 ,2022,74,129-1 9。 N. Freund,A。I。Taylor,St.Franklin,N。Subraman,S.-Y。 Peak-Chew,A。M. Whitaker,B。D. Freudental,M。Abramov,P。Holliger,Nat。 化学。 ,2023,15,91-1 10。 J. R. D. D. Freund,G。G。G. G. Dalwal,P。Holly和A. I. Taylor,RSC Chem。 大。 ,2022,3,1209-1 11。 C. Liu,C。Cozens,F。Jaziri,J。Rozenski,A。Marshal,St.Dumbre,V。 am。 化学。P. Ghosh,H。M. Cross,K。am。化学。Soc。,2022,144,10556-18。N. Freed,M。J。J. J.opine。生物技术。,2022,74,129-19。N. Freund,A。I。Taylor,St.Franklin,N。Subraman,S.-Y。 Peak-Chew,A。M. Whitaker,B。D. Freudental,M。Abramov,P。Holliger,Nat。 化学。 ,2023,15,91-1 10。 J. R. D. D. Freund,G。G。G. G. Dalwal,P。Holly和A. I. Taylor,RSC Chem。 大。 ,2022,3,1209-1 11。 C. Liu,C。Cozens,F。Jaziri,J。Rozenski,A。Marshal,St.Dumbre,V。 am。 化学。N. Freund,A。I。Taylor,St.Franklin,N。Subraman,S.-Y。Peak-Chew,A。M. Whitaker,B。D. Freudental,M。Abramov,P。Holliger,Nat。化学。,2023,15,91-110。J. R. D. D. Freund,G。G。G. G. Dalwal,P。Holly和A. I. Taylor,RSC Chem。大。,2022,3,1209-111。C. Liu,C。Cozens,F。Jaziri,J。Rozenski,A。Marshal,St.Dumbre,V。am。化学。Soc。,2018,140,6690-612。C. A. A. Jerome,St。Hoshika,K。M。Bradley,St.A。natl。学院。SCI。 美国,2022,119,226111SCI。美国,2022,119,226111
核苷酸还原酶,淋病的新型药物靶点
摘要 由于耐多药 (MDR) 菌的数量不断增加,抗生素耐药性淋病奈瑟菌 (Ng) 正成为一种新出现的公共卫生威胁。我们发现了两种新型口服抑制剂 PTC-847 和 PTC-672,它们对 Ng 包括 MDR 分离株表现出较窄的活性谱。通过筛选对新型抑制剂有耐药性的菌并对其基因组进行测序,我们确定了一个新的治疗靶点——Ia 类核苷酸还原酶 (RNR)。Ng 中的耐药突变位于 α 亚基的 N 端锥体结构域,我们在此显示在 β 亚基和变构效应物 dATP 存在的情况下,该结构域参与形成受抑制的 α 4 β 4 状态。酶测定证实 PTC-847 和 PTC-672 抑制 Ng RNR,并揭示变构效应物 dATP 增强了抑制作用。口服 PTC-672 可以减少小鼠模型中的 Ng 感染,并且可能对治疗对当前药物有耐药性的 Ng 具有治疗潜力。
鉴定DNA结合基序结构和核苷酸序列
皮革,P.B。大自然,359,505-511)Marblestin,R.,Carey,M.,Ptashne,M。和Harrison,S.C。(1992)自然,356,408-412)Nikolov,D.B.,Hu,S.H,Lin,J.,Gasch,A.,Hoffmann,A.,Horikoshi,M.,Chua,N.H.,Roeder,R.G。和Bur-Ley,S.K。 (1992)自然,360,40-46。 13)Pabo,C.O。 和Sauer,R.T。 (1992)安。 修订版 生物化学。 61,1053-1 14)Branden,C。和Tooze,J。 (1991)Pro- 简介和Bur-Ley,S.K。(1992)自然,360,40-46。13)Pabo,C.O。和Sauer,R.T。 (1992)安。修订版生物化学。61,1053-114)Branden,C。和Tooze,J。(1991)Pro-
在单核苷酸分辨率的DNA中测序尿嘧啶
。cc-by-nd 4.0国际许可证(未经同行评审证明)获得的是作者/资助者,他已授予Biorxiv的许可证,以永久显示预印本。这是该版本的版权持有人,该版本发布于2021年6月21日。 https://doi.org/10.1101/2021.06.20.449152 doi:biorxiv preprint