XiaoMi-AI文件搜索系统
World File Search System补充材料
补充材料。材料与方法文库制备和 Miseq (Illumina®) 测序使用文库制备指南 (LPG) ( https://support.illumina.com/downloads/16s_metagenomic_sequencing_library_preparation.html ) 中报告的 Illumina 接头序列和引物悬垂部分(正向和反向)扩增 16S rRNA 基因的 460 bp V3-V4 高变区。使用以下 PCR 反应扩增每个 DNA 样本:2.5 µl 5 ng/ µl DNA、5 µl 引物正向悬垂部分、5 µl 引物反向悬垂部分、12.5 µl 2x KAPA HiFi HotStart ReadyMix (KAPA Biosystems)。使用 LPG 中报告的循环程序。 PCR 产物在 2% 琼脂糖凝胶(GellyPhor LE,Euroclone SPA,意大利米兰)上电泳分离,并用 GelRed™ 核酸凝胶染料(Biotium,美国加利福尼亚州海沃德)染色。通过紫外光透射仪观察预期长度的 PCR 产物的存在。然后,用 NucleoMag 试剂盒纯化 DNA 扩增子以清理和选择 NGS 文库制备反应的大小(Macherey-Nagel),并按照制造商的说明使用 Illumina® DNA/RNA UD Indexes Tagmentation 试剂盒对每个样本进行索引。在验证和定量之前,对文库进行进一步纯化。在 Agilent 4150 TapeStation D1000 ScreenTape 检测仪(安捷伦科技公司)上对文库进行验证,以验证大小,而定量则使用 Qubit 4 荧光计(赛默飞世尔科技,美国)。根据 DNA 扩增子的大小,应用 Illumina LPG 中报告的公式,以 nM 为单位计算最终的 DNA 浓度。最后,将每个文库中的 5 µl 稀释 DNA 等分试样混合,以合并具有唯一索引的文库。在 Miseq 加载之前,根据 Illumina LPG 说明对合并的文库进行变性和稀释。使用 MiSeq Reagent Micro Kit v2(500 个循环)加载合并的文库,运行包括 20% PhiX 作为内部对照。生物信息学分析测序数据包含在包含带有原始读取的 FASTQ 文件的文件夹中(R1 文件包含每个样本的正向读取,R2 文件包含每个样本的反向读取),使用 FastQC(英国剑桥 Babraham Institute)进行质量检查。然后,使用 DADA2 R 包(Callahan 等人,2016 年)处理 R1 和 R2 文件以生成扩增子序列变体 (ASV)(图 1)。最终生成了 ASV 表,总结了每个样本的不同 ASV 的数量。
瑞典蜂蜜中的乳酸细菌在美国fulbrood爆发期间
J.,Grochowalski,。,Strapagiel,D.,Gnat,S.,Załuski,D.,Gancarz,M.,Rusinek,R.,Krutmuang,P.,MartínHernánandez,R.对来自真菌和植物的细菌及其2个区域的可变16S rRNA的扩增子测序,使蜜蜂对疾病的易感性重新易感性,这是由于其在人为景观下的饲料可用性而引起的。病原体,10,381。https://doi。Org/10. 3390/Patho Gens1 0030381 R Core Team。(2022)。r:用于统计计算的语言和环境。r统计计算基础。https://www.r- proje ct。(2018)。肠道微生物组在
对于 TakeCare 实验室分析的取样人员
家族性高胆固醇血症(FH)的基因研究。调查包括两个阶段:1)卡罗琳斯卡 FH 面板,这是一种 SNP 面板测试,使用 Agena MassARRAY 系统和 iPLEX Gold 化学方法(根据 SWEDAC 的 ISO/IEC 17025 认证)分析三个最重要的 FH 基因中已知的 119 个突变和缺失。 2) 使用卡罗琳斯卡 FH 面板未发现突变的样本将使用 Devyser 的 FH v2 扩增子测序方法和 Illumina 的 NextSeq 仪器进行进一步分析。根据样本类型选择分析:血液、纯化 DNA 或唾液。
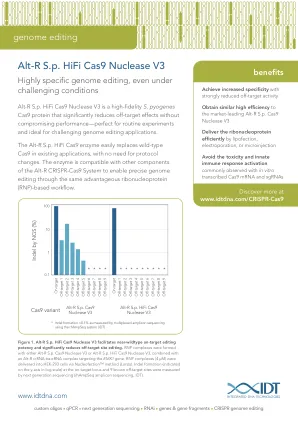
IDT Alt-R Sp HiFi Cas9 核酸酶 V3 (CRS-10083-FL 03/19)
图 1. Alt-R Sp HiFi Cas9 Nuclease V3 促进近野生型靶向编辑效力并显著减少脱靶位点编辑。RNP 复合物由 Alt-R Sp Cas9 Nuclease V3 或 Alt-R Sp HiFi Cas9 Nuclease V3 与靶向 EMX1 基因的 Alt-R crRNA:tracrRNA 复合物结合形成。RNP 复合物 (4 µM) 通过 Nucleofection™ 方法 (Lonza) 递送到 HEK-293 细胞中。通过下一代测序 (rhAmpSeq 扩增子测序,IDT) 测量了靶位点和 9 个已知脱靶位点处的插入/缺失形成 (在 y 轴上以对数标度表示)。
20240806 CRISPR RNP 案例研究布局
与 CRISPR QC 合作为科学家提供了优化基因编辑策略的强大优势。我们的协作方法与我们的尖端平台相结合,可以全面评估 CRISPR 工作流程中的每个步骤。CRISPR 分析平台使科学家能够定量评估 gRNA 和 Cas 核酸酶复合物的结合和稳定性,确定 RNP 复合物与靶标(扩增子)DNA 之间的结合效率,测试靶标和非靶标 DNA 样本中的 RNP CRISPR 复合物裂解频率,并分析成功多重编辑的关键动力学排名。通过利用这些工具,我们可以帮助科学家快速获得可操作的见解,以优化他们的 CRISPR 策略。
种子微生物组和生物信息学(M/f/d) 意大利的MSCA机会| EURAXESS “非洲倡议II” -Ueraxess-欧盟 BEPA和BATT4EU合作伙伴关系简介
微生物学,生物信息学,计算生物学或相关领域的博士学位。具有高通量测序数据分析(例如元基因组学,扩增子测序)及其在种子或植物微生物中的应用。强大的编程技能(例如Python,R)和对生物信息学工具(例如Qiime2,dada2,Silva)的熟悉。出版记录,显示了微生物组科学或相关领域的高质量研究,尤其是植物或种子微生物组。微生物技术的知识,包括种子相关微生物的培养和表征。愿意为研究建议和项目报告做出贡献。英语中出色的沟通技巧;对德语的了解是一个优势。能够独立工作并作为协作团队的一部分。
化学修饰的 AsCas12a 向导 RNA 可改善不同组织中脂质纳米颗粒介导的体内基因编辑
(Cytiva)。LNP 货物是编码工程 AsCas12a 核酸酶和 gRNA 的 mRNA,重量比为 1:1。通过 RiboGreen 测定法(ThermoFisher Scientific)评估 LNP 的包封率大于 80%,多分散性指数 (PDI) <0.2,通过 Zetasizer 分析(Malvern Panalytical,型号 ZSU3205)评估平均直径大小 <105 nm。• 细胞培养处理:用指定浓度的包封 AsCas12a mRNA 的 LNP 处理细胞,并在转染后 72 小时分离 gDNA。原代人肝细胞 (PHH) 的转染包括重组人载脂蛋白 E。进行基于扩增子的下一代测序 (NGS) 以确定编辑百分比。 • 小鼠眼内体内编辑:通过前房内注射将 LNP 递送至每只 hMYOC Y437H(具有 Y437H 突变的人类肌动蛋白基因)敲入小鼠的一只眼中。注射后一周,解剖眼球,从前房分离 mRNA。采用基于转录本的 RT-ddPCR 检测来测量剩余 hMYOC mRNA 的程度。 • 小鼠肝脏内体内编辑:通过尾静脉静脉注射将 LNP 递送至 hMYOC Y437H 小鼠。注射后一周,解剖肝脏,分离 gDNA,并进行基于扩增子的 NGS 以确定编辑百分比。 • 体外结合亲和力测量:将标记的未修饰的向导与重组工程 AsCas12a 和浓度不断增加的修饰“测试”向导混合,在室温下孵育 3 小时,然后在硝酸纤维素印迹膜 (Cytiva) 和 Hybond N+ 膜 (Cytiva) 上进行双重过滤分离。在每个膜上定量荧光标记的未修饰向导,并计算结合的未修饰向导的百分比。标记的未修饰向导的结合百分比降低是由于来自修饰的“测试”向导的结合竞争。
通过先进的原型技术探索安第斯高海拔湖泊极端微生物
摘要:快速鉴定和表征来自极端环境的分离物目前是一项挑战,但对于探索地球的生物多样性却非常重要。由于这些分离物原则上可能与已知物种有远亲关系,因此需要采用技术来可靠地鉴定它们所属的生命分支。通过串联质谱法对这些环境分离物进行蛋白质分型提供了一种快速且经济有效的方法,可以使用它们的肽谱进行鉴定。在本研究中,我们记录了第一种用于环境嗜极菌和嗜盐菌分离物的高通量蛋白质分型方法。微生物是从智利高原高海拔安第斯山脉湖泊(海拔 3700 - 4300 米)的样本中分离出来的,这些湖泊代表的地球环境与其他星球的条件相似。总共培养了 66 种微生物,并通过蛋白质分型和 16S rRNA 基因扩增子测序进行了鉴定。两种方法对所有分离物都揭示了相同的属鉴定结果,但三种分离物除外,这三种分离物可能代表尚未根据其肽组进行分类学表征的生物。蛋白质分型能够表明副球菌科和 Chromatiaceae/Alteromonadaceae 科中存在两个潜在的新属,而这些属仅被 16S rRNA 扩增子测序方法所忽略。本文强调,蛋白质分型有可能发现来自极端环境的未描述的微生物。关键词:串联质谱蛋白质分型、阿塔卡马沙漠、高原、高海拔安第斯山脉湖泊、极端微生物、嗜盐菌■简介
种子微生物组和生物信息学(M/f/d) 博士学位的科学家(100%)专注于人工... dofdoc土壤微生物组和动物群(m/f/d)(100%) 生物信息学(M/f/d)中的DostDoc
微生物学,生物信息学,计算生物学或相关领域的博士学位。具有高通量测序数据分析(例如元基因组学,扩增子测序)及其在种子或植物微生物中的应用。强大的编程技能(例如Python,R)和对生物信息学工具(例如Qiime2,dada2,Silva)的熟悉。出版记录,显示了微生物组科学或相关领域的高质量研究,尤其是植物或种子微生物组。微生物技术的知识,包括种子相关微生物的培养和表征。愿意为研究建议和项目报告做出贡献。英语中出色的沟通技巧;对德语的了解是一个优势。能够独立工作并作为协作团队的一部分。