机构名称:
¥ 1.0
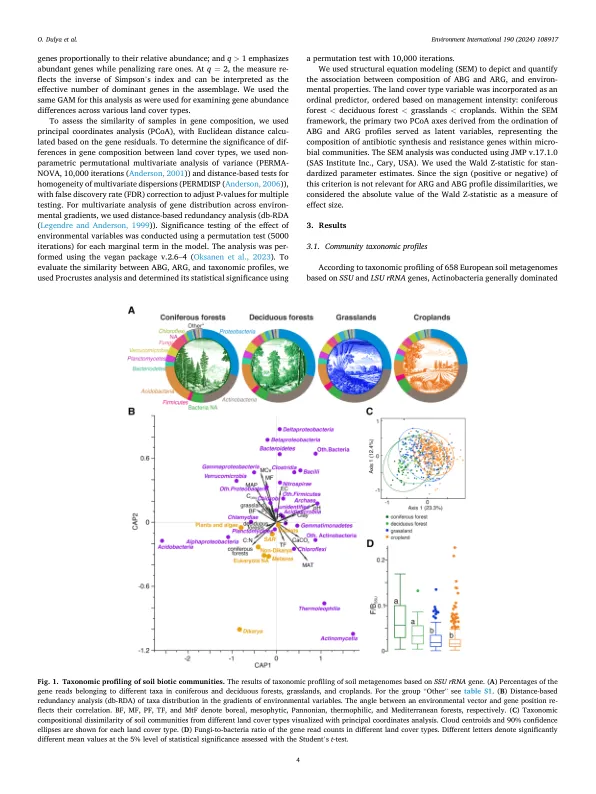
抗生素耐药性危机决定了对抗药性监测的需求和寻找新的抗生素。受监测方案的开发受到了许多阻力因素的多样性的阻碍,而“街道光效应”否认了基于现有数据库发现新型药物的可能性。在这项研究中,我们使用从基于特征的生态学角度观察的高通量环境筛查来解决这些挑战。通过对跨越欧洲的658个表土样品的宏基因组进行深入分析,我们探索了241个原核生物和真菌基因的分布,负责生产具有抗生物性特性和485种抗生素耐药性基因的代谢物。我们分析了这些基因收集的多样性,并在环境梯度上对每个基因的分布进行了建模。我们的分析揭示了编码酶促途径的顺序步骤的几种非平行分布模式,该基因合成了大型抗生素基团,指出了现有数据库中的差距,并提出了发现已知抗生素的新类似物的潜力。我们表明,农业活动引起了微生物抗生素相关机械的大陆规模均匀化,这强调了在景观镶嵌中维持本地生态系统的重要性。基于宏基因组中基因比例与主要预测因子(土壤pH,土地覆盖类型,气候温度和湿度)之间的关系,我们说明了化学结构的性质如何决定跨环境中其合成的基因的分布。有了这种理解,我们提出了一般原则,以促进抗生素的发现,包括主要是新的,建立了抗生素抗性基因的丰富基准,并预测它们的传播。
环境国际
主要关键词