机构名称:
¥ 1.0
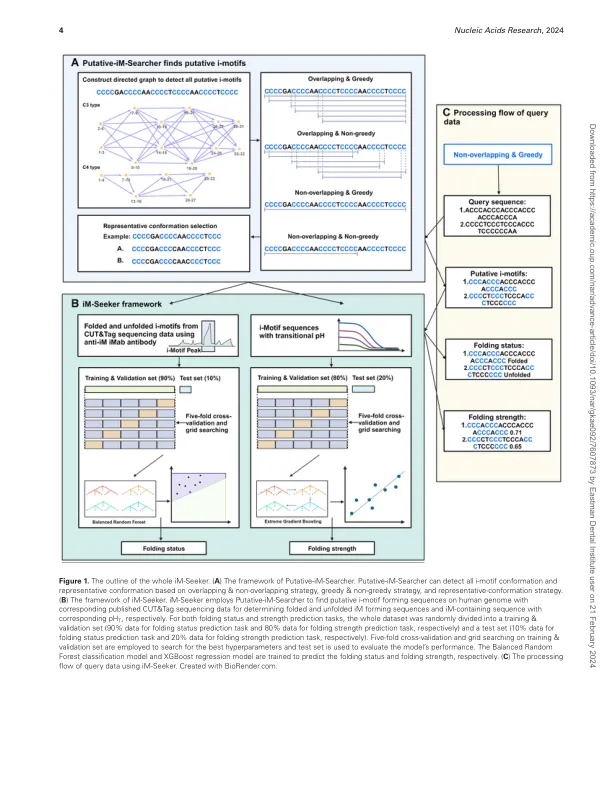
摘要i-motifs(IMS)是在富含细胞质的DNA序列中形成的次级str uct uct uct,在基因组中的多个功能中均在v olv中。尽管Putativ e Im forming序列被广泛分布在人类基因组中,但推定的IMS的折叠状态和强度变化了。muc h先前的研究IM已重点是使用生物含量xperiments评估IM折叠特性。ho w e v er,没有专门的计算工具来预测IM结构的折叠状态和强度。在这里,我们介绍了一条机器学习管道,即IM-Weeker,以预测DNA IMS的折叠状态和结构性折叠状态。该程序Im-seeker结合了一个平衡的随机森林分类器,该森林分类器在全基因组IMAB抗体基于基于IMAB的剪切和标记测序数据中训练,以预测折叠状态和极端的梯度增强回归器,以根据文献生物物理数据和我们的内部生物物理实验来估算折叠强度。im-seeker以81%的分类精度预测DNA IM F旧状态,并在测试集上以0.642的确定系数(R 2)估算了F旧强度。模型的解释证实,富含C的序列的核苷酸组成显着影响Im stabilit Y,与含有胞嘧啶和胸腺氨酸的序列具有正相关,并且与鸟嘌呤和腺嘌呤的负相关。
通过机器学习的DNA I -MOTIF的预测-UCL Discovery
主要关键词