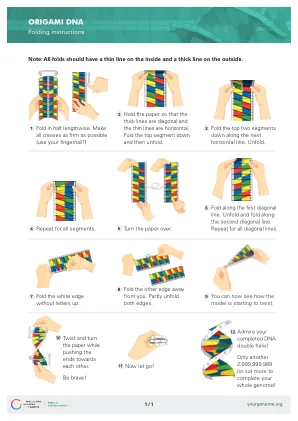
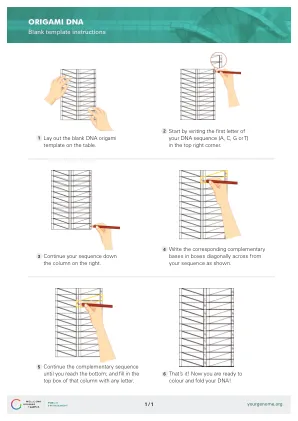
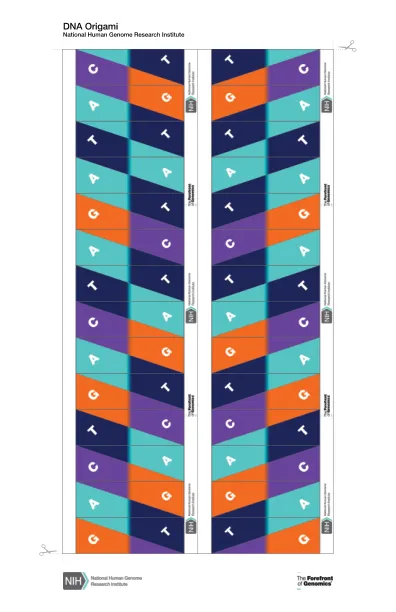
机构名称:
¥ 1.0
许多实验和计算工作试图了解DNA折叠的折叠,但是此过程的时间和长度尺寸构成了显着的挑战。在这里,我们提出了一种使用可切换力场的介观模型来捕获单链和双链DNA基序的行为以及它们之间的过渡,从而使我们能够模拟DNA折纸的折叠,最多可达几个千千目标。对小结构的布朗动力学模拟揭示了一个层次折叠过程,涉及将其拉入的折叠前体,然后结晶成最终结构。我们阐明了各种设计选择对折叠顺序和动力学的影响。较大的结构显示出异质的主食掺入动力学,并且在亚稳态状态中频繁捕获,而不是表现出第一阶动力学和实际上无缺陷的折叠的更容易接近的结构。该模型开辟了一条途径,以更好地理解和设计DNA纳米结构,以提高产量和折叠性能。
通过介观模拟阐明的DNA折纸折叠的机制
主要关键词