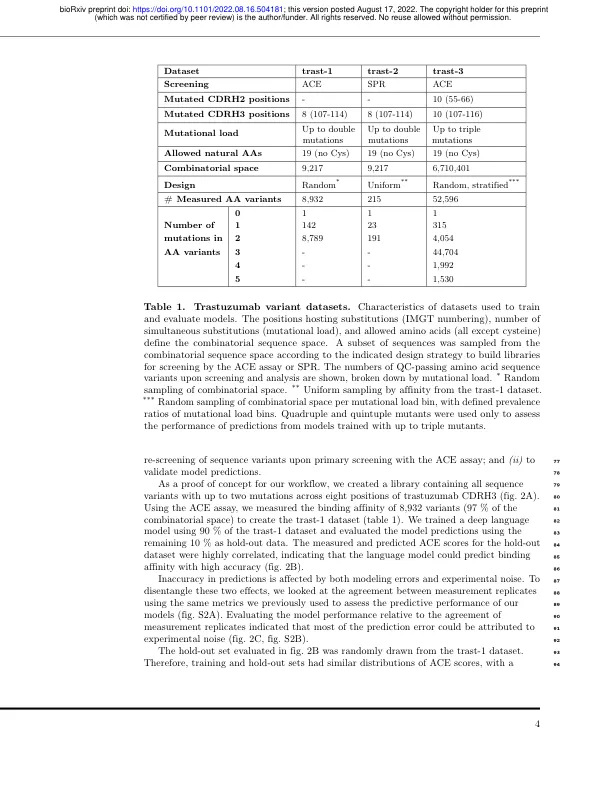
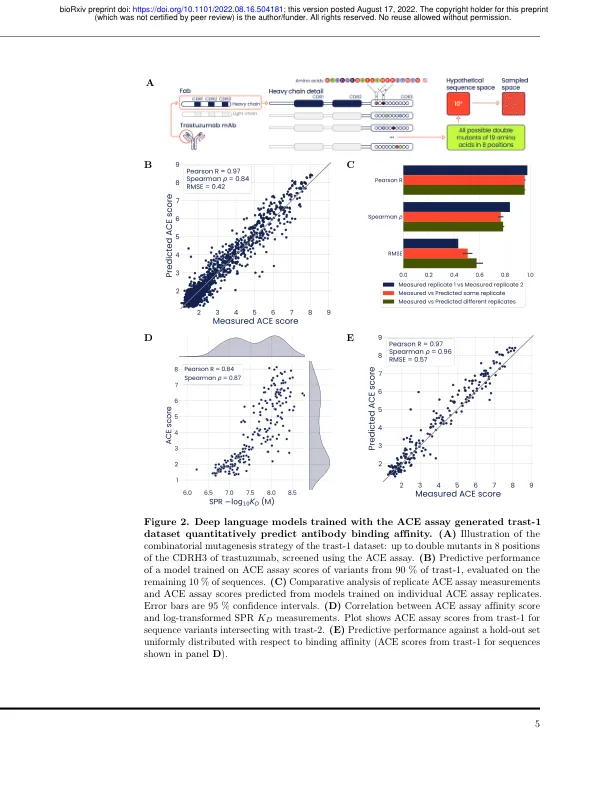
机构名称:
¥ 3.0
传统的抗体优化方法涉及筛选可用序列空间的一小部分,通常会导致候选药物具有次优的结合亲和力、可开发性或免疫原性。基于两种不同的抗体,我们证明,在高通量亲和力数据上训练的深度上下文语言模型可以定量预测未见抗体序列变体的结合。这些变体在很大的突变空间中跨越了三个数量级的 KD 范围。我们的模型显示出强大的上位效应,这凸显了对智能筛选方法的需求。此外,我们引入了“天然性”的建模,这是一种对抗体变体与天然免疫球蛋白的相似性进行评分的指标。我们表明,天然性与药物可开发性和免疫原性的指标有关,并且可以使用遗传算法与结合亲和力一起对其进行优化。这种方法有望加速和改善抗体工程,并可能提高开发新型抗体和相关候选药物的成功率。
通过人工智能预测结合亲和力和自然性实现抗体优化