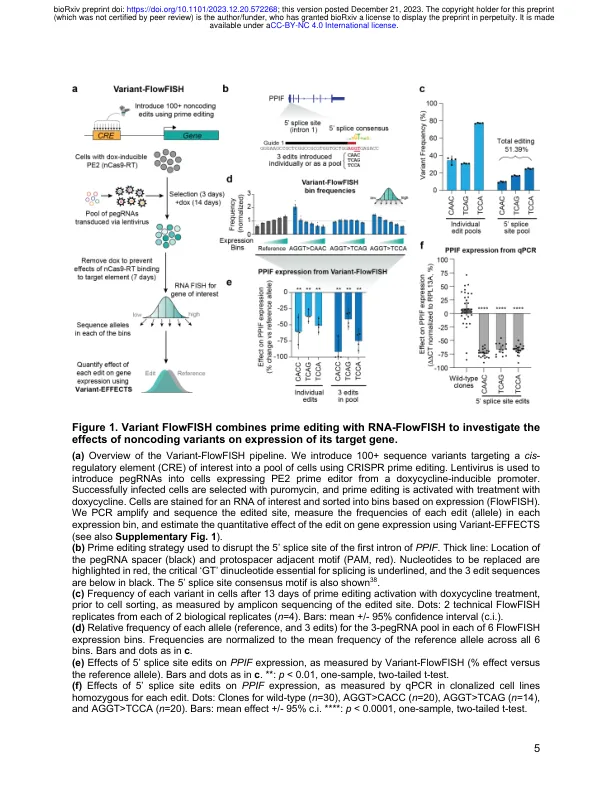
1美国加利福尼亚州斯坦福大学医学院,美国加利福尼亚州斯坦福大学2基础科学与工程倡议,斯坦福儿童健康,贝蒂·艾琳·摩尔·摩尔儿童心脏中心,美国加利福尼亚州斯坦福,美国加利福尼亚州斯坦福大学3卡利科人生活科学,南旧金山,加利福尼亚州南旧金山,加利福尼亚州,加利福尼亚州,加利福尼亚州,美国4号计算机科学系,美国斯坦福大学。麻省理工学院和哈佛研究所,美国马萨诸塞州剑桥市6基因法规天文台,麻省理工学院和哈佛大学,马萨诸塞州剑桥,美国7 7号麻省理工学院和哈佛大学,马萨诸塞州剑桥大学,美国,美国剑桥8次地址:美国马萨诸塞州剑桥,马萨诸塞州剑桥市,美国,美国9号,美国马萨诸塞州,美国马萨诸塞州,美国摩托学,美国10号。美国加利福尼亚州斯坦福大学斯坦福大学的心血管研究所 *同等贡献。任何作者都可以首先列出。摘要:增强子和启动子内的调节DNA序列结合转录因子与编码基因表达的细胞类型特异性模式。但是,此类DNA序列的调节效应和可编程性仍然难以映射或预测,因为我们缺乏可扩展的方法来精确编辑调节性DNA并量化内源基因组环境中的效果。在这里,我们提出了一种方法,可以通过将合并的CRISPR Prime编辑与RNA荧光原位杂交和细胞分选(变体流鱼)相结合,来衡量数百种设计的DNA序列变体对基因表达的定量效应。我们将这种方法应用于在两个免疫细胞系中PPIF的增强子和PPIF启动子中的调节DNA序列。672变体类型对,我们识别影响PPIF表达的497。这些变体似乎通过多种机制作用,包括破坏或优化现有的转录因子结合位点以及创建从头站点。破坏单个内源性转录因子结合位点通常会导致表达变化很大(增强子的–40%,启动子中的–50%)。相同的变体通常在细胞类型和状态上具有不同的影响,表明了高度可调的调节景观。我们使用这些数据来基准基于基因调节的基于序列的预测模型的性能,并发现某些类型的变体无法通过现有模型准确预测。最后,我们在计算上设计了185个小序列变体(<10 bp),并优化它们以对硅中表达的特定影响。这些合理设计的编辑中有84%显示出预期的效果方向,有些对表达产生了巨大影响(–100%至 +202%)。变体 - 流鱼因此提供了一种强大的工具,可以绘制变异和转录因子结合位点对基因表达,测试和改善基因调控的计算模型以及重编程调节DNA的影响。
重写调节性DNA以剖析和重编程基因表达
主要关键词