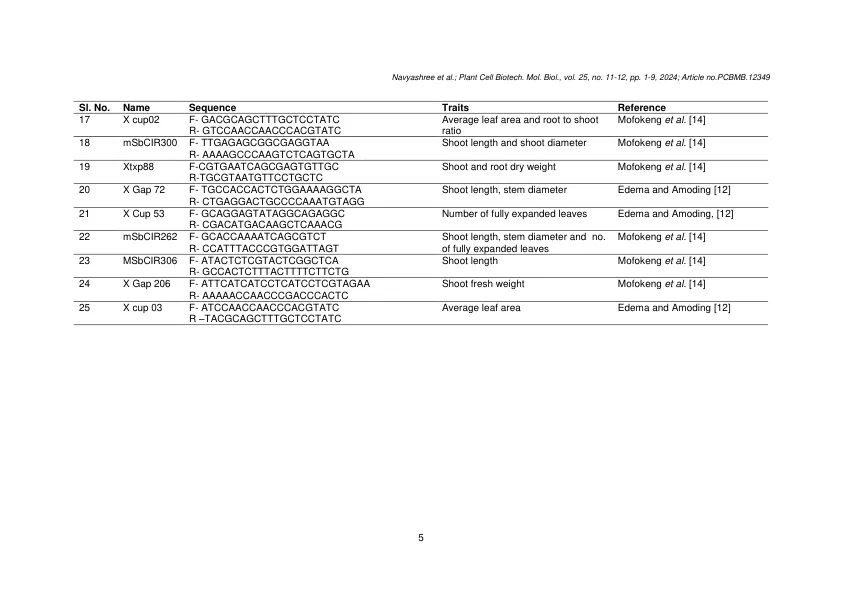
通过CTAB方法提取,这是一种获得高质量DNA的公认方案。量化DNA样品,以确保通过25 SSR引物扩增的一致模板浓度。PCR扩增,然后进行琼脂糖凝胶电泳,以分离和可视化SSR带模式,然后根据频带的存在或不存在二进制矩阵格式记录。在所使用的SSR引物中,有9个是多态性的,产生了13个可记分标记,突出了基因型之间的遗传变异性。跨SSR基因座的多态性信息含量(PIC)值,XTXP145基因座的最高PIC值为0.998,表明其在基因型之间区分的高歧视能力和信息性。遗传相似性指数,并通过使用算术平均值(UPGMA)方法对数据进行群集分析。所产生的树状图将基因型分为七个主要簇,以50%的相似性阈值分组,强调了所研究的高粱基因型中存在的遗传多样性。群集I包含单个基因型SVD-1272R,而群集II包括七个具有亚集群形成的基因型。群集III包括一个未分组的基因型SPV-486。群集IV包括八种基因型,而簇V,VI和VII均包含一个单一的未分组基因型。该树状图说明了高粱基因型之间的遗传多样性和关系,基于相似性指数。1。这项研究的结果证实了SSR标记在评估遗传多样性方面的功效,并强调了它们在旨在提高干旱耐受性的繁殖计划中的潜在效用。关键字:高粱; SSR;图片底漆;相似性指数。简介高粱[高粱双色(L.)Moench],被称为“小米之王”,其谷物尺寸较大,是Kharif(Rainy)和Rabi(Postrainy)季节种植的一种关键谷物作物。在印度,马哈拉施特拉邦,卡纳塔克邦和安得拉邦是产生高粱的主要国家,占国家产出的80%,占全球生产的约16%。尽管只有5%的高粱区域被灌溉,但毛毛高粱对于印度半岛的雨林地区至关重要。干旱应力对高粱的生理和生化过程产生负面影响,需要改善干旱耐受性特征,例如根生长,叶片发育和用水效率[2]。然而,由于谷物填充和圆锥花槽大小的应力程度相互作用,这些特征的表型选择是复杂的[3]。要应对这些挑战,评估高粱基因型的遗传多样性[4]和干旱耐受性至关重要[5]。栽培物种中的遗传多样性是提高作物生产率和质量以及发展耐药性品种的宝贵资源。分子标记物,尤其是简单的序列重复(SSR),为评估遗传变异和鉴定耐旱基因型提供了强大的方法[6]。SSR标记,由于其高可重现性和多重变化,对于基因组映射和标记 -
_____________________________________________________________________________________________________ *Corresponding author: E-mail: navyashreeshakti
主要关键词