机构名称:
¥ 1.0
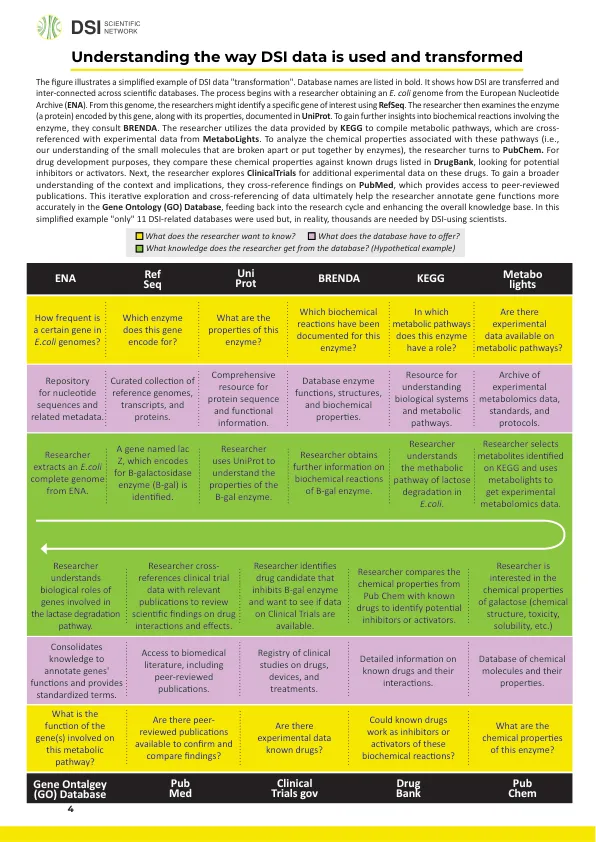
该图说明了DSI数据“转换”的简化示例。数据库名称以粗体列出。它显示了如何在科学数据库中转移和相互连接的DSI。该过程始于研究人员从欧洲核苷酸档案(ENA)中获得大肠杆菌基因组。从该基因组中,研究人员可能使用RefSeq确定了特定的感兴趣基因。然后,研究人员检查了该基因编码的酶(一种蛋白质),以及其特性,记录在Uniprot中。他们会进一步了解涉及酶的生化反应,他们咨询Brenda。研究人员利用KEGG提供的数据来编译代谢途径,这些数据与来自代谢的实验数据交叉引用。要分析与这些途径相关的化学特性(即,我们对酶破裂或将其放在一起的小分子的理解),研究人员转向Pubchem。出于药物开发的目的,它们将这些化学性质与在药品库中列出的已知药物,寻找潜在的抑制剂或激活剂进行了比较。接下来,研究人员探讨了有关这些药物的其他实验数据的临床试验。为了对上下文和含义有更广泛的了解,他们在PubMed上进行了交叉引用的发现,该发现提供了对同行评审出版物的访问。这种数据的迭代探索和交叉引用最终可帮助研究人员注释基因在基因本体论(GO)数据库中更准确地发挥作用,从而恢复了研究周期并增强了整体知识库。在这个简化的示例中,使用了11个与DSI相关的数据库,但实际上,使用DSI的科学家需要数千个。
映射DSI数据库的景观:
主要关键词