机构名称:
¥ 1.0
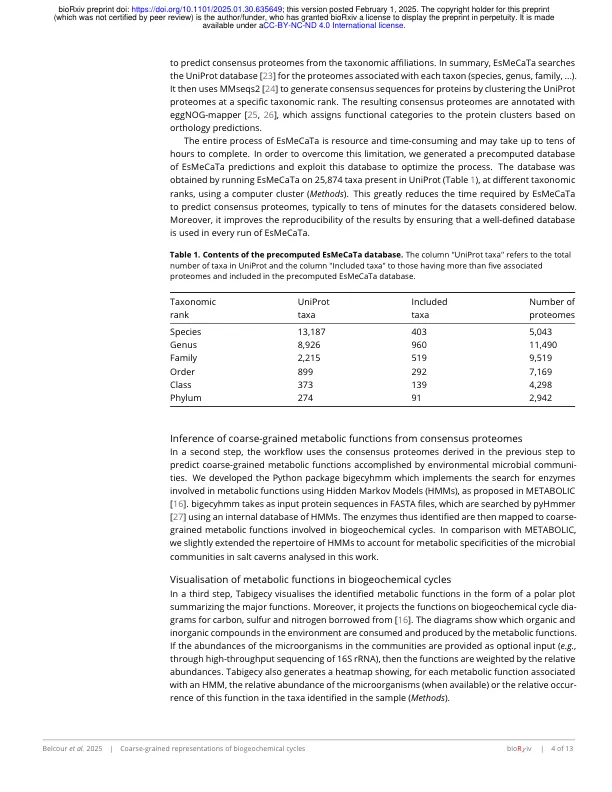
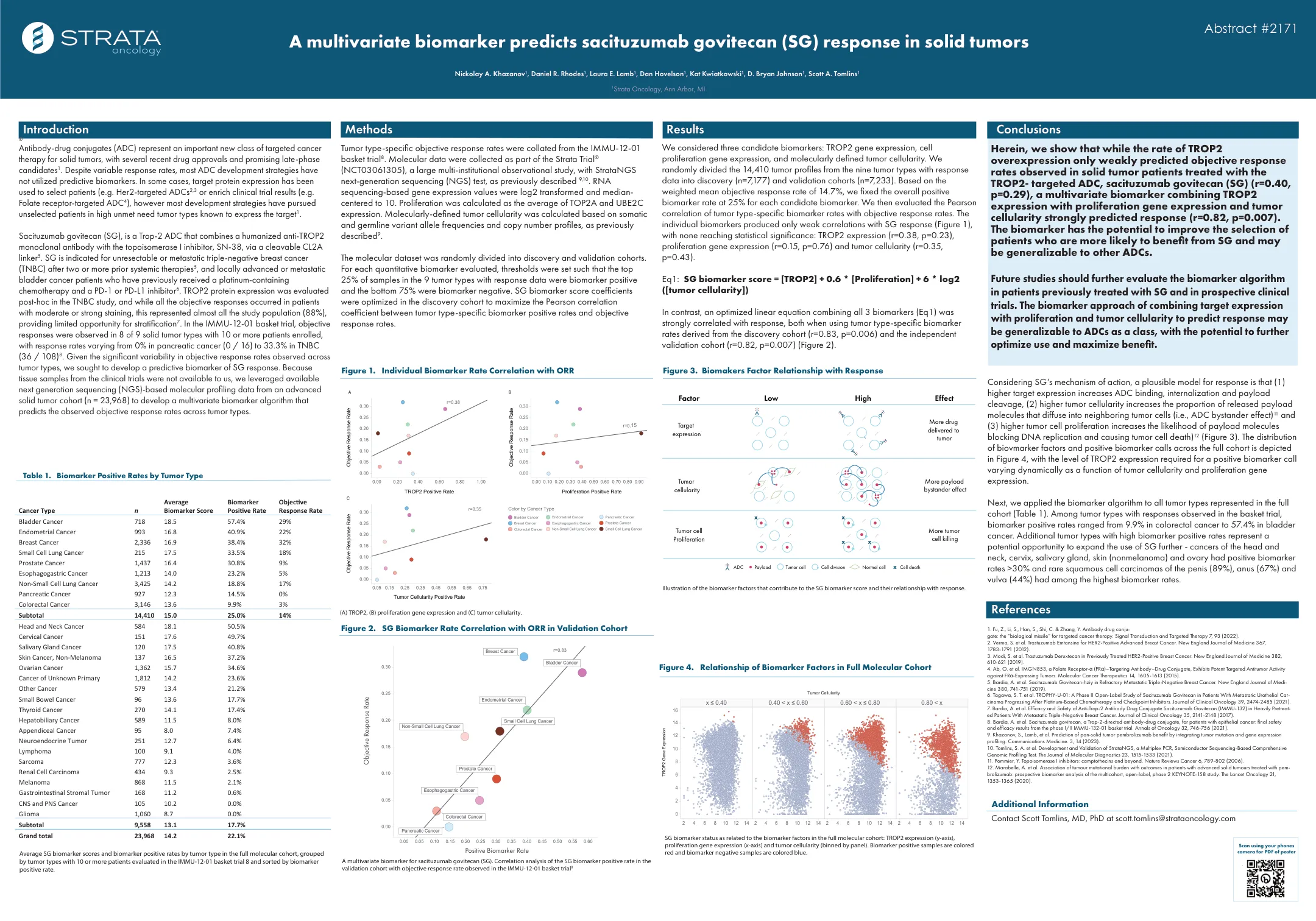
抽象动机:由于DNA测序的进步,现在常规地进行了环境微生物群落的分类学分析。确定这些群落在全球生物地球化学周期中的作用需要鉴定其代谢功能,例如氢氧化,还原和碳固定。这些功能可以直接从宏基因组学数据中推断出来,但是在许多环境应用中,MetabarCoding仍然是选择的方法。从元法编码数据及其整合到地球化学循环的粗粒表示中,代谢功能的重建仍然是当今有效的生物信息学问题。结果:我们开发了一条称为Tabigecy的管道,该管道利用分类学官员来预测构成生物地球化学周期的代谢功能。在第一个步骤中,Tabigecy使用该工具Esmecata从输入液位中预测共识蛋白质组。为了优化此过程,我们生成了一个预先计算的数据库,其中包含来自Uniprot的2,404个分类单元的信息。使用BigeCyhmm搜索了共有的蛋白质组织,BigeCyhmm是一个新开发的Python软件包,依靠隐藏的Markov模型来识别参与生物地球化学周期代谢功能的关键酶。然后将代谢功能投射到周期的粗粒表示上。我们将塔博基(Tabigecy)应用于两个盐洞数据集,并通过对样品进行的微生物活性和水力化学测量结果验证了其预测。结果突出了研究微生物群落对地理化学过程的影响的方法。关键字:微生物群落,生物地球化学周期,代谢功能,分类学官员
从跨编码数据中预测生物地球化学周期的粗粒表示
主要关键词