机构名称:
¥ 1.0
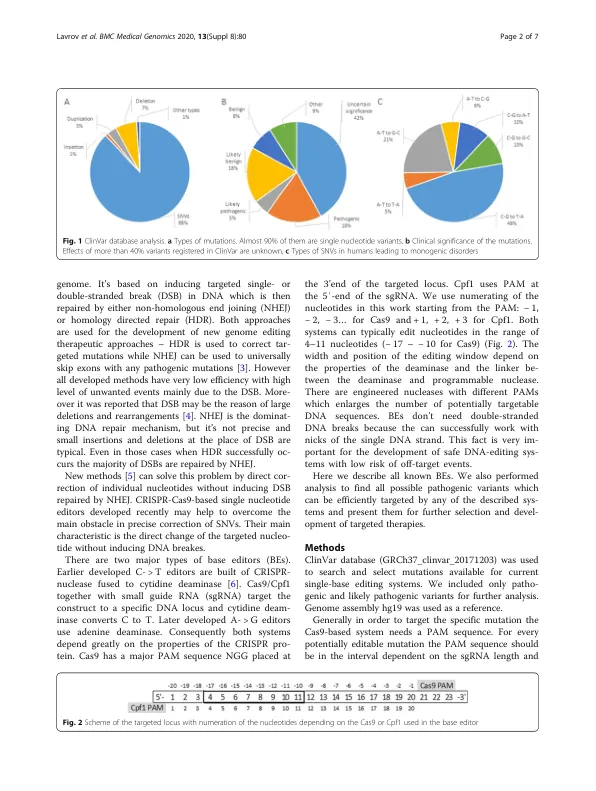
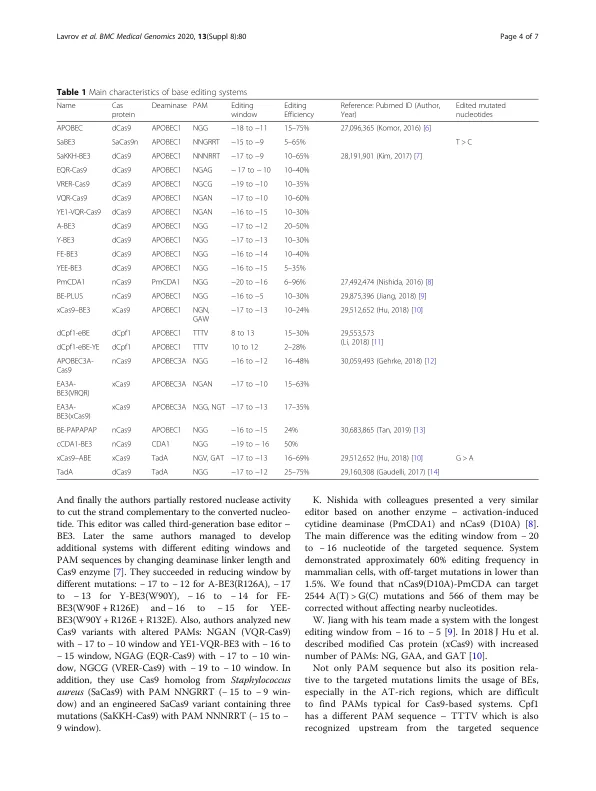
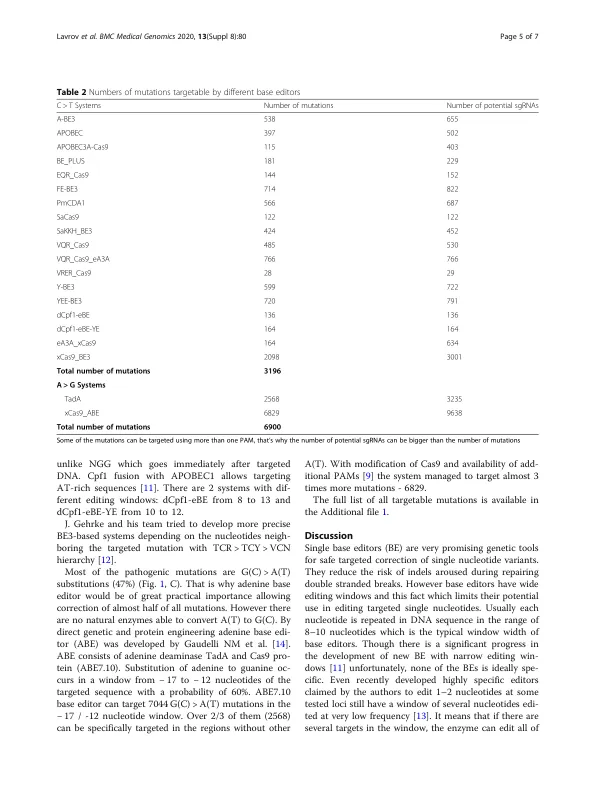
方法:使用 ClinVar 数据库 (GRCh37_clinvar_20171203) 搜索和选择可用于当前单碱基编辑系统的突变。我们仅纳入致病和可能致病的变异以供进一步分析。对于每一个可能可编辑的突变,我们检查 PAM 的存在。如果发现 PAM,我们会分析序列以找到只编辑一个核苷酸而不改变相邻核苷酸的可能性。用于搜索 Clinvar 数据库和分析序列的脚本代码是用 R 编写的,可在附录中找到。结果:我们分析了目前在 9 篇出版物中报道的 21 个编辑系统。每个系统都有不同的工作特性,例如编辑窗口和 PAM 序列。C > T 碱基编辑器可以精确定位 3196 个突变(占所有致病 T > C 变异的 46%),A > G 编辑器可以精确定位 6900 个突变(占所有致病 G > A 变异的 34%)。
针对单碱基编辑的致病变异的基因组规模分析