机构名称:
¥ 2.0
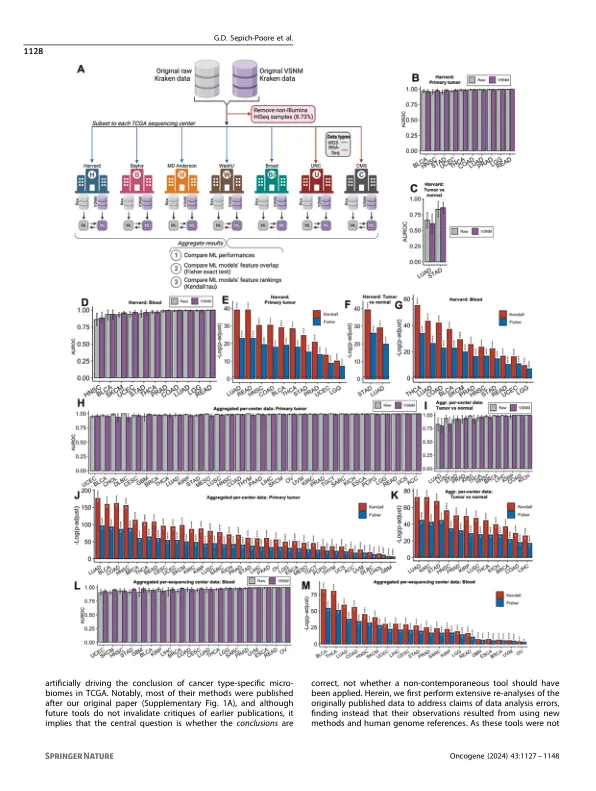
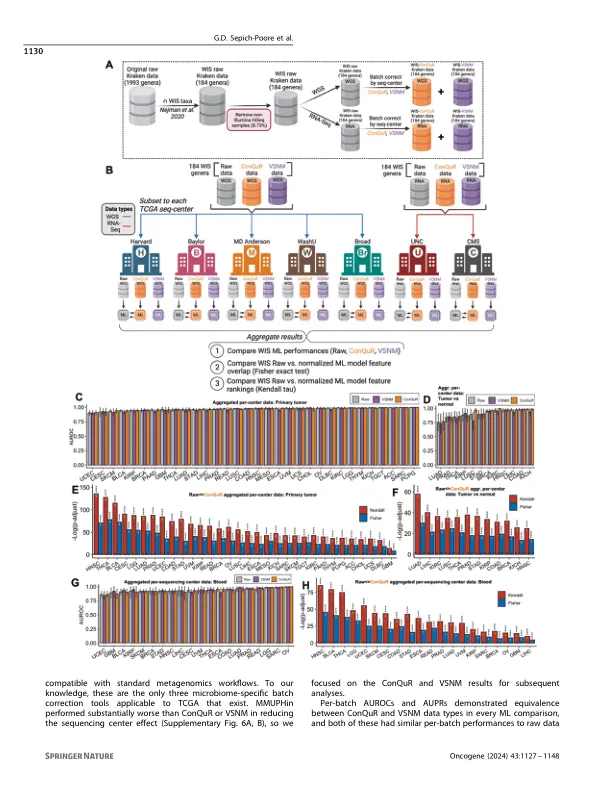
在2020年,我们鉴定了癌症基因组图集(TCGA)中的癌症特异性微生物信号[1]。多个同行评审的论文独立验证或扩展了我们的发现[2-12]。鉴于这种影响,我们仔细考虑了Gihawi等人的关注。[13]批处理校正和数据库污染具有人工的宿主序列,从而产生了癌症类型特异性微生物组的外观。(1)我们通过比较了每批次的原始和VOOM-SNM校正数据,测试了批处理校正,发现了预测等效性和显着性相似的功能。我们发现了使用现代微生物组特异性方法(Conqur [14])的一致结果,并且在限制在独立的,高度污染的队列中发现的分类单元时。(2)使用conterminator [15],我们发现原始数据库中的人类污染水平较低(基因组的约1%)。我们证明了Gihawi等人对人类读物的发现增加。[13]是由于使用了较新的人类基因组参考。(3)我们开发了详尽的方法,这是清洁repseq的敏感性两倍的方法。我们全面拥有许多人类(PAN)基因组参考的宿主数据。我们对此重复了所有分析和Gihawi等。[13]管道,发现癌症类型的微生物组。这些广泛的重新分析和更新的方法验证了我们最初的结论,即TCGA中存在癌症类型的微生物特异性标志,并表明它们对方法论很强。
癌症微生物组信号的鲁棒性在广泛的方法学变化上
主要关键词