机构名称:
¥ 2.0
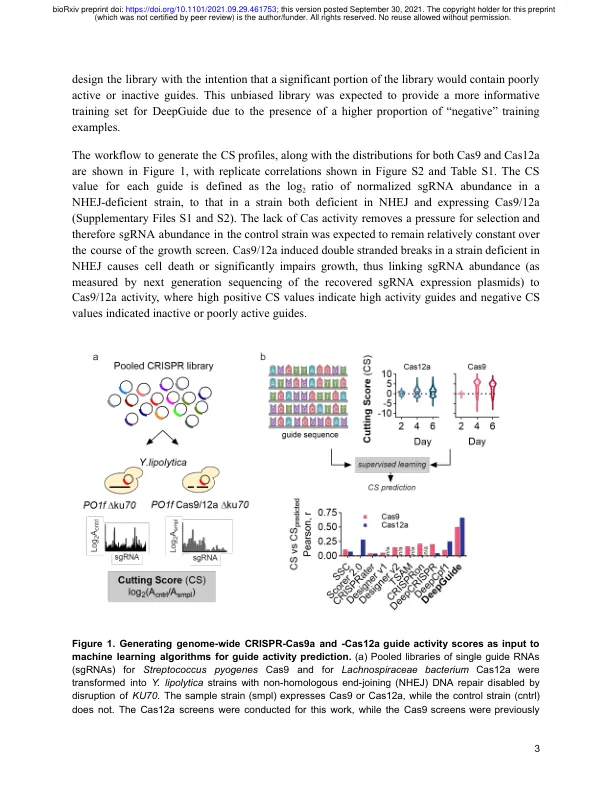
摘要 全基因组功能性遗传筛选已成功发现基因型-表型关系并设计新表型。虽然广泛应用于哺乳动物细胞系和大肠杆菌,但在非常规微生物中的使用受到限制,部分原因是无法准确设计此类物种的高活性 CRISPR 向导。在这里,我们开发了一种针对所选生物体(在本例中为产油酵母解脂耶氏酵母)的 sgRNA 设计实验计算方法。在不存在非同源末端连接(主要的 DNA 修复机制)的情况下进行负选择筛选,用于生成 SpCas9 和 LbCas12a 的单个向导 RNA (sgRNA) 活性谱。这种全基因组数据作为深度学习算法 DeepGuide 的输入,该算法能够准确预测向导活性。 DeepGuide 使用无监督学习来获取基因组的压缩表示,然后通过监督学习来映射具有指导活性的 sgRNA 序列、基因组背景和表观遗传特征。全基因组和选定基因子集的实验验证证实了 DeepGuide 能够准确预测高活性 sgRNA。DeepGuide 提供了一种生物体特异性的 CRISPR 指导活性预测因子,可广泛应用于真菌物种、原核生物和其他非常规生物。
Cas12a 在解脂耶氏酵母中起指导作用
主要关键词