机构名称:
¥ 3.0
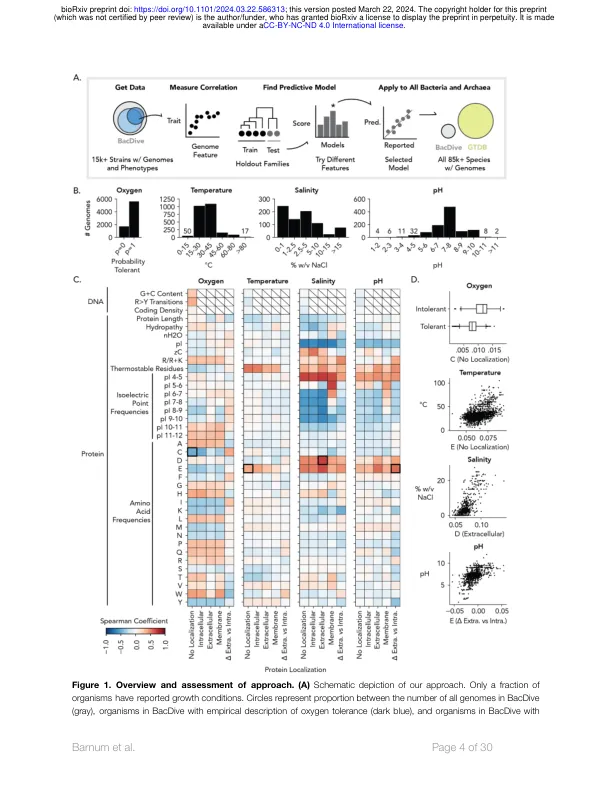
在实验室中生长微生物的能力可以使其遗传学的可重复研究和工程化。不幸的是,由于识别培养条件所需的努力,生命树中的大多数微生物仍然没有耕种。对指导实验测试的可行生长条件的预测将是非常可取的。虽然可以通过注释的基因在计算上预测碳和能源,但很难预测其他生长的要求,例如氧,温度,盐度和pH。在这里,我们开发了基于基因组的计算模型,能够预测氧耐受性(92%平衡精度),最佳温度(r 2 = 0.73),盐度(r 2 = 0.81)和pH(r 2 = 0.48),而新的分类微生物家族无需功能基因注释。使用15,596种细菌和古细菌的生长条件和基因组序列,我们发现氨基酸频率可预测生长需求。只有两个氨基酸可以预测氧气耐受性,其精度为88%。使用蛋白质的细胞定位来计算氨基酸频率改善了pH的预测(r 2增加0.36)。由于这些模型不依赖于特定基因的存在或不存在,因此可以将它们应用于不完整的基因组中,仅需要10%的完整性。我们应用模型来预测所有85,205种测序细菌和古细菌的增长需求,发现未养殖物种富含嗜热,厌氧菌和嗜酸菌。这项工作指导了对不同微生物实验室种植的生长限制的识别。最后,我们将模型应用于具有元基因组组装的基因组的3,349个环境样品,并表明社区中的个别微生物具有不同的增长需求。
预测氨基酸组成的微生物生长条件
主要关键词