机构名称:
¥ 1.0
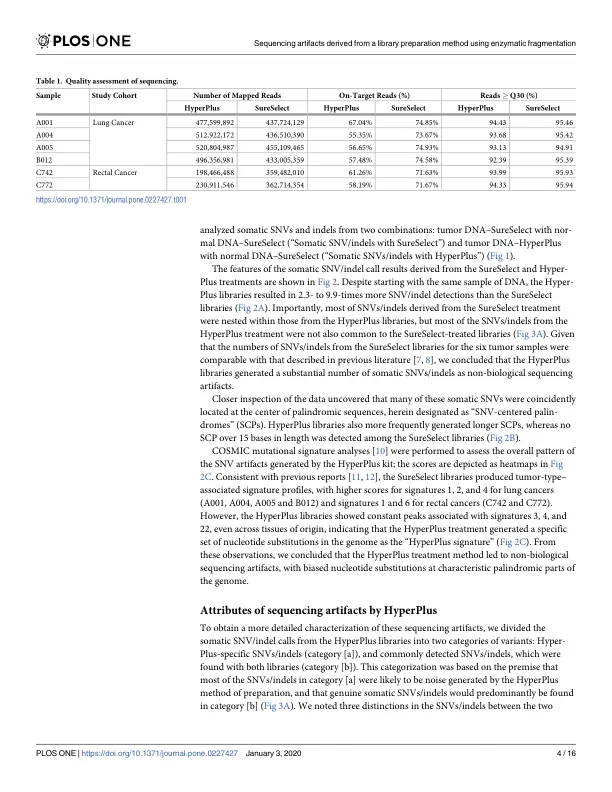
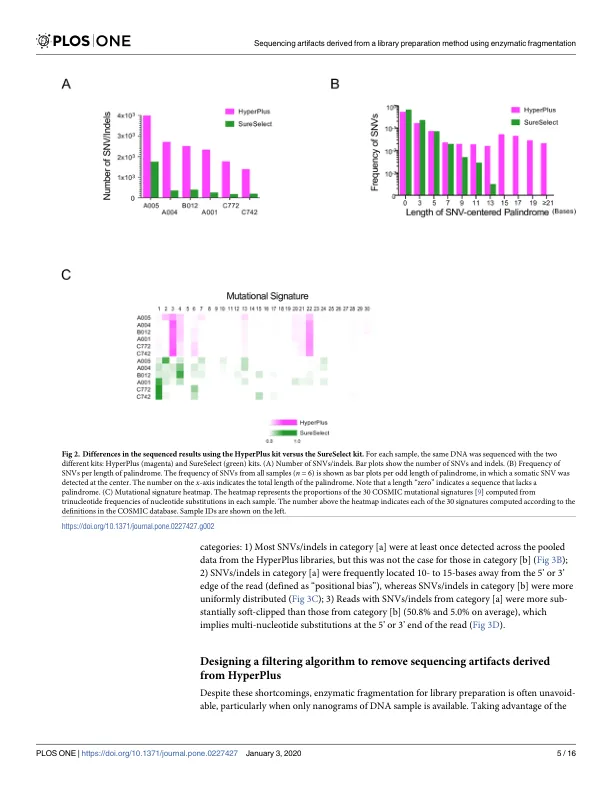
DNA 片段化是基于杂交捕获的短读测序中文库制备过程中的一个基本步骤。迄今为止,人们一直使用超声波来制备适当大小的 DNA,但这种方法会导致大量 DNA 样本损失。最近,研究采用了依赖于 DNA 内切酶酶促片段化的文库制备方法来最大限度地减少 DNA 损失,尤其是在纳米量样本中。然而,尽管它们被广泛使用,但酶促片段化对所得序列的影响尚未得到仔细评估。在这里,我们对使用超声波和酶促片段化方法制备的相同肿瘤 DNA 样本的体细胞变异进行了成对比较。我们的分析显示,与通过超声波创建的文库相比,内切酶处理的文库中反复出现的人工 SNV/indel 数量要多得多。这些人工制品以基因组背景下的回文结构、测序读取中的位置偏差和多核苷酸替换为标志。利用这些独特的特性,我们开发了一种过滤算法,可以高特异性和灵敏度地区分真正的体细胞突变和人为噪声。噪声消除恢复了肿瘤样本中突变特征的组成。因此,我们提供了一种信息学算法来解决因内切酶介导的碎片化而产生的测序错误,这是本研究中首次强调的。
采用酶促碎裂法制备文库所得的测序产物
主要关键词