机构名称:
¥ 3.0
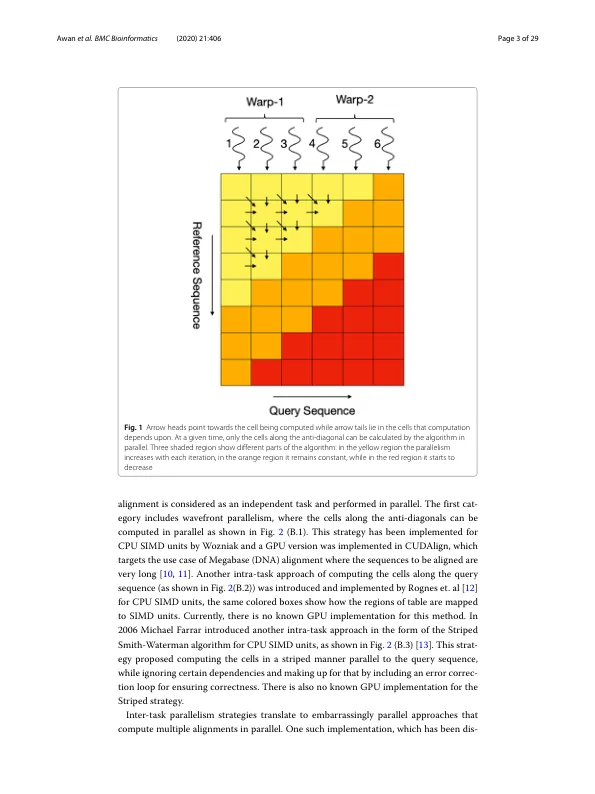
摘要背景:生物信息学工作流程经常使用自动基因组组装和蛋白质聚类工具。在大多数这些工具的核心中,执行时间的很大一部分用于确定两个序列之间的最佳局部比对。此任务使用 Smith-Waterman 算法执行,这是一种基于动态规划的方法。随着现代测序技术的出现以及基因组和蛋白质数据库的规模不断扩大,对更快的 Smith-Waterman 实现的需求应运而生。CPU 提供了多种 Smith-Waterman 算法的 SIMD 策略。然而,随着 HPC 设施向基于加速器的架构的转变,对高效的 GPU 加速策略的需求也随之而来。现有的基于 GPU 的策略要么针对特定类型的字符(核苷酸或氨基酸)进行了优化,要么仅针对少数应用用例进行了优化。结果:在本文中,我们提出了一种新的 GPU 架构序列比对策略 ADEPT,它与领域无关,支持来自基因组和蛋白质的序列比对。我们提出的策略使用 GPU 特定的优化,不依赖于序列的性质。我们通过实施 Smith-Waterman 算法并将其与类似的 CPU 策略以及每个领域中已知的最快 GPU 方法进行比较,证明了该策略的可行性。ADEPT 的驱动程序使其能够跨多个 GPU 进行扩展,并可以轻松集成到利用大规模计算系统的软件管道中。我们已经证明,基于 ADEPT 的 Smith-Waterman 算法在 Cori 超级计算机的单个 GPU 节点(8 个 GPU)上分别针对基于蛋白质和基于 DNA 的数据集展示了 360 GCUPS 和 497 GCUP 的峰值性能。总体而言,与相应的 SIMD CPU 实现相比,ADEPT 在节点到节点的比较中显示出 10 倍更快的性能。结论:ADEPT 表现出相当或更好的性能(下页继续)
ADEPT:一种适用于 GPU 架构的域独立序列比对策略
主要关键词