机构名称:
¥ 2.0
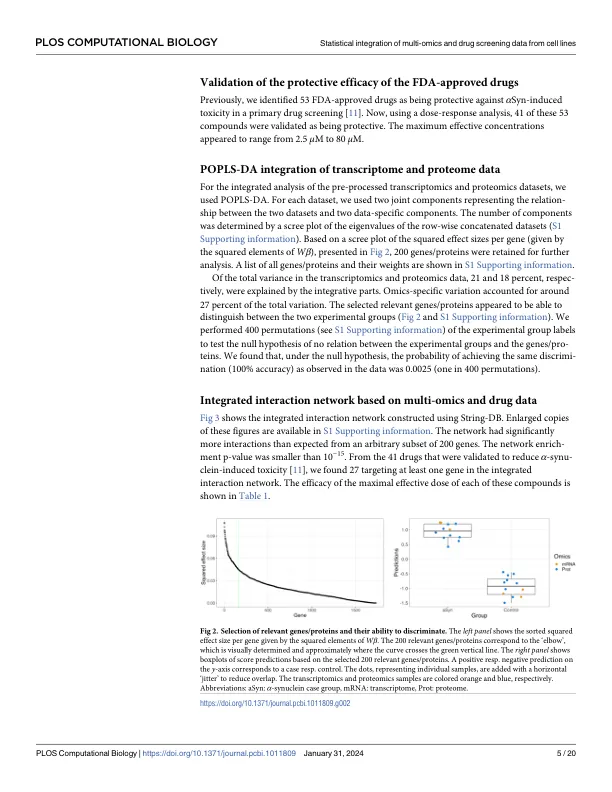
数据集成方法用于获得多个数据集的统一摘要。对于多模态数据,我们提出了一种计算工作流程来联合分析来自细胞系的数据集。该工作流程包括一种用于多组学数据的新型概率数据集成方法,称为 POPLS-DA。该工作流程的动机是对突触核蛋白病的研究,其中在受影响的 LUHMES 细胞系和对照中测量转录组学、蛋白质组学和药物筛选数据。目的是突出显示与突触核蛋白病有关的潜在可用药途径和基因。首先,使用 POPLS-DA 优先考虑最能区分病例和对照的基因和蛋白质。对于这些基因,构建了一个集成的相互作用网络,其中结合了药物筛选数据以突出显示网络中的可用药基因和途径。最后,进行功能富集分析以识别保护性药物靶向的突触和溶酶体相关基因和蛋白质簇。将 POPLS-DA 与其他单组学和多组学方法进行了比较。我们发现,热休克蛋白 70 家族成员 HSPA5 是经过验证的药物(尤其是 AT1 阻滞剂)最常针对的基因之一。HSPA5 和 AT1 阻滞剂之前已被证实与 α-突触核蛋白病理和帕金森病有关,这表明我们的发现具有相关性。我们的计算工作流程确定了治疗突触核蛋白病的新方向。与其他单组学和多组学方法相比,POPLS-DA 提供了更大的可解释基因集。基于 R 和 markdown 的实现可在线免费获取。
来自细胞系的多组学和药物筛选数据的统计整合
主要关键词