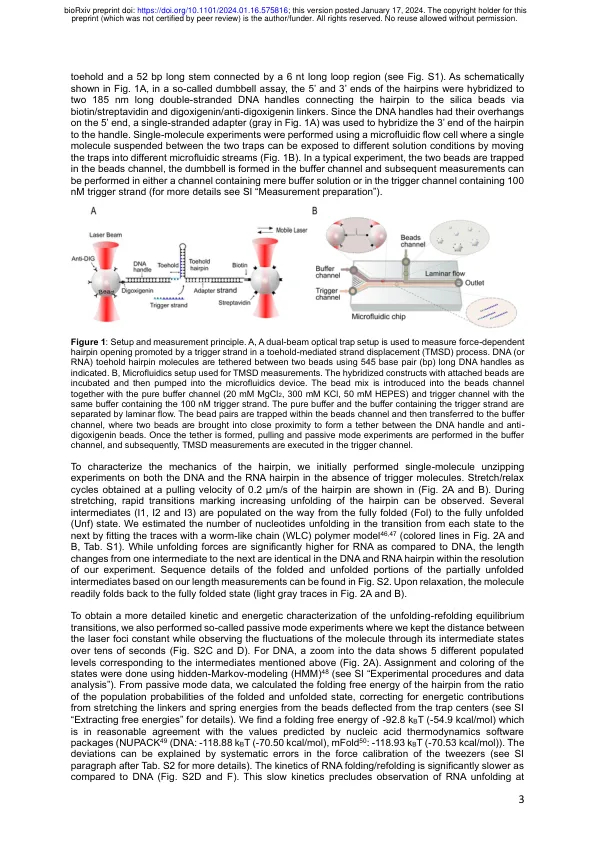
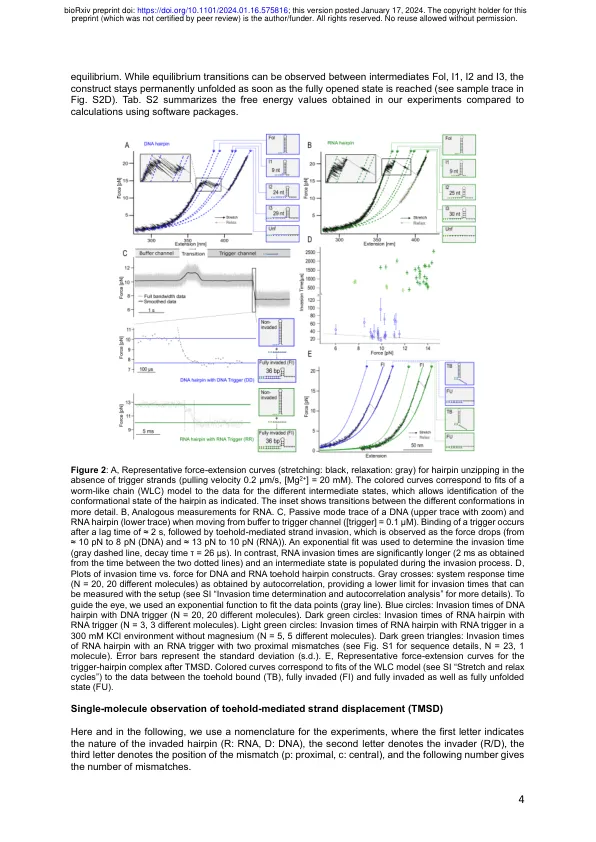
toehold介导的链位移的单分子力光谱Andreas Walbrun 1,*,Tianhe Wang 2,*,Michael Matthies 2,Petršulc2,3,Friedrich C. Simmel 2,+ Matthias Rief,Matthias Rief 1慕尼黑技术大学生物科学系综合蛋白质科学中心(CPA),Ernst-Otto-Fischer-STR。8,85748德国Garching。 电子邮件:matthias.rief@mytum.de 2。 慕尼黑技术大学,TUM自然科学学院,生物科学系,AM COULOMBWALL 4A,85748 GARCHING,德国。 电子邮件:simmel@tum.de 3。 亚利桑那州立大学生物设计学院的分子科学和分子设计与生物仪中心,美国亚利桑那州南卡利斯特大街1001号,美国亚利桑那州坦佩市85281,美国 *这些作者同样贡献:安德烈亚斯·沃尔布伦(Andreas Walbrun) (TMSD)在动态DNA纳米技术中广泛使用,并且是多种基于DNA或RNA的反应电路的基础。 以前的研究通常依赖于散装荧光测量值来研究TMSD的动力学,该动力学仅提供有效的,散装平均的反应速率,并且无法在单个分子甚至碱基对的水平上解决该过程。 在这项工作中,我们使用单分子力光谱(SMF)探索单分子水平的链位移过程的动力学,并具有由最先进的粗粒元模拟支持的光学陷阱。 此外,我们使用力研究了DNA入侵RNA的动力学,这一过程很少发生力。8,85748德国Garching。电子邮件:matthias.rief@mytum.de 2。慕尼黑技术大学,TUM自然科学学院,生物科学系,AM COULOMBWALL 4A,85748 GARCHING,德国。电子邮件:simmel@tum.de 3。亚利桑那州立大学生物设计学院的分子科学和分子设计与生物仪中心,美国亚利桑那州南卡利斯特大街1001号,美国亚利桑那州坦佩市85281,美国 *这些作者同样贡献:安德烈亚斯·沃尔布伦(Andreas Walbrun) (TMSD)在动态DNA纳米技术中广泛使用,并且是多种基于DNA或RNA的反应电路的基础。 以前的研究通常依赖于散装荧光测量值来研究TMSD的动力学,该动力学仅提供有效的,散装平均的反应速率,并且无法在单个分子甚至碱基对的水平上解决该过程。 在这项工作中,我们使用单分子力光谱(SMF)探索单分子水平的链位移过程的动力学,并具有由最先进的粗粒元模拟支持的光学陷阱。 此外,我们使用力研究了DNA入侵RNA的动力学,这一过程很少发生力。亚利桑那州立大学生物设计学院的分子科学和分子设计与生物仪中心,美国亚利桑那州南卡利斯特大街1001号,美国亚利桑那州坦佩市85281,美国 *这些作者同样贡献:安德烈亚斯·沃尔布伦(Andreas Walbrun) (TMSD)在动态DNA纳米技术中广泛使用,并且是多种基于DNA或RNA的反应电路的基础。以前的研究通常依赖于散装荧光测量值来研究TMSD的动力学,该动力学仅提供有效的,散装平均的反应速率,并且无法在单个分子甚至碱基对的水平上解决该过程。在这项工作中,我们使用单分子力光谱(SMF)探索单分子水平的链位移过程的动力学,并具有由最先进的粗粒元模拟支持的光学陷阱。此外,我们使用力研究了DNA入侵RNA的动力学,这一过程很少发生力。通过探测toehold结构的发夹的末端,我们可以通过微秒和纳米分辨率实时触发和观察TMSD。使用微流体测定法,我们将发夹暴露于触发链的溶液中,我们发现在负载下,TMSD的进行非常迅速,单步时间为1 µs。将不匹配引入入侵者序列使我们能够调节稳定性,以使入侵和重新染色在均衡中也发生,即使在负载下也是如此。这使我们能够在单个分子上研究数千个入侵/入侵事件,并分析入侵过程的动力学。将我们的发现推送到零载荷,我们发现DNA入侵DNA的单步速度比入侵RNA快的速度快四倍。我们的结果揭示了序列效应对TMSD过程的重要性,并且对于核酸纳米技术和合成生物学的广泛应用至关重要。关键字:肋骨调节器,脚趾介导的链位移,分支迁移,单分子力光谱
toehold介导的链位移的单分子力光谱
主要关键词