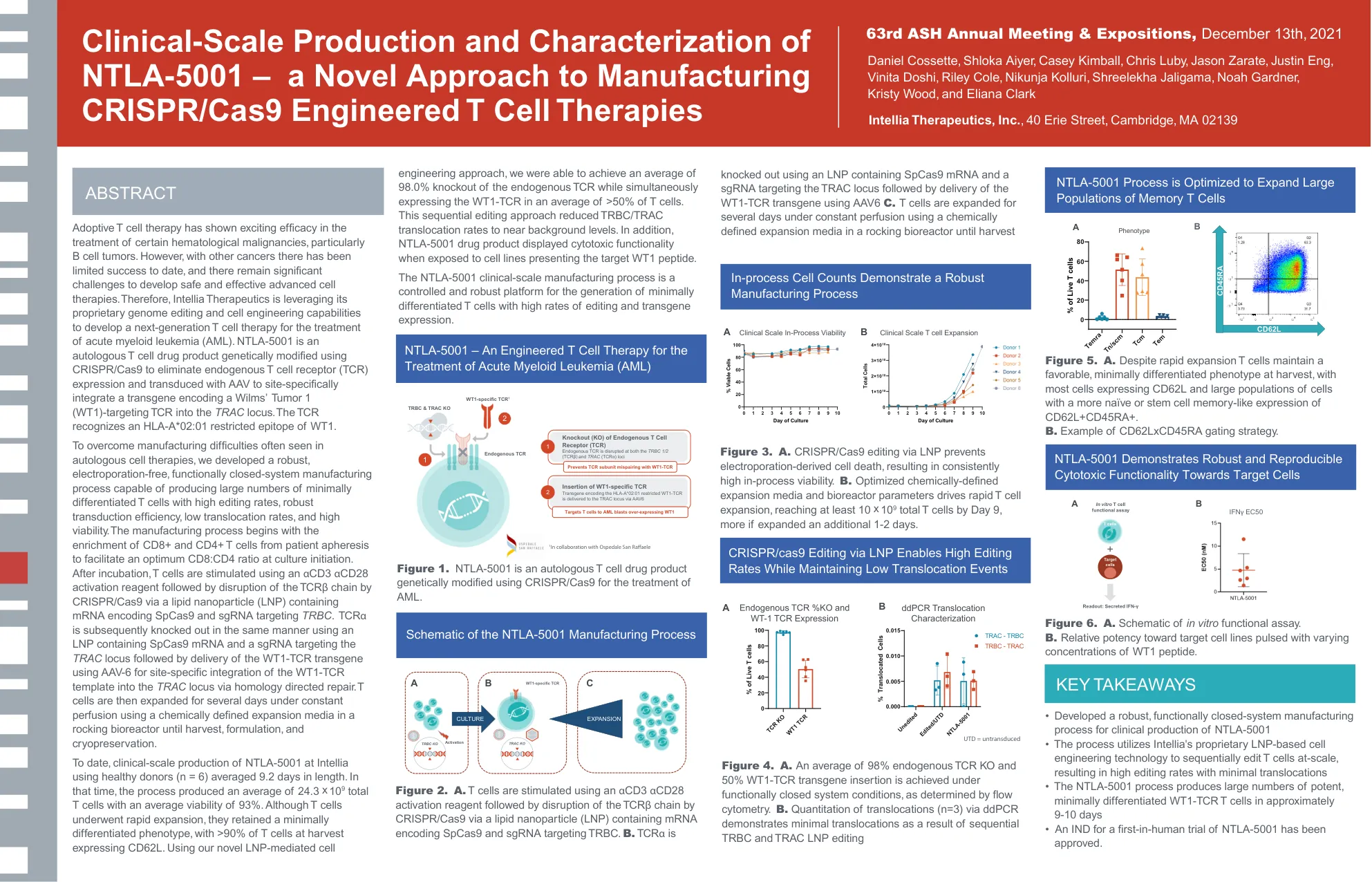
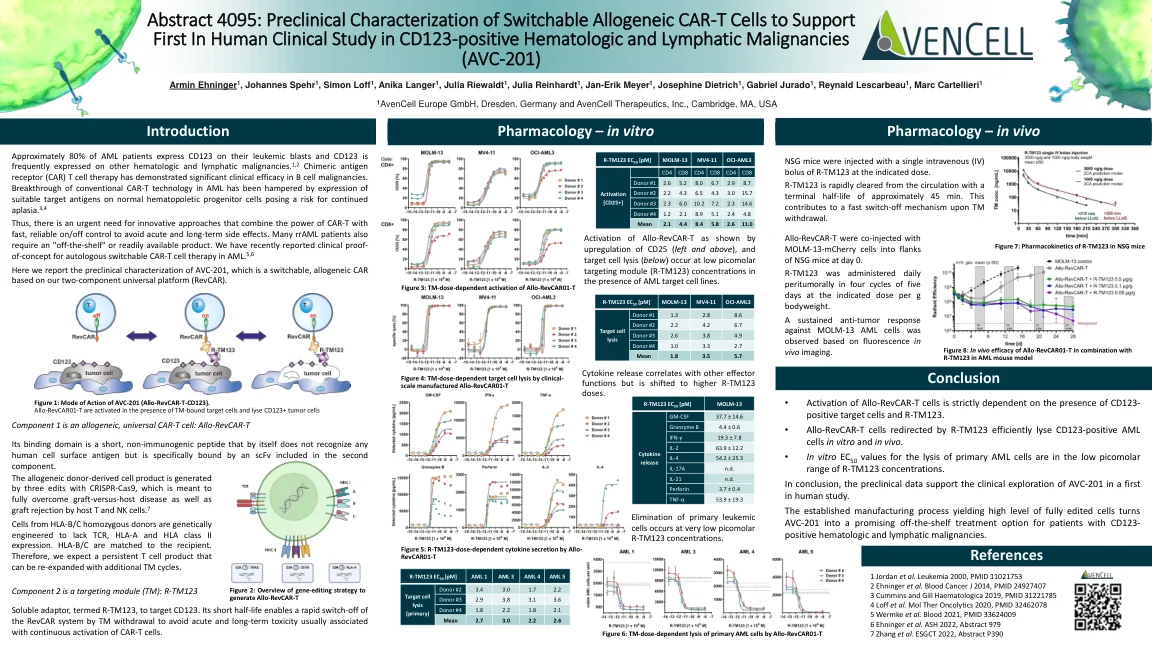
机构名称:
¥ 1.0
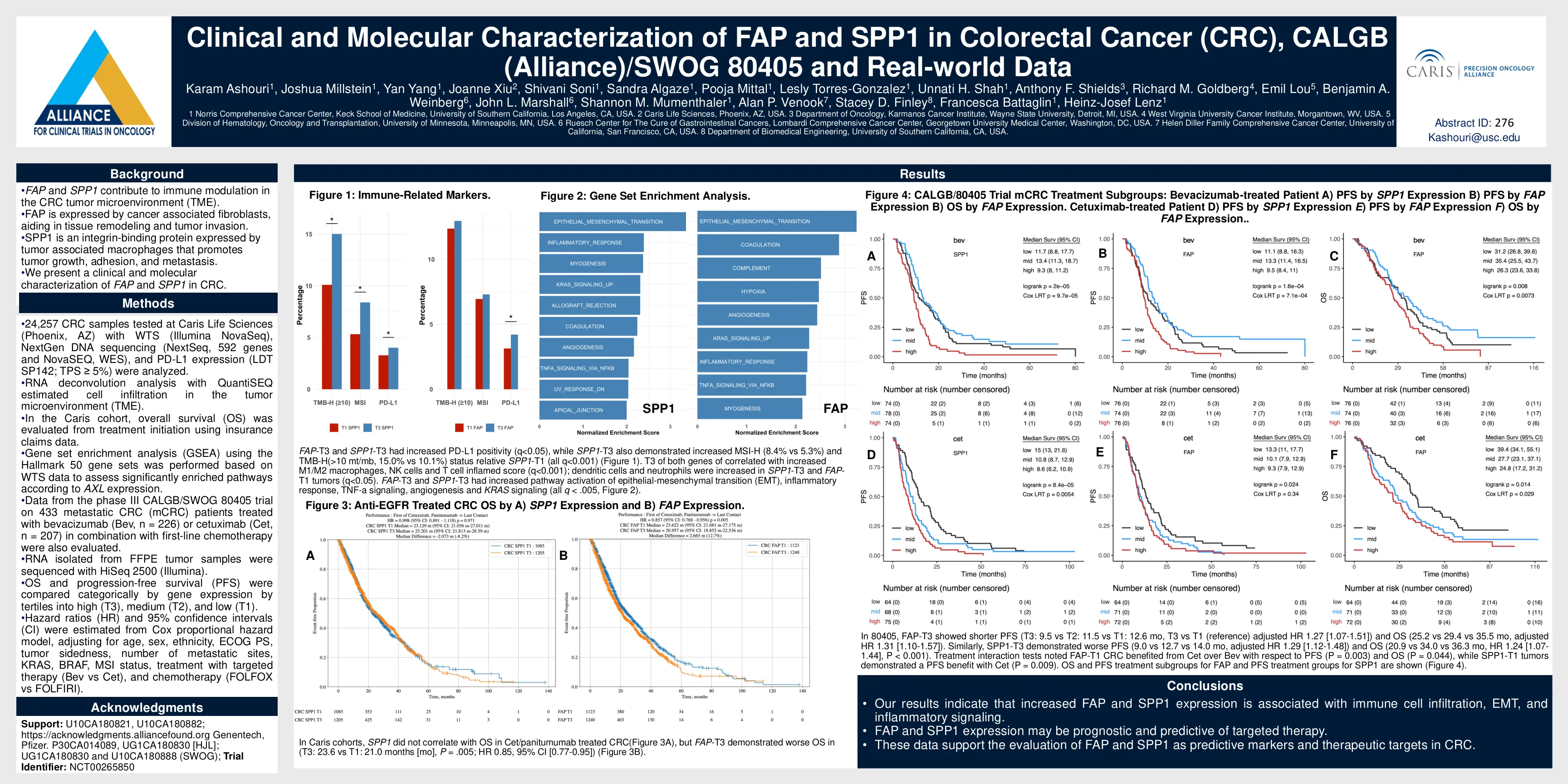
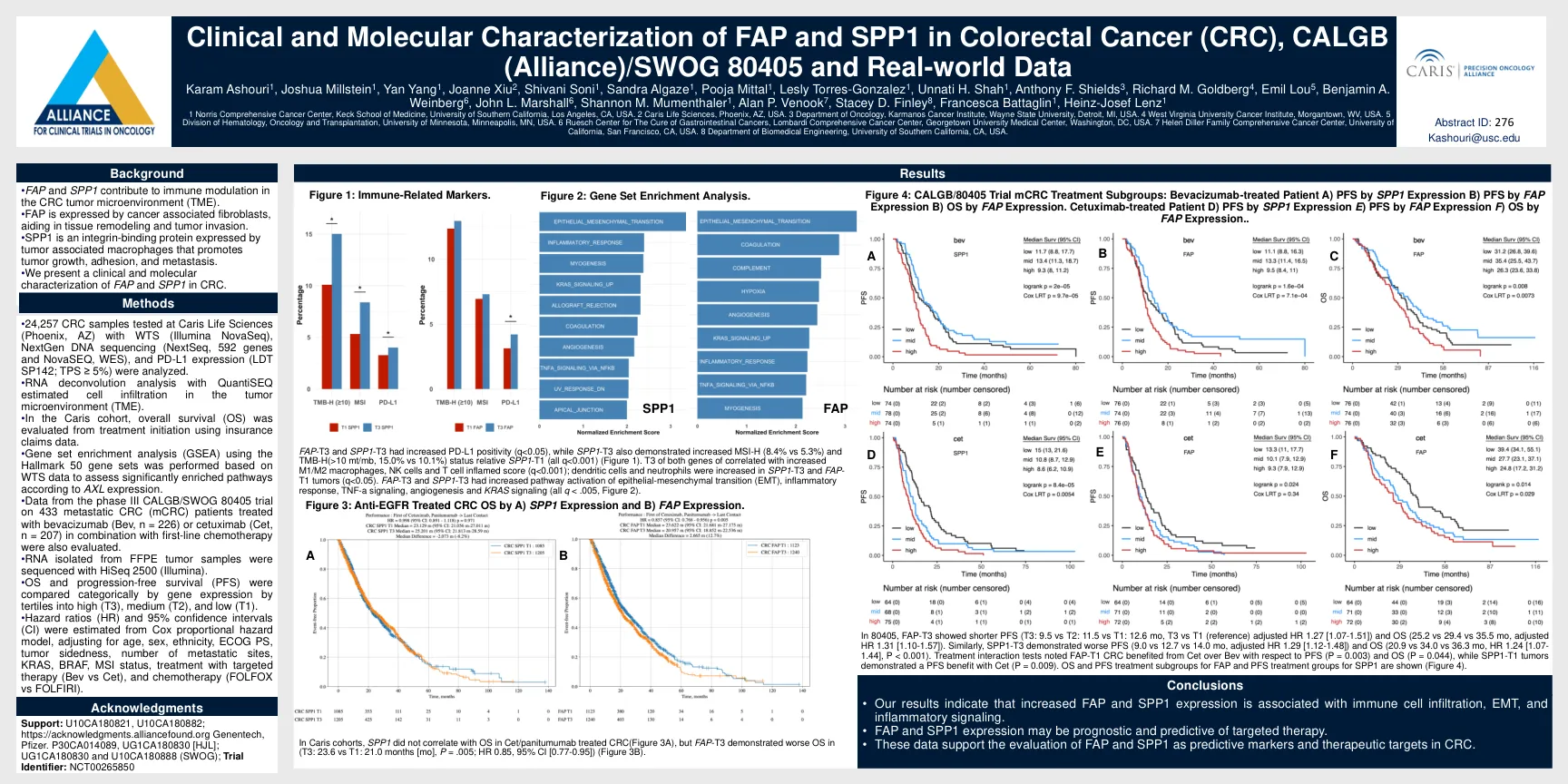
• 分析了 Caris Life Sciences(亚利桑那州凤凰城)使用 WTS(Illumina NovaSeq)、NextGen DNA 测序(NextSeq、592 个基因和 NovaSEQ、WES)和 PD-L1 表达(LDT SP142;TPS ≥ 5%)测试的 24,257 个 CRC 样本。• 使用 QuantiSEQ 进行 RNA 反卷积分析估计肿瘤微环境 (TME) 中的细胞浸润。• 在 Caris 队列中,使用保险索赔数据从治疗开始评估总体生存率 (OS)。• 根据 WTS 数据使用 Hallmark 50 基因组进行基因集富集分析 (GSEA),以根据 AXL 表达评估显著富集的通路。 • 还评估了 III 期 CALGB/SWOG 80405 试验的数据,该试验涉及 433 名接受贝伐单抗 (Bev,n = 226) 或西妥昔单抗 (Cet,n = 207) 联合一线化疗治疗的转移性 CRC (mCRC) 患者。 • 使用 HiSeq 2500 (Illumina) 对从 FFPE 肿瘤样本中分离的 RNA 进行测序。 • 根据基因表达将 OS 和无进展生存期 (PFS) 分为高 (T3)、中 (T2) 和低 (T1) 三分位数进行分类比较。 • 根据 Cox 比例风险模型估计风险比 (HR) 和 95% 置信区间 (CI),并根据年龄、性别、种族、ECOG PS、肿瘤单侧性、转移部位数量、KRAS、BRAF、MSI 状态、靶向治疗 (Bev vs Cet) 和化疗 (FOLFOX vs FOLFIRI) 进行调整。
FAP 和 SPP1 的临床和分子表征...
主要关键词