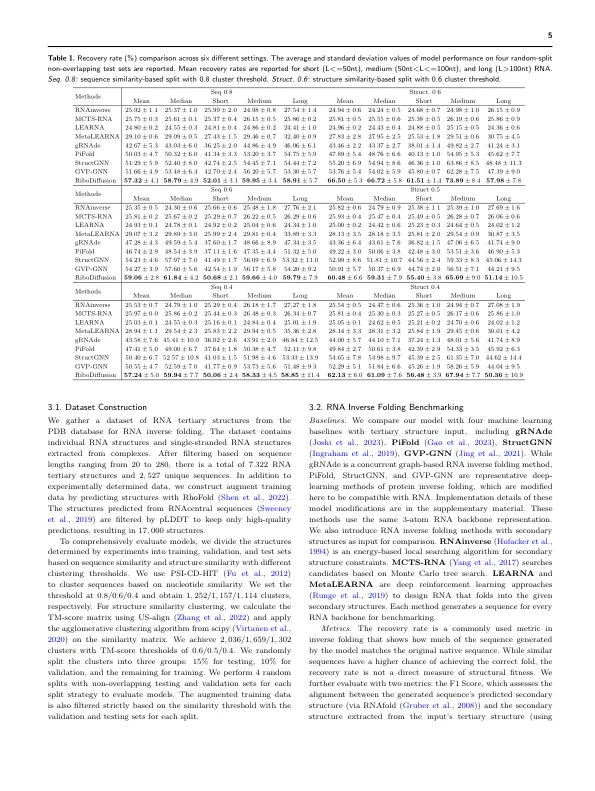
RNA设计显示了RNA在各种生物过程中的关键作用驱动的合成生物学和治疗剂中越来越多的应用。 一个基本的挑战是找到满足结构约束的功能性RNA序列,称为反折叠问题。 已经出现了基于二级结构的计算方法来解决此问题。 然而,由于数据的稀缺,非唯一的结构序列映射和RNA构象的灵活性,直接从3D结构设计RNA序列仍然具有挑战性。 在这项研究中,我们提出了RECODI↵一种用于RNA逆折叠的生成二次模型,可以学习给定3D主链结构的RNA序列的条件分布。 我们的模型由基于图神经网络的结构模块和基于变压器的序列模块组成,该模块将随机序列转换为所需的序列。 通过调整采样重量,我们的模型允许序列恢复和多样性之间进行交易,以探索更多的候选者。 我们将基于RNA聚类的测试集使用DI↵Cut-O↵S序列或结构相似性。 我们的模型在序列恢复中的表现优于基准,序列相似性分裂的平均相对改善为11%,结构相似性分裂的平均相对提高为16%。 此外,Ribodi↵在各种RNA长度类别和RNA类型中的表现始终如一。 我们还施加了内部折叠,以验证生成的序列是否可以折叠到给定的3D RNA骨架中。RNA设计显示了RNA在各种生物过程中的关键作用驱动的合成生物学和治疗剂中越来越多的应用。一个基本的挑战是找到满足结构约束的功能性RNA序列,称为反折叠问题。已经出现了基于二级结构的计算方法来解决此问题。然而,由于数据的稀缺,非唯一的结构序列映射和RNA构象的灵活性,直接从3D结构设计RNA序列仍然具有挑战性。在这项研究中,我们提出了RECODI↵一种用于RNA逆折叠的生成二次模型,可以学习给定3D主链结构的RNA序列的条件分布。我们的模型由基于图神经网络的结构模块和基于变压器的序列模块组成,该模块将随机序列转换为所需的序列。通过调整采样重量,我们的模型允许序列恢复和多样性之间进行交易,以探索更多的候选者。我们将基于RNA聚类的测试集使用DI↵Cut-O↵S序列或结构相似性。我们的模型在序列恢复中的表现优于基准,序列相似性分裂的平均相对改善为11%,结构相似性分裂的平均相对提高为16%。此外,Ribodi↵在各种RNA长度类别和RNA类型中的表现始终如一。我们还施加了内部折叠,以验证生成的序列是否可以折叠到给定的3D RNA骨架中。我们的方法可能是RNA设计的强大工具,可以探索庞大的序列空间并为3D结构约束发现新颖的解决方案。
ribodifusion:基于第三结构的RNA逆折叠与生成扩散模型
主要关键词