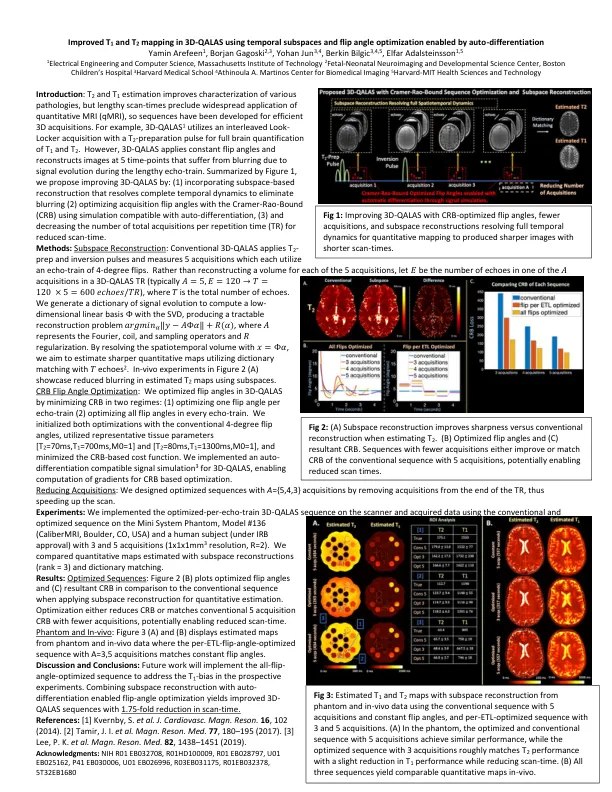
简介:T 2 和 T 1 估计可改善各种病理的特征描述,但较长的扫描时间阻碍了定量 MRI (qMRI) 的广泛应用,因此已经开发了序列以实现高效的 3D 采集。例如,3D-QALAS 1 利用交错的 Look-Locker 采集和 T 2 准备脉冲来对 T 1 和 T 2 进行全脑量化。但是,3D-QALAS 应用恒定翻转角并在 5 个时间点重建图像,这些时间点由于冗长的回波序列期间的信号演变而出现模糊。总结图 1,我们建议通过以下方式改进 3D-QALAS:(1) 结合基于子空间的重建来解决完整的时间动态以消除模糊 (2) 使用与自动微分兼容的模拟通过 Cramer-Rao 界限 (CRB) 优化采集翻转角,(3) 并减少每重复时间 (TR) 的总采集次数以缩短扫描时间。方法:子空间重建:传统 3D-QALAS 应用 T 2 准备和反转脉冲并测量 5 次采集,每次采集都利用 4 度翻转的回声序列。不是为 5 次采集中的每次采集重建一个体积,而是让 𝐸 成为 3D-QALAS TR 中 𝐴 采集之一中的回声数量(通常 𝐴= 5,𝐸= 120 →𝑇= 120 × 5 = 600 𝑒𝑐ℎ𝑜𝑒𝑠/𝑇𝑅 ),其中 𝑇 是回声总数。我们生成一个信号演化字典,用 SVD 计算低维线性基 Φ,从而产生一个易于处理的重建问题 𝑎𝑟𝑔𝑚𝑖𝑛 𝛼 ‖𝑦−𝐴Φ𝛼‖ + 𝑅(𝛼) ,其中 𝐴 表示傅里叶、线圈和采样算子以及 𝑅 正则化。通过使用 𝑥= Φ𝛼 解析时空体积,我们旨在利用与 𝑇 回声 2 的字典匹配来估计更清晰的定量图。图 2 (A) 中的体内实验表明,使用子空间可以减少估计的 T 2 图中的模糊。 CRB 翻转角优化:我们通过最小化两种方式的 CRB 来优化 3D-QALAS 中的翻转角:(1) 优化每个回波序列的一个翻转角 (2) 优化每个回波序列中的所有翻转角。我们使用传统的 4 度翻转角初始化了这两种优化,利用了代表性组织参数 [T 2 =70ms、T 1 =700ms、M0=1] 和 [T 2 =80ms、T 1 =1300ms、M0=1],并最小化了基于 CRB 的成本函数。我们为 3D-QALAS 实现了自动微分兼容信号模拟 3,从而能够计算基于 CRB 的优化的梯度。减少采集:我们通过从 TR 末尾移除采集,设计了具有 A ={5,4,3} 采集的优化序列,从而加快了扫描速度。实验:我们在扫描仪上实施了针对每个回波序列进行优化的 3D-QALAS 序列,并使用 Mini System Phantom、型号 #136(CaliberMRI,美国科罗拉多州博尔德)和人类受试者(经 IRB 批准)上的常规和优化序列采集数据,进行了 3 次和 5 次采集(1x1x1mm3 分辨率,R=2)。我们比较了使用子空间重建(秩 = 3)和字典匹配估计的定量图。结果:优化序列:图 2(B)绘制了优化的翻转角和(C)与应用子空间重建进行定量估计时的传统序列相比的所得 CRB。优化可以减少 CRB 或者以更少的采集次数匹配传统的 5 次采集 CRB,从而有可能缩短扫描时间。模型和体内:图 3(A)和(B)显示了从模型和体内数据估计的图,其中每个 ETL 翻转角优化的序列(A=3,5 次采集)与恒定翻转角匹配。讨论和结论:未来的工作将实施全翻转角优化序列来解决未来实验中的 T 1 偏差。将子空间重建与自动微分启用的翻转角优化相结合,可获得改进的 3D-QALAS 序列,并将扫描时间缩短 1.75 倍。参考文献:[1] Kvernby, S. et al. J. Cardiovasc. Magn. Reson. 16 , 102 (2014)。[2] Tamir, JI 等人 Magn. Reson. Med. 77 , 180–195 (2017)。[3] Lee, PK 等人 Magn. Reson. Med. 82 , 1438–1451 (2019)。致谢:NIH R01 EB032708、R01HD100009、R01 EB028797、U01 EB025162、P41 EB030006、U01 EB026996、R03EB031175、R01EB032378、5T32EB1680
使用时间改进的 3D-QALAS 中的 T1 和 T2 映射...
主要关键词