机构名称:
¥ 1.0
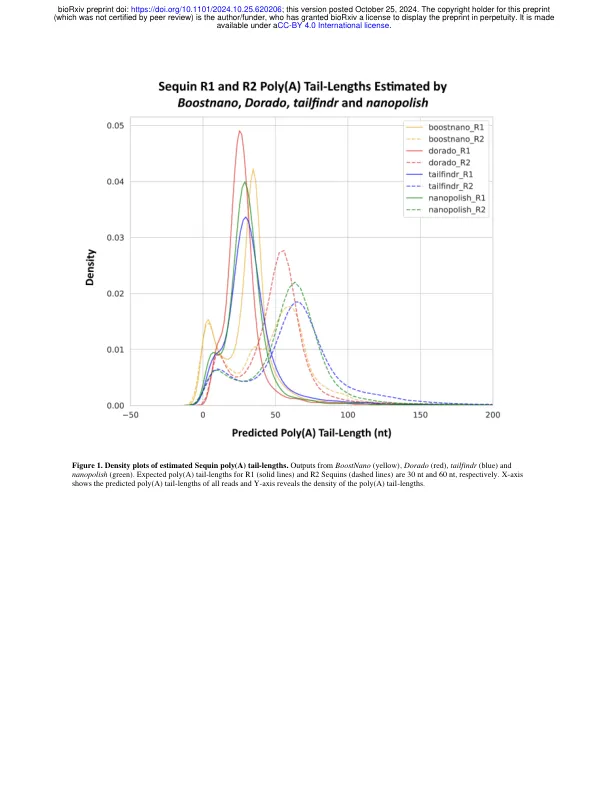
多聚腺苷酸化是一个动态过程,在细胞生理学中非常重要。Oxford Nanopore Technologies 的直接 RNA 测序提供了一种对全长 RNA 分子进行测序以及对转录组和表观转录组进行分析的策略。目前有几种可用于 poly(A) 尾长估计的工具,包括 tailfindr 和 nanopolish 等成熟工具,以及两种较新的深度学习模型:Dorado 和 BoostNano。然而,这些工具的准确性与金标准数据集的基准测试有限。在本文中,我们使用合成 RNA 标准(Sequins)评估了四种 poly(A) 估计工具,这些标准具有已知的 poly(A) 尾长,并提供了一种衡量 poly(A) 尾长估计准确性的有效方法。所有四种工具生成的平均尾长估计值都在正确值的 12% 以内。总体而言,由于 Dorado 运行时间相对较快、变异系数低并且易于使用且可与碱基调用集成,因此被推荐作为首选方法。
使用合成 RNA 作为 RNA 直接测序中 poly(A) 长度推断的基准
主要关键词