机构名称:
¥ 2.0
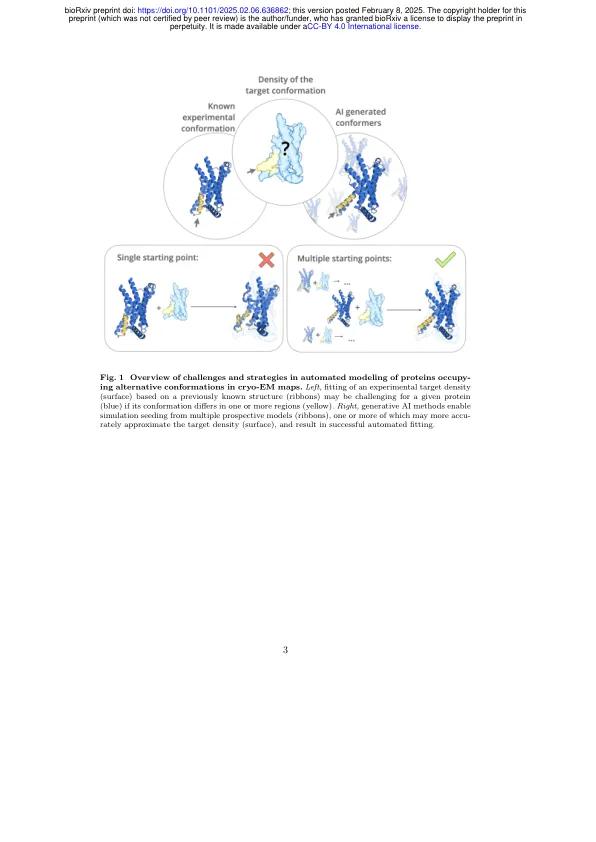
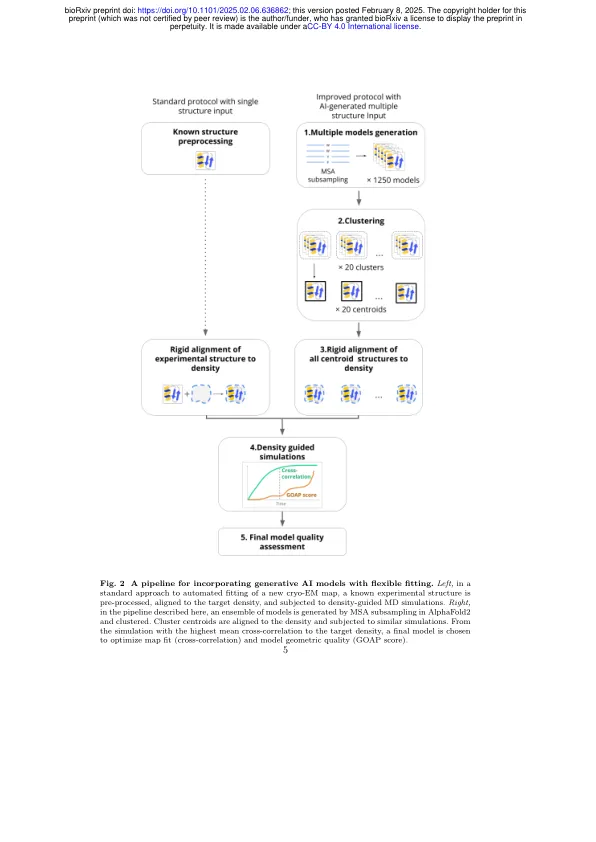
建模原子坐标为目标冷冻电子显微镜图是结构确定的关键步骤。尽管最近进步,但具有多个功能状态的蛋白质仍然是一个挑战 - 尤其是当某些状态无法使用合适的分子模板时,地图分辨率不足以构建从头模型。这是一种常见的情况,例如,在药理学相关的膜结合受体和转运蛋白中。在这里,我们介绍了一种改进方法,其中i)几个初始模型是通过Alphafold2中多个序列比对(MSA)空间的随机次采样生成的,ii)将对基于结构的聚类进行基于结构的群集,iii)密度引导的分子动力学模拟从中心结构和IV中进行了模型,并在模型中进行了模型。与三种膜蛋白(降钙素受体样受体,L型氨基酸转运蛋白和丙氨酸 - 二孢菌碱转运蛋白)相比,这种方法提高了拟合精度。我们的结果表明,使用生成AI和基于模拟的精炼结合使用的集合结构有助于在几种膜蛋白家族中建立替代状态。
对具有生成AI和密度引导的替代状态中的冷冻EM结构进行建模