机构名称:
¥ 2.0
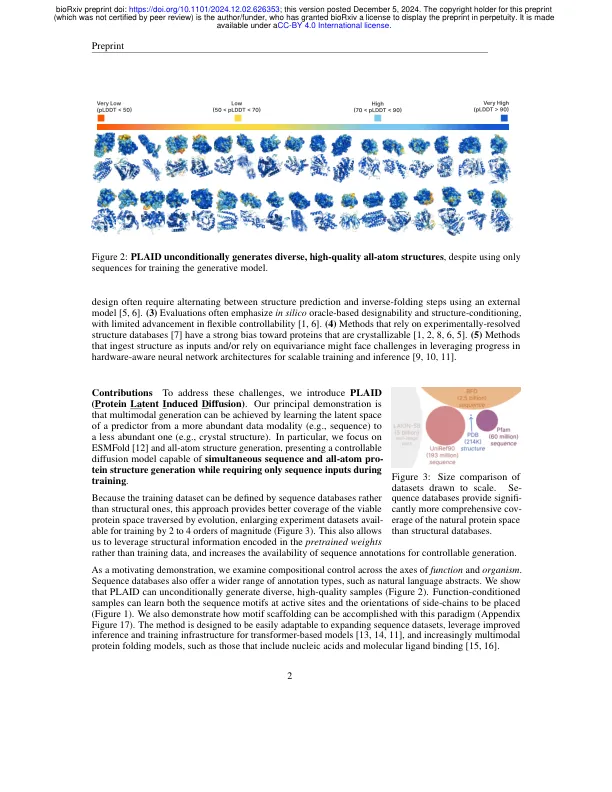
蛋白质设计的生成模型对其潜在的科学影响引起了人们的兴趣。但是,蛋白质功能是由许多模态介导的,同时产生多种方式仍然是一个挑战。我们提出了格子(p Rotein la tent i doffusion),这是一种多模式蛋白产生的方法,它从预测变量的潜在空间中学习和样品,从更丰富的数据模式(例如序列)映射到较少丰富的一种(例如,晶体结构)。具体来说,我们解决了全原子结构的生成设置,该设置需要产生3D结构和1D序列以定义侧链原子的位置。重要的是,格子只需要序列输入才能在训练过程中获得潜在表示,从而使序列数据库用于生成模型训练,并且与实验结构数据库相比,将数据分布增加了2至4个数量级。仅序列训练还允许访问更多的注释以进行调节。作为示范,我们对基因本体论的2,219个功能和生命之树的3,617种生物使用组成条件。尽管在训练过程中不使用结构输入,但生成的样品表现出强大的结构质量和一致性。功能条件的世代学习活跃位点的侧链残基身份和原子位置,以及跨膜蛋白的疏水模式,同时保持整体序列多样性。型号的权重和代码可在github.com/amyxlu/plaid上公开获得。
从仅序列训练数据中生成全原子蛋白结构
主要关键词