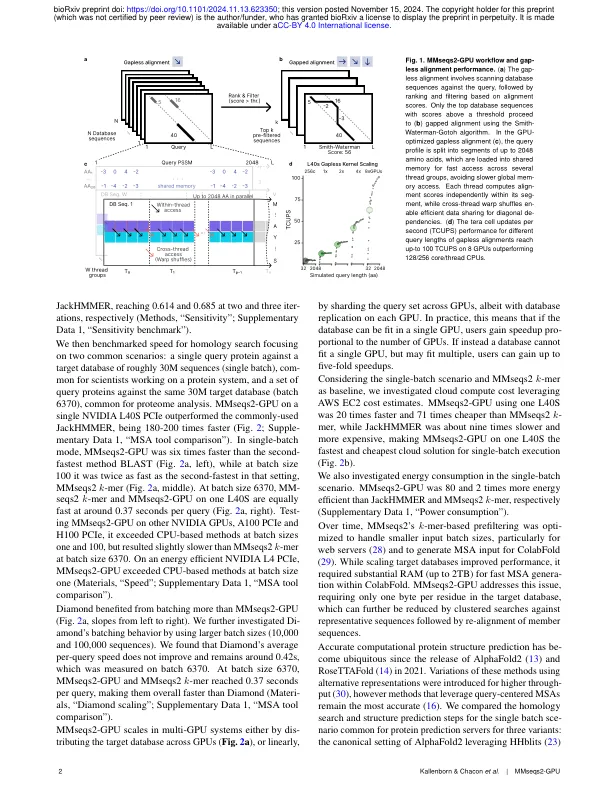
通过计算工具从参考数据库中检索进化相关的序列(HO-MOLOGS)已经实现了许多生物学的进步(1-4)。在基于序列的蛋白质同源性范式上构建这些工具(5,6),通过搜索类似的氨基酸性序列来检测数百万到数十亿参考条目中输入查询的同源物。在数十年中,同源性搜索对于推断蛋白质特性至关重要(7-9),例如二级结构预测(10),检测蛋白质残基对之间的直接耦合(11)和第三纪结构预测,长期以来对生物学的巨大挑战(12)。特定的远程同源物已被证明是对当代深度学习方法(如Alphafold2等)(13 - 15)(13-15)的输入,以预测准确的结构(16-18)。要检索远程同源物,需要在数据库中查询和参考序列之间检测对成对的相似性的敏感工具。从理论上讲,可以通过应用基于动态编程的,间隙的史密斯 - 水手-GotoH算法(19,20)来找到高灵敏度,以在每个查询参考对准时找到最佳路径(对准)(21)。但是,参考序列数据库的不断增长的大小(17)使这种详尽的方法不切实际。结果,基于启发式的方法,例如BLAST(1),PSI-BLAST(22),MMSEQS2(4)和DIAMOND(3),在执行计算价格昂贵的间隙计算之前,融合了预滤波技术,以修剪大多数不同的序列。这通常是通过采用种子和扩展策略来完成的,其中简短的k-mer单词(“种子”)被索引和匹配,然后将其扩展到间隙比对。敏感的对准器(2)和hhblits(23)都采用了简化的动态编程方法,该方法在序列对之间的对齐矩阵的所有无间隙路径(严格的对角线)中得分,以找到最高得分的未射程匹配。与基于k的方法不同,是较低的比对的较低结合的近似值,无间隙对准会导致所有对以计算效率为代价的分数。探索了几种方法以达到更高的执行速度,无论启发式如何,例如中央处理单元
使用MMSEQS2
主要关键词