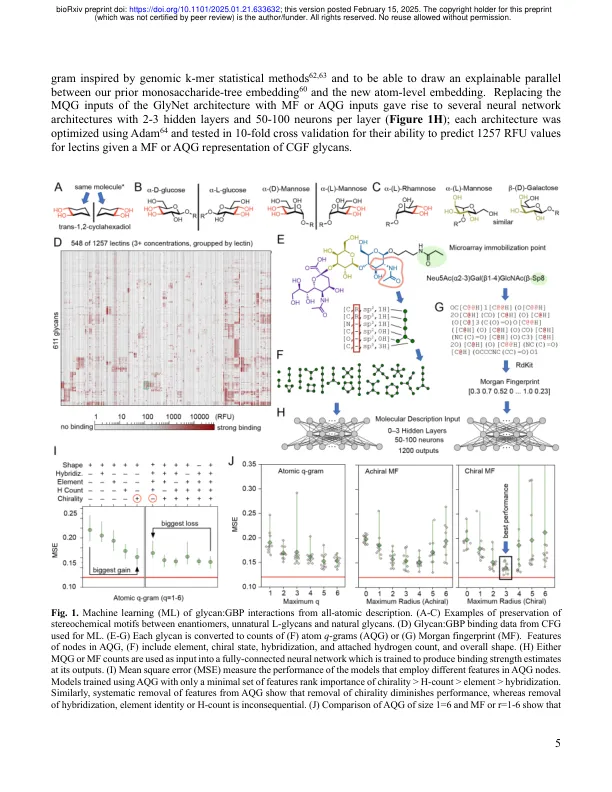
糖生物学中的跨性识别是生物学上常规蛋白质与生物聚糖的对映异构体之间的相互作用(例如,L蛋白与L-己糖结合的L蛋白质与L-Hexoses结合)与生命王国的生物体中的相互作用。通过对称性,它还描述了手性镜面蛋白与正常D-聚糖的相互作用。跨性识别的知识对于理解现有生命形式与人造镜像形式的潜在相互作用至关重要,但是目前已知的蛋白质 - 聚糖相互作用规则不足。为了构建一种学习这种相互作用的方法,我们构建了机器学习模型,以预测代表原子图的蛋白质和聚糖之间的结合强度,而不是单糖。基于聚糖的基于原子Q -gram和Morgan指纹(MF)表示,可以训练ML模型,以预测所有天然聚糖的聚糖,糖化化合物和对映异构体的凝集素结合特性。对此训练的关键是将不同的数据合并 - 某些数据与来自Glycan微阵列的相对荧光单元(RFU),而来自ITC的K d值的其他数据则是在特定的凝集素浓度下使用通用的“分数结合”参数F。MCNET是一个完全连接的神经网络体系结构,将MF和浓度(C)作为输入,并返回147个凝集素的F。MCNET的性能与Glynet模型相媲美,并且通过代理与其他最新的最先进的模型来预测蛋白质 - 聚糖相互作用的强度。MCNET有效预测了糖化化合物与甘叶蛋白1、3和7的结合。糖化化合物)。从基于单糖的描述中脱离,使MCNET可以预测跨性识别。我们使用液态聚糖阵列来验证一些预测,例如L-甘露糖与D-Mannose结合凝集素,纯化的CONA和DC-SIGN显示在细胞上的DC-SIGN以及L-MAN与半乳糖糖结合的凝集素的弱结合。MCNET的原子级输入使得从生活和非聚糖结构的所有王国中的各种聚糖中结合蛋白质 - 聚糖数据是可能的(例如,通用分数结合参数使得可以统一不同的定量观测值(K D / IC 50,RFU,色谱保留时间等)。我们认为,这种方法将有助于从不同的糖生物学数据集中合并知识,并预测与当前ML模型无法获得的不常见/不自然的聚糖的蛋白质相互作用。
蛋白质 - 聚糖相互作用的原子级机器学习糖生物学中的蛋白质 - 聚糖相互作用和跨性识别
主要关键词