机构名称:
¥ 2.0
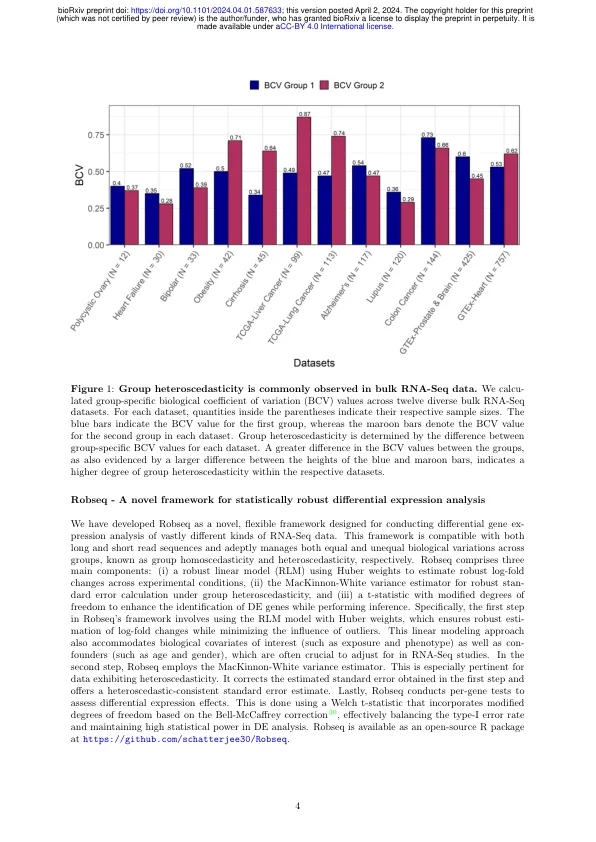
尽管有几种备受瞩目的最先进的方法可用,但分析批量RNA-Seq数据仍在面临重大挑战。最近的研究的证据表明,流行的差异表达(DE)工具(例如EDGER和DESEQ2)容易受到惊人的错误发现率(FDR)的影响。这些研究表明,在这些模型中观察到的FDR通货膨胀可能归因于诸如违反参数假设的问题或无法有效处理数据中的异常值。在这里,我们认为群体异质性也可以促进这一提升的FDR,这一现象在很大程度上被研究界忽略了。我们介绍了一种新型的统计模型Robseq,该模型旨在在差异分析中有效的每种功能建模,当时是当群均均一的假设未得到满足时。Robseq利用了稳健的统计文献中建立的统计机制,包括M估计量来稳健地估计基因表达水平变化和Huber-Cameron方差估计器来计算异性设置中的鲁棒标准误差。此外,出于推理目的,它还结合了Welch T统计量的自由度调整,有效地解决了RNA-Seq差异表达中FDR通胀的问题。通过详细的模拟和全面的基准测试,我们表明Robseq成功地将错误的发现和I型错误率保持在名义级别,同时与众所周知的DE方法相比保留了高统计能力。对种群级RNA-seq数据的分析进一步表明,Robseq能够鉴定出与复杂人类疾病有关的具有生物学上重要的信号和途径,这些信号和途径涉及复杂的人类疾病,否则这些信号和途径否则无法通过已发表的方法揭示。Robseq的实现可在https://github.com/schatterjee30/robseq上公开提供。
组异质性
主要关键词