机构名称:
¥ 2.0
为了表征基因组编辑产品的脱靶效应,不仅需要通过计算机分析预测与目标基因序列相似序列的存在,还需要使用实验方法来分析整个人类基因组中潜在的脱靶位点[32-35]。寻找候选脱靶位点的实验方法包括在基因组编辑过程中在切割位点引入合成 DNA 标签,并在整个基因组中分析该标签的掺入情况的方法(GUIDE-seq)[36],以及 DIGENOME-seq [37, 38]、CIRCLE-seq [39] 和 SITE-seq [40] 等方法,它们使用从细胞中提取的基因组来寻找基因组编辑酶可能的切割位点。这些分析可能包括识别癌症相关基因中的单核苷酸变异 (SNV)/插入和缺失 (Indel) 和拷贝数变异 (CNV) 等突变[41]。通过计算机分析和实验方法检测到的脱靶候选位点是否确实发生了切割或缺失的潜在方法包括对经过基因组编辑的细胞进行全基因组测序(WGS)[33, 35] 和扩增子测序,通过 PCR 扩增候选位点并进行深度测序 [42]。在这些分析中,检测灵敏度取决于碱基序列的读取深度。但是,由于新一代测序(NGS)的错误频率,检测脱靶效应非常困难,其发生频率低于0.1%(表1)。
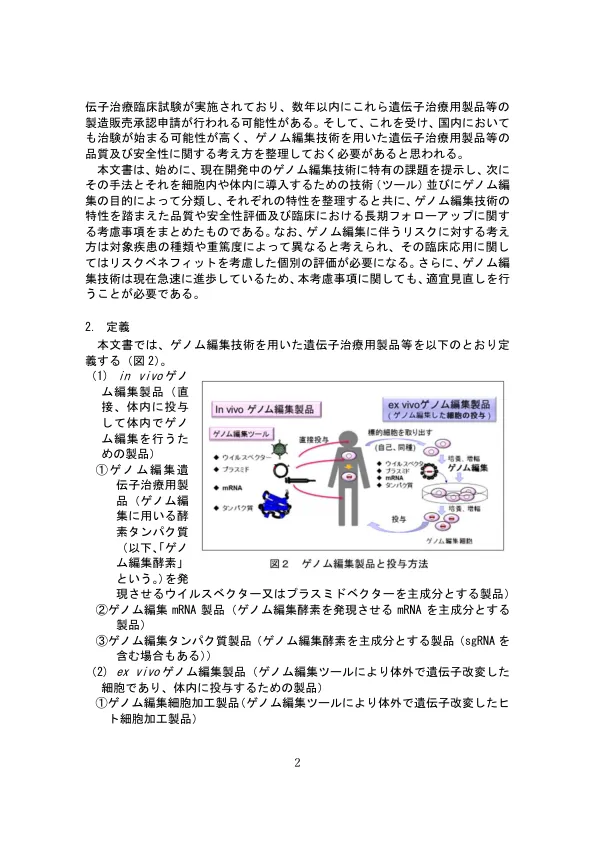
利用基因组编辑技术的基因治疗产品的质量及质量保证……